生物信息法推算青光眼小鼠視乳頭及視網膜功能變化
劉靜坤,王霖邦,王 兵,盛亞玲,賀 靜,夢粉鴿
?
·實驗研究·
生物信息法推算青光眼小鼠視乳頭及視網膜功能變化
劉靜坤1,王霖邦2,王兵3,盛亞玲1,賀靜1,夢粉鴿1
1Department of Ophthalmology, Xi’an Honghui Hospital, Xi’an 710054, Shaanxi Province, China;2Chongqing Medical University, Chongqing 400016, China;3Department of the Laboratory, Shaanxi Province Health Inspection Institution, Xi’an 710077, Shaanxi Province, China
?METHODS: The data in this study is from Gene Expression Omnibus(GEO) which belong to Nation Center for Biotechnology Information (NCBI), the quality of the raw data CEL files was processed and analyzed by the Expression software which belong to Affymetrix Inc., Santa Clare, CA, USA. Significant analysis method (SAM) which base on the T test was used to identified the significant genes. Based on GRNInfer and Gvedit soft we set up gene networks of optic and retina of mice and further more enriched analysis which based on DAVID and MAS3.0 online software were processed.
?RESULTS: The analysis between the group of the optic nerve heads and retinas in different stage of glaucoma showed that the amount of significant different expressed genes in the optic never head group increased significantly comparing with the group of retina in the early stage of glaucoma, the analysis of the genes network construction show that: the node genes of optic nerve heads included Unc13c、Kif5a、TRPM1、PANX; and the node genes of retina include POU4F1, NEFL, BC03870, CALB2. Metabolic pathways enrichment analysis which based on MAS3.0 online platform show that there was mainly the amyotrophic lateral sclerosis, tyrosine metabolism, melanogenesis, Nitrogen metabolism, Gap junction, Leukocyte transendothelial migration metabolism pathway enriched out in optic nerve head; and there was mainly amyotrophic lateral sclerosis, neurodegenerative disorders, prostate cancer, leukocyte transendothelial migration metabolism pathway enriched out in retina.
?CONCLUSION: By understanding bioinformatics result, it seems optic were more sensitive than the retina to high intraocular pressure, and weather high expression of TYrp1 gene can be as a sensitive diagnostic item require more evidence back up. Functional enrich analysis of node gene showed that cytoskeleton reconstructed,molecular motor and nutrients transport function improve in optic; and in retina, the most prominent finding in retina was enrichment function modules were focus on regeneration, repairing and differentiation of cells, which remind that we should reinforce research on reparation of retina of primary glaucoma. Metabolic pathways enrichment analysis show that inflammatory response plays prominent place in optic and retina of primary glaucoma, because of the optic narrow and crowed anatomic shape, nutrient metabolism and substances transfer enrichment modules play an important role in optics of primary glaucoma.
目的:本研究運用生物信息學軟件,利用數據庫資料,推測青光眼早期小鼠視乳頭及視網膜可能的信號路徑及基因生物功能模塊,為研究青光眼發病機制提供新的途徑。
方法:本研究的數據是從美國生物技術信息中心GEO基因表達數據庫獲得。利用美國昂飛公司Expression Console軟件對原始的CEL數據進行標準化及對數化轉換處理。利用以t檢驗為基礎的基因表達差異顯著性分析方法SAM對基因芯片數據進行顯著性差異分析,分析后篩選顯著性差異表達基因,采用GNRInfer軟件構建了小鼠視乳頭及視網膜前50個有顯著差異表達基因的調控網絡,同時我們利用MAS3.0分子注釋系統軟件及DAVID軟件這兩種在線分析平臺中進一步富集基因信號通路。
結果:青光眼各組視乳頭和視網膜及其相對應組的顯著性差異基因分析表明,在青光眼早期視乳頭組及視網膜組較之正常組相比視乳頭組顯著性差異基因數量明顯增多,青光眼視乳頭及視網膜網絡構建顯示,視乳頭基因網絡中主要調控節點基因包括Unc13c、Kif5a、TRPM1、PANX;視網膜基因網絡中主要調控節點基因包括POU4F1、NEFL、BC03870、CALB2。MAS在線信號通路分析顯示,視乳頭組織中主要的信號代謝通路包括肌萎縮側索硬化代謝通路、神經退行性紊亂、白細胞穿內皮性遷移及前列腺癌信號通路。視網膜組織主要代謝通路包括肌萎縮側索硬化代謝、酪氨酸代謝、黑色素生成、氮代謝、縫隙連接、白細胞穿內皮遷移。
結論:早期青光眼階段視乳頭較視網膜對眼壓更為敏感,特別是Tyrp1基因在早期高眼壓的表達能否作為青光眼早期生物學指標有待進一步探討。在青光眼高眼壓壓力下,節點分子生物學功能顯示在視乳頭組織中,細胞骨架的重排、生物驅動馬達動力、物質代謝及運輸力均為增強;而在視網膜組織中,最突出的表現在細胞的再生、分化及修復作用,此結果提示我們在青光眼的研究中應重視哺乳動物視網膜損傷后自身修復的研究。代謝通路富集分析顯示,炎性反應在視乳頭及視網膜的病理反應中均起到非常重要的作用,而在視乳頭中由于其狹窄而擁擠的解剖結構在青光眼發病中存在營養代謝及物質轉運障礙。
青光眼;生物計算;信號通路;生物標記
引用:劉靜坤,王霖邦,王兵,等.生物信息法推算青光眼小鼠視乳頭及視網膜功能變化.國際眼科雜志2016;16(11):2014-2018
0 引言
青光眼可引起眼部多部位損傷,是眼科常見的退化性病變,全世界約有7億患病人群,高眼壓是引起青光眼患者眼部損傷的重要因素,但目前降低眼壓治療并不是總是有效,且有很多毒副作用,因此為改善治療效果,研究青光眼病理生理學分子生物學機制有重要意義。青光眼屬于多基因致病及環境因素致病性疾病,在高眼壓作用下視網膜組織與對照組織基因芯片顯著性差異表達基因數量較多,如何確定重要的節點基因還有很多新的思路及方法有待探索。目前對青光眼早期致病性基因研究相對較少。為進一步探究早期青光眼發病機制,我們挖掘NCBI中GEO數據庫數據,運用生物信息學方法構建并推算早期青光眼視網膜及視乳頭可能發生信號通路及代謝的改變,并進一步運用線性代數法構建視網膜及視乳頭基因調控網絡,推算基因節點基因在相應組織的功能。
1 材料和方法
1.1材料本研究數據來源于美國生物技術信息中心(Nation Center for Biotechnology Information,NCBI)的GEO(Gene Expression Omnibus)數據庫,數據樣本號為GSE26299(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE26299)。我們選取了樣本中的100個基因芯片數據。樣本分組定義:對照組;青光眼1組:無青光眼癥狀第一階段(NO1);青光眼2組,無青光眼癥狀第二階段(NO2);青光眼3組,出現中度青光眼癥狀(MOD);青光眼4組,出現重度青光眼癥狀(SEV)。樣本分組:對照組中視乳頭組及視網膜組各10眼;青光眼1組,視乳頭組及視網膜組各10眼;青光眼2組,視乳頭組及視網膜組各10眼;青光眼3組,視乳頭組及視網膜組各10眼;青光眼4組,視乳頭組及視網膜組各10眼。
表1青光眼各組視網膜與對照組顯著高表達基因數目及功能富集模塊數目
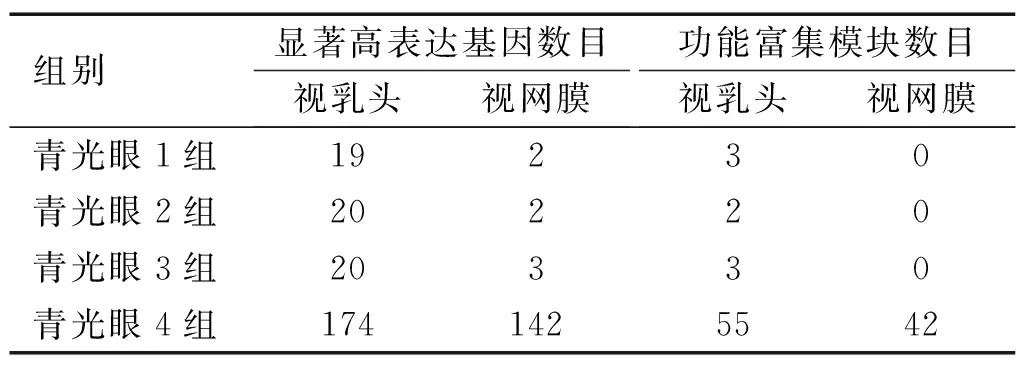
組別顯著高表達基因數目視乳頭視網膜功能富集模塊數目視乳頭視網膜青光眼1組19230青光眼2組20220青光眼3組20330青光眼4組1741425542
1.2方法
1.2.1原始數據處理原始數據均為CEL格式,原始數據的處理采用美國昂飛公司Expression Console軟件對原始數據進行標準化及對數化轉換處理。
1.2.2基因芯片顯著性差異表達分析基因表達差異顯著性分析方法(significance analysis of microarray,SAM)是一種對基因芯片數據進行顯著性差異分析的方法,該方法是一種以t檢驗為基礎的統計學分析,本研究中SAM分析是采用TM4軟件的子軟件MuliExperiment(MeV)的SAM分析功能。本研究中,分別對比視乳頭及視網膜組中D2-Glaucoma與相應的NOE1組、NOE2組、MOD組及SEV組。
1.2.3基因調控網絡構建基因調控網絡屬于系統生物學的研究范疇,使用GNRInfer軟件是在線性代數基礎上推算基因間相互關系的工具軟件,結合GVedit(http://www.graphviz.org)畫圖軟件可將以基因表達數值矩陣轉化為基因間相互作用的可視化圖像。GNRInfer[1]中等式(1)代表一組數據所有的可能網絡。
J=(A'-A)U^1VT+YV=J+YVT(1)
1.2.4 DAVID聚類分析DAVID(Database for Annotation,Visualization,and Integrated Discovery)(http://www.david.niaid.nih.gov)由數據分析軟件及生物學數據庫構成,用數學算法從大量生物信息中挖掘有用的生物信息,并進一步對基因的功能進行注釋、富集及功能聚類[2-3]。
1.2.5分子注釋系統3.0(MAS 3.0)分子注釋系統MAS3.0(Molecule Annotation System 3.0)是對高通道基因芯片提供基因功能注釋及富集分析的免費在線分析平臺。MAS3.0 在線分析平臺將Genebank等生物數據庫信息通過富集分析提供給研究者基因本體注釋、信號通路等知識。本研究應用MAS3.0系統分別富集視乳頭及視網膜顯著差異高表達基因信號通路富集圖。
2 結果
2.1青光眼各組視乳頭及視網膜與其相應對照組的SAM分析分析表明,在青光眼視乳頭組4組高表達基因分別有19、20、20和174個,而視網膜組高表達基因數分別為2、2、3和142個(表1)。其中視乳頭組青光眼1組中DAVID富集功能模塊包括:膜結合、黑色素代謝、信號傳遞、轉錄調節;2組中DAVID富集功能模塊包括膜結合、黑色素代謝模塊;3組中DAVID富集功能模塊包括膜結合、信號傳遞及分泌、轉錄調節;4組中DAVID富集功能模塊則富集出55個功能模塊。視網膜組青光眼1、2、3組中均未富集出DAVID功能模塊;4組中則富集出42個功能模塊。
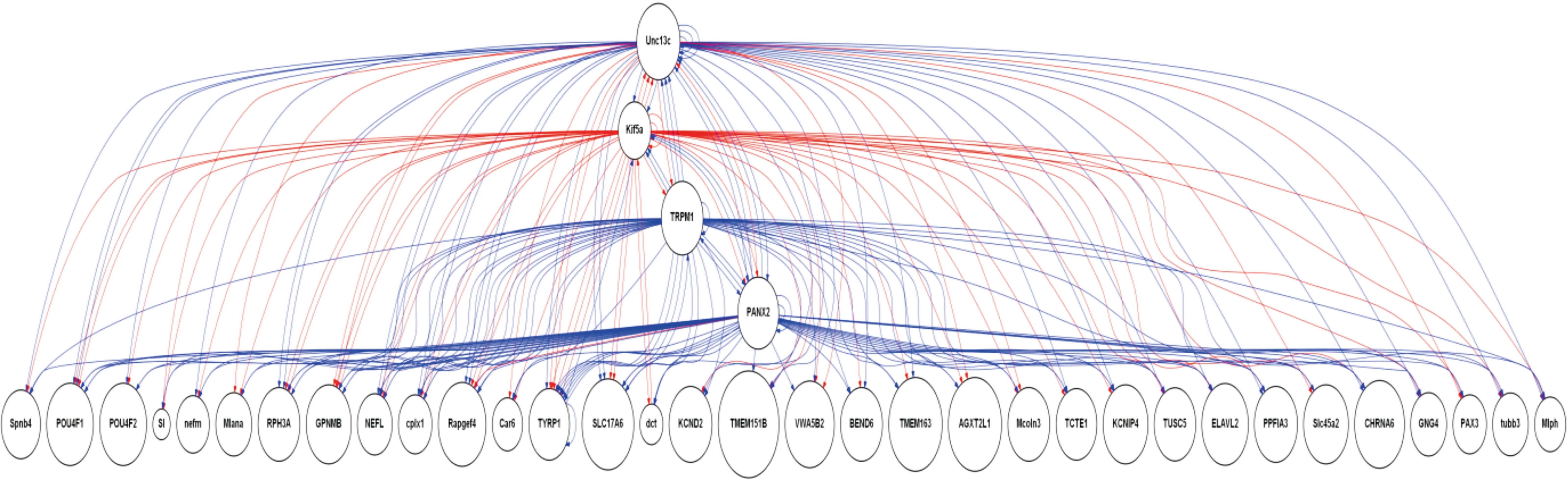
圖1視乳頭顯著高表達基因網絡(圖中圓圈的內容均為基因的名稱,藍色線條及箭頭均表示上游基因對下游基因的減弱作用,紅色線條及箭頭均表示上游基因對下游基因的加強作用)。
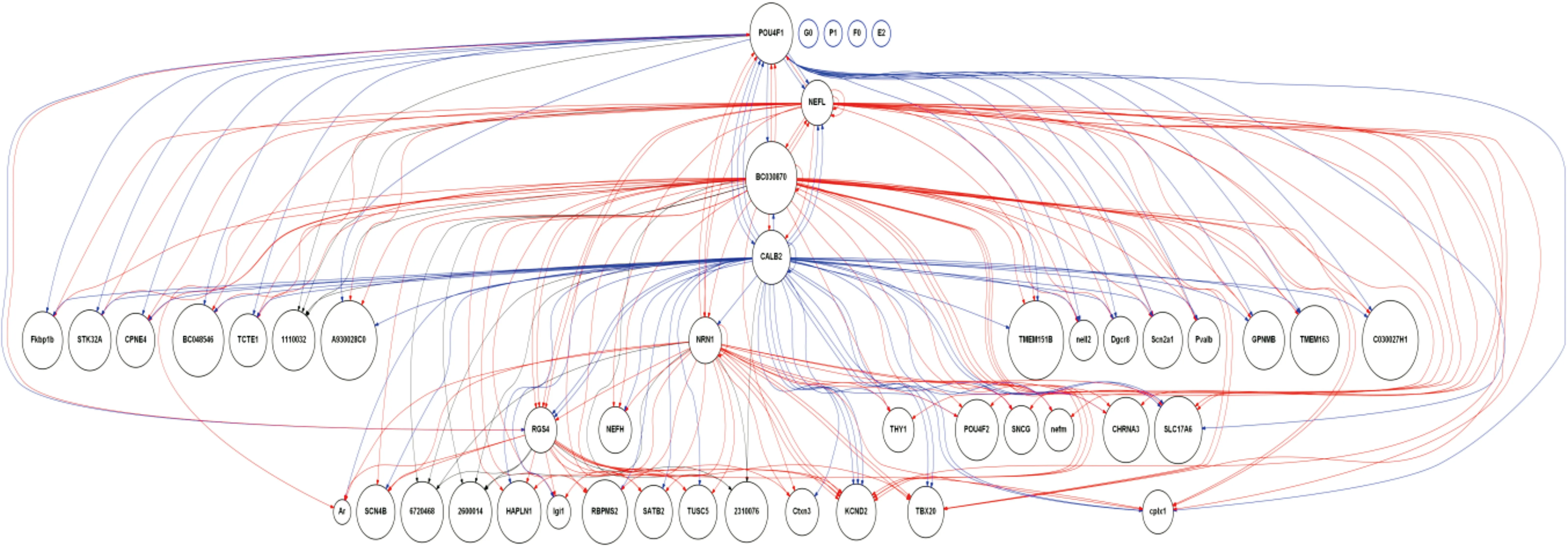
圖2視網膜顯著高表達基因網絡(圖中圓圈的內容均為基因的名稱,藍色線條及箭頭均表示上游基因對下游基因的減弱作用,紅色線條及箭頭均表示上游基因對下游基因的加強作用)。
2.2視乳頭和視網膜顯著高差異表達基因網絡構建本研究分別應用線性代數方法將縱軸為基因序列號、橫軸為時間軸顯著高表達基因矩陣轉化為基因相互作用網絡,網絡中每個基因均有其上游基因及下游基因,基因間相互關系分為加強或減弱,其中紅線及箭頭表示加強作用,藍線及箭頭表示減弱作用。圖1為視乳頭顯著高表達基因網絡,圖2為視網膜顯著高表達基因網絡。視乳頭基因網絡中主要調控節點基因包括Unc13c、Kif5a、TRPM1、PANX2,視網膜基因網絡中主要調控節點基因包括POU4F1、NEFL、BC03870、CALB2。
2.3 MAS3.0富集結果結果顯示,視乳頭組織中主要的信號代謝通路網絡如圖3,主要代謝通路包括肌萎縮側索硬化代謝、酪氨酸代謝、黑色素生成、氮代謝、縫隙連接、白細胞穿內皮遷移;視網膜組織中主要代謝通路網絡如圖4,主要的信號代謝通路包括肌萎縮側索硬化代謝通路、神經退行性紊亂、白細胞穿內皮性遷移及前列腺癌信號通路。
3 討論
既往研究顯示,病理性高眼壓小鼠模型可發生視網膜神經節細胞的凋亡、視乳頭杯盤比的增大及視神經纖維束腫脹斷裂[4]等病理性病變,但早期青光眼視網膜及視乳頭病理生理學發病機制及其基因網絡構建的研究較為缺乏,為探索其發病機制,我們挖掘了GEO數據庫并利用線性代數軟件進一步推算其數據中所蘊含的生物學信息。

圖3視乳頭顯著高表達基因網絡信號通路網絡圖(圖中矩形內容為所富集的信號通路,紅色的深度代表富集值的強度,顏色越深,富集值越高,黃色橢圓內容代表基因名稱;Amyotrophic lateral sclerosis:ALS,肌萎縮側索硬化信號通路;Tyrosine metabolism:酪氨酸代謝信號通路;Melanogenesis:黑色素代謝信號通路;Nitrogen metabolism:氮代謝信號通路;Gap junction:縫隙連接信號通路;Leukocyte transendothelial migration:白細胞游走遷移信號通路)。
早期青光眼視網膜及視乳頭顯著差異基因分析表明,視乳頭組在青光眼早期及中期即青光眼1組、2組及3組顯著增高基因數目較視網膜明顯增多,富集青光眼1組中19個基因我們發現了3個基因功能模塊,主要包括細胞膜結合功能、色素代謝模塊、轉錄功能,以上結果表明在早期青光眼階段視乳頭較視網膜對眼壓更為敏感。從解剖學原因分析,視乳頭的組織或細胞及視網膜動、靜脈受到壓力后可以緩沖的外界環境較視網膜所處的環境相比比較受限,視乳頭不僅受到機械壓力,同時高眼壓對動靜脈的壓力可造成視乳頭組織及細胞的缺血缺氧等反應,這些原因均可對早期青光眼小鼠視乳頭基因的表達產生影響。
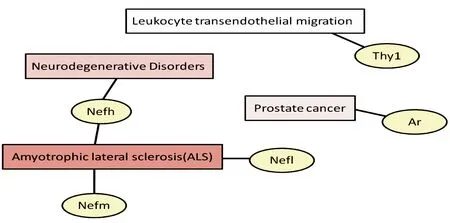
圖4視乳頭顯著高表達基因網絡信號通路網絡圖(圖中矩形內容為所富集的信號通路,紅色的深度代表富集值的強度,顏色越深,富集值越高,黃色橢圓內容代表基因名稱;Amyotrophic lateral sclerosis:ALS,肌萎縮側索硬化信號通路;Neurodegenerative Disorders:神經退行性信號通路;Prostate cancer:前列腺癌信號通路;Leukocyte transendothelial migration:白細胞游走遷移信號通路)。
青光眼其本質是一種神經退行性疾病,病理性眼壓升高被認為是青光眼疾病發生的重要危險因素。視乳頭組及視網膜組相應組別中高表達差異基因的富集結果顯示,在青光眼早期,這些基因對壓力較為敏感,對抵御損傷較為重要。既往研究顯示,青光眼組織的損傷可導致一系列的信號傳導通路障礙,包括線粒體功能異常、蛋白水解級聯反應、內質網壓力及氧化應激等,但我們的研究卻推測視乳頭在青光眼早期高表達基因主要加強了基因轉錄、黑色素代謝、信號傳遞、膜結合信號傳遞功能,這些功能常出現在急性的各種炎癥、機械等壓力下[5]。黑色素代謝模塊中的酪氨酸酶相關蛋白1(Tyrp1)控制黑色素生物合成鏈的遠端反應,在早期高眼壓視乳頭中Tyrp1基因的高表達能否作為青光眼損傷的早期敏感性指標之一有待于進一步研究。
構建基因網絡并推算基因間的相互作用方式是近年來系統生物學的研究熱點之一。各種生物分子及其相互作用可被簡化為一個基因網絡。通過基因芯片的矩陣數據,應用線性代數計算出網絡拓撲結構圖,從而揭示基因間復雜的作用機制,并推測相關基因功能。隨著高通量基因檢測技術發展,網絡構建成為從生物系統學層次上研究基因的功能提供有效的手段,并依次可識別節點“hub”基因。同時可以通過“連坐法”(guilt-by-association,GBA)來發現新的基因功能[6],另外越來越多的證據表明表型是由基因社團(community)來決定,社團間基因相互作用可提供更多的生物表型方式[7],因此基因的網絡構建為從系統層次揭示生命本質提供新的機遇。
本研究所構建青光眼視乳頭基因網絡中的節點基因包括Unc13c、Kif5a、TRPM1、PANX2。研究認為Munc13對神經元發育來說甚至必不可少[8],并參與了神經的可塑性過程,與細胞骨架的重排有關[9]。能高效地將三磷酸腺苷(adenosine triphosphate,ATP)結合以及水解產生的化學能轉化為機械能[10],同時攜帶著“貨物”沿著微管定向移動[11-13]。TRPM1屬瞬時受體電位離子通道(transient receptor potential channels,TRP channels)。維持細胞內外環境的離子穩態等眾多生命活動[14-17]。PANX2屬于Pannexin蛋白,是縫隙連接通道的亞單位,可傳遞突融的傳遞[18-19]、ATP的釋放[20]、金屬離子的傳遞[21]和感覺信號的傳導[22]。從以上生物信息學結果可推測,視乳頭在青光眼的機械壓力下,篩板各層的的物理壓力可引起相應基因的表達增高,并進一步加強視乳頭組織中細胞骨架的重排、生物驅動馬達的動力的增強、ATP、離子通道等運輸的加強以及囊泡運輸的增強,這些分子標志為青光眼的診斷、治療及防治提供新思路。
而在視網膜中的節點基因中,Pou4F1屬于第四類POU結構域(POU4),它們對各種感覺神經系統的發育及神經元的分化方向選擇中起著重要作用[23-29]。研究發現屬于POU結構域轉錄因子家族成員Brn3在維持RGC亞型多樣性中所發揮著獨特作用[30]。神經絲輕鏈多肽(neurofilament light polypeptide,NEFL)在多種神經系統炎癥和非炎癥性神經系統疾病均有不同程度增高,對維持神經是細胞骨架的關鍵成分,在維持神經細胞形態及使有髓鞘的軸突再生方面有重要的作用[31-34]。鈣視網膜蛋白(Calretinin)又稱CALB2。當細胞受到外界刺激可使細胞質內Ca2+濃度增加時,CaBP即與Ca2+結合,隨即與相應靶蛋白結合發揮生物學效應鈣結合蛋白(CaBP),在細胞內激活酶或蛋白質,參與調節細胞的功能代謝,同時在細胞核內對細胞分化、發育、增殖、壞死和凋亡等功能也起重要作用[35-37]。Neuritin(Nrn1)基因是1996年Nedivi等通過大鼠光刺激誘導實驗,在視皮質中首次發現并報道的可塑性相關候選基因15(CPG15)。在神經系統受損后發揮重要的再生修復作用[38-39]。以往的研究顯示哺乳動物視網膜組織的再生能力非常有限,但我們的研究顯示視網膜受到高眼壓后,節點基因功能富集均與視網膜的再生有關,提示我們應重視哺乳動物視網膜修復功能的研究。
MAS3.0視乳頭及視網膜差異表達基因中富集網絡信號通路圖3和4發現均存在肌萎縮硬化代謝通路(ASL)的改變,目前ASL的發病主要以基因突變學說為主,ASL主要侵犯脊髓前角細胞、腦干運動神經元及錐體束,但視網膜屬感覺神經末梢,強烈的富集值進一步證明相同的基因群在不同的發病部位可引起不同的疾病,疾病致病原因可由基因突變引起,也可與基因數量表達調控有關。視乳頭及視網膜也共同富集出白細胞內皮遷移,說明炎癥性反應在青光眼病理生理發病進程中起到非常重要的作用。而視乳頭獨有縫隙連接通路、酪氨酸代謝、黑色素生成、氮代謝,說明視乳頭代謝特點上存在營養代謝及物質轉運困難的視乳頭狹窄而擁擠的解剖結構及高眼壓下組織水腫壓迫血管,更進一步加重局部的血供及營養物質的供應。
綜上所述,通過生物信息學推算,我們發現早期青光眼階段視乳頭較視網膜對眼壓更為敏感,特別是Tyrp1基因在早期高眼壓的表達能否作為青光眼早期生物學指標有待進一步探索。在青光眼高眼壓壓力下,節點分子生物學功能顯示,在視乳頭組織中,細胞骨架的重排、生物驅動馬達動力、物質代謝及運輸力均為增強;而在視網膜組織中,最突出的表現為細胞的再生、分化及修復作用,此結果提示我們在青光眼的研究中應重視哺乳動物視網膜損傷后自身修復的研究。在一定的病理條件下必定是一些特定的基因團而不是單個基因發生了調節(表達增高或表達減弱),利用生物信息學方法富集出這些基因團的功能模塊、代謝通路,可科學地、全面掌握病理條件下基因表達高低所調節的功能改變的方向,例如本研究發現在小鼠眼壓升高時節點基因功能模塊主要表現為視網膜的再生模塊,那么進一步對這些節點基因研究,就可對視網膜再生機制、視網膜保護機制、抗青光眼藥物及視網膜再生性研究提供基礎。代謝通路富集分析顯示炎性反應在視乳頭及視網膜的病理反應中均起到非常重要的作用,而在視乳頭中由于其狹窄而擁擠的解剖結構在青光眼發病中存在營養代謝及物質轉運障礙,這些結論與既往研究及視網膜及視乳頭的解剖結構的一致性證明,再次證明生物信息學研究的可行性。
1 Wang Y, Joshi T, Zhang XS,etal. Inferring gene regulatory networks from multiple microarray datasets.Bioinformatics2006;22(19):2413-2420
2 Dennis G Jr, Sherman BT, Hosack DA,etal. DAVID: Database for Annotation, Visualization, and Integrated Discovery.GenomeBiol2003;4(5):3
3 Huang DW, Sherman BT, Tan Q,etal. The DAVID Gene Functional Classification Tool: a novel biological module-centric algorithm to functionally analyze large gene lists.GenomeBiol2007;8(9):R183
4 Gross RL, Ji J, Chang P,etal. A mouse model of elevated intraocular pressure: retina and optic nerve findings.TransAmOphthalmolSoc2003;101:163-171
5 Liu J, Wang B, Wang W,etal. Computational networks of activating transcription factor 3 gene in Huh7 cell lines and hepatitis C virus-infected Huh7 cell lines.MolMedRep2015;12(1):1239-1246
6 Gillis J, Pavlidis P. "Guilt by association" is the exception rather than the rule in gene networks.PLoSComputBiol2012;8(3):e1002444
7 Lee I, Lehner B, Crombie C,etal. A single gene network accurately predicts phenotypic effects of gene perturbation in Caenorhabditis elegans.NatGenet2008;40(2):181-188
8 Ashery U, Varoqueaux F, Voets T,etal. Munc13-1 acts as a priming factor for large dense-core vesicles in bovine chromaffin cells.EMBOJ2000;19(14):3586-3596
9 Rosenmund C, Sigler A, Augustin I,etal. Differential control of vesicle priming and short-term plasticity by Munc13 isoforms.Neuron2002;33(3):411-424
10 Rice S, Lin AW, Safer D,etal. A structural change in the kinesin motor protein that drives motility.Nature1999;402(6763):778-784
11 Svoboda K, Schmidt CF, Schnapp BJ,etal. Direct observation of kinesin stepping by optical trapping interferometry.Nature1993;365(6448):721-727
12 Vale RD. The molecular motor toolbox for intracellular transport.Cell2003;112(4):467-480
13 Hirokawa N, Nitta R, Okada Y. The mechanisms of kinesin motor motility: lessons from the monomeric motor KIF1A.NatRevMolCellBiol2009;10(12):877-884
14 Clapham DE. TRP channels as cellular sensors.Nature2003;426(6966):517-524
15 Huang CL. The transient receptor potential superfamily of ion channels.JAmSocNephrol2004;15(7):1690-1699
16 Clapham DE, Julius D, Montell C,etal. International Union of Pharmacology. XLIX. Nomenclature and structure-function relationships of transient receptor potential channels.PharmacolRev2005;57(4):427-450
17 Ramsey IS, Delling M, Clapham DE. An introduction to TRP channels.AnnuRevPhysiol2006;68:619-647
18 Zoidl G, Kremer M, Zoidl C,etal. Molecular diversity of connexin and pannexin genes in the retina of the zebrafish Danio rerio.CellCommunAdhes2008;15(1):169-183
19 Thompson RJ, Jackson MF, Olah ME,etal. Activation of pannexin-1 hemichannels augments aberrant bursting in the hippocampus.Science2008;322(5907):1555-1559
20 Ellsworth ML, Forrester T, Ellis CG,etal. The erythrocyte as a regulator of vascular tone.AmJPhysiol1995;269(6Pt2):H2155-2161
21 Bunse S, Schmidt M, Hoffmann S,etal. Single cysteines in the extracellular and transmembrane regions modulate pannexin 1 channel function.JMembrBiol2011;244(1):21-33
22 Huang YA, Roper SD. Intracellular Ca2+and TRPM5-mediated membrane depolarization produce ATP secretion from taste receptor cells.JPhysiol2010;588(Pt13):2343-2350
23 Andersen B, Rosenfeld MG. POU domain factors in the neuroendocrine system: lessons from developmental biology provide insights into human disease.EndocrRev2001;22(1):2-35
24 Gerrero MR, McEvilly RJ, Turner E,etal. Brn-3.0: a POU-domain protein expressed in the sensory, immune, and endocrine systems that functions on elements distinct from known octamer motifs.ProcNatlAcadSciUSA1993;90(22):10841-10845
25 Ninkina NN, Stevens GE, Wood JN,etal. A novel Brn3-like POU transcription factor expressed in subsets of rat sensory and spinal cord neurons.NucleicAcidsRes1993;21(14):3175-3182
26 Turner EE, Jenne KJ, Rosenfeld MG. Brn-3.2: a Brn-3-related transcription factor with distinctive central nervous system expression and regulation by retinoic acid.Neuron1994;12(1):205-218
27 Xiang M, Zhou L, Macke JP,etal. The Brn-3 family of POU-domain factors: primary structure, binding specificity, and expression in subsets of retinal ganglion cells and somatosensory neurons.JNeurosci1995;15(7Pt1):4762-4785
28 Xiang M, Gan L, Li D,etal. Role of the Brn-3 family of POU-domain genes in the development of the auditory/vestibular, somatosensory, and visual systems.ColdSpringHarbSympQuantBiol1997;62:325-336
29 Xiang M. Requirement for Brn-3b in early differentiation of postmitotic retinal ganglion cell precursors.DevBiol1998;197(2):155-169
30 Loscher CJ, Hokamp K, Kenna PF,etal. Altered retinal microRNA expression profile in a mouse model of retinitis pigmentosa.GenomeBiol2007;8(11):R248
31 Filali M, Dequen F, Lalonde R,etal. Sensorimotor and cognitive function of a NEFL(P22S) mutant model of Charcot-Marie-Tooth disease type 2E.BehavBrainRes2011;219(2):175-180
32 Koutsis G, Pandraud A, Karadima G,etal. Floroskufi P, Wood NW, Houlden H: Mutational analysis of PMP22, EGR2, LITAF and NEFL in Greek Charcot-Marie-Tooth type 1 patients.ClinGenet2013;83(4):388-391
33 Yum SW, Zhang J, Mo K,etal. A novel recessive Nefl mutation causes a severe, early-onset axonal neuropathy.AnnNeurol2009;66(6):759-770
34 Bhagavati S, Maccabee PJ, Xu W. The neurofilament light chain gene (NEFL) mutation Pro22Ser can be associated with mixed axonal and demyelinating neuropathy.JClinNeurosci2009;16(6):830-831
35 Augusto D, Leteurtre E, De La Taille A,etal. Calretinin: a valuable marker of normal and neoplastic Leydig cells of the testis.ApplImmunohistochemMolMorphol2002;10(2):159-162
36 Lugli A, Forster Y, Haas P,etal. Calretinin expression in human normal and neoplastic tissues: a tissue microarray analysis on 5233 tissue samples.HumPathol2003;34(10):994-1000
37 Bar-Shira Maymon B, Yavetz H, Yogev L,etal. Detection of calretinin expression in abnormal immature Sertoli cells in non-obstructive azoospermia.ActaHistochem2005;107(2):105-112
38 Naeve GS, Ramakrishnan M, Kramer R,etal. a gene induced by neural activity and neurotrophins that promotes neuritogenesis.ProcNatlAcadSciUSA1997;94(6):2648-2653
39 Pahnke J, Mix E, Knoblich R,etal. Overexpression of glial cell line-derived neurotrophic factor induces genes regulating migration and differentiation of neuronal progenitor cells.ExpCellRes2004;297(2):484-494
Application of mathematical algorithm for calculating changes of optic and retina function in mice model of glaucoma
Jing-Kun Liu1, Lin-Bang Wang2, Bing Wang3, Ya-Ling Sheng1, Jing He1, Fen-Ge Meng1
Jing-Kun Liu. Department of Ophthalmology, Xi’an Honghui Hospital, Xi’an 710054, Shaanxi Province, China. 1768697234@qq.com
2016-03-21Accepted:2016-09-30
?AIM: To be one of the primary cause injury to multiple sites of ocular of glaucoma which affects over 70 million people worldwide. We applied data mining techniques, linear and the matrix operations, efficiently calculated the network and estimated the possible function of the “node” genes of the retina and optic of glaucoma, in order to provide new thought and method on the pathogenesis of glaucoma.
glaucoma; biological computing; signaling pathway; biomarker
1(710054) 中國陜西省西安市紅會醫院;2(400016)中國重慶市,重慶醫科大學;3(710077)中國陜西省西安市,中國陜西省衛生監督所
劉靜坤,畢業于西安交通大學,碩士,副主任醫師,研究方向:醫學大數據挖掘及青光眼。
劉靜坤.1768697234@qq.com
2016-03-21
2016-09-30
Liu JK, Wang LB, Wang B,etal. Application of mathematical algorithm for calculating changes of optic and retina function in mice model of glaucoma.GuojiYankeZazhi(IntEyeSci) 2016;16(11):2014-2018
10.3980/j.issn.1672-5123.2016.11.08

