豬囊尾蚴半胱氨酸蛋白酶TsCL-1動態結構柔性分析研究
楊琳,朱月勛,楊艷超,趙琴平,楊力權*
(1.武漢大學基礎醫學院,武漢430071;2.云南省大理白族自治州血吸蟲病防治研究所,云南大理671000;3.大理大學農學與生物科學學院,云南大理671003)
豬囊尾蚴半胱氨酸蛋白酶TsCL-1動態結構柔性分析研究
楊琳1,2,朱月勛3,楊艷超3,趙琴平1,楊力權3*
(1.武漢大學基礎醫學院,武漢430071;2.云南省大理白族自治州血吸蟲病防治研究所,云南大理671000;3.大理大學農學與生物科學學院,云南大理671003)
通過對豬囊尾蚴半胱氨酸蛋白酶TsCL-1的結構模型進行分子動力學模擬,并對其進行模擬過程中的結構穩定性和動態結構柔性分析。結果表明,模擬過程中TsCL-1總體結構相對穩定;分子的結構骨架具有較小的波動,而分子的表面環區和底物結合區域則具有較大的波動和較強的構象柔性。研究結果將為進一步深入研究TsCL-1的動力學行為和功能關系奠定基礎。
半胱氨酸蛋白酶TsCL-1;分子動力學模擬;動態結構柔性;結構功能研究
豬帶絳蟲(Taenia solium),是一種以豬、人和其他雜食或草食動物為中間宿主,以人為唯一終末宿主的帶科絳蟲寄生蟲〔1〕。豬囊尾蚴病(Cysticercosis cellulosae)是由豬帶絳蟲的幼蟲豬囊尾蚴(Cysticercus cellulose)在中間宿主體內肌肉或組織內寄生,而引起的一種疾病〔1-2〕。豬囊尾蚴幼蟲寄生于人體的位置和數量不同,會導致不同的臨床癥狀。該病是一種危害嚴重、分布廣泛的人獸共患寄生蟲病〔1〕,給人類健康帶來嚴重危害〔3〕。
研究表明,由寄生蟲分泌的半胱氨酸蛋白酶(Cysteine Protease,CP)不僅是寄生蟲入侵宿主的因子之一〔4〕,也可能在寄生蟲免疫逃避機制及寄生蟲治療中有著重要作用〔4-6〕。半胱氨酸蛋白酶TsCL-1是由豬囊尾蚴分泌的一種半胱氨酸蛋白酶,屬于半胱氨酸蛋白酶CA蛋白酶中的C1族蛋白酶。由于其晶體結構未被測定,因此對其與宿主相互作用機制還未進行廣泛深入的研究。本課題組之前通過同源模建技術構建了豬囊尾蚴半胱氨酸蛋白酶TsCL-1的三維結構模型并對其進行了結構分析〔7〕。通過對TsCL-1三維結構模型的構建及分析,得以對TsCL-1的空間結構及與功能的關系進行初步研究。但是,由于蛋白質生物學功能的行使是一個動態的過程,其功能的發揮根植于蛋白質的動力學行為〔8〕。因此,僅僅研究蛋白質的靜態空間結構并不能全面地反映蛋白質結構與功能的關系,還需要進一步分析其動力學行為才能深入理解結構-功能間的關系。分子動力學模擬(Molecular dynamics simulations),是目前一種在分子水平通過動力學模擬手段研究生物大分子結構和動力學關系的有效手段〔9-10〕。通過分子動力學模擬可以得到體系內任意時刻各原子的位置和速度,當分子結構穩定且模擬時間足夠長時,便可得到較好的隨時間變化的分子結構動力學軌跡〔11〕。
本文通過對豬囊尾蚴半胱氨酸蛋白酶TsCL-1的結構模型進行分子動力學模擬,并對其動力學模擬軌跡及動態結構特征進行分析,研究其模擬過程中的總體和局部動力學特征和構象柔性,以及這些結構動力學性質對功能發揮的可能影響。本文結果將進一步為在分子水平上揭示豬囊尾蚴蟲半胱氨酸蛋白酶與宿主的相互作用機制和致病機理奠定基礎。
1 材料和方法
1.1 材料本文采用課題組之前通過同源模建技術構建的豬囊尾蚴半胱氨酸蛋白酶TsCL-1的三維結構模型進行動力學模擬〔7〕。TsCL-1的結構模型具備半胱氨酸蛋白酶CA族典型的α∕β折疊模式,結構骨架主要由兩個不同的結構域所組成,一個以α-螺旋為主要結構的L區(L domain),另一個以反平行β片層為主要結構的R區(R domain),催化三聚體(Cys25-His163-Asn183)位于兩個結構域的表面〔12-13〕,結構模型中存在3對二硫鍵,分別由Cys22和Cys65、Cys56和Cys97、Cys156和Cys205構成。見封三圖4。
1.2 動力學模擬方法及步驟本文對半胱氨酸蛋白酶TsCL-1的分子動力學模擬研究由Gromacs 4.6軟件包〔14-15〕完成。具體步驟為:首先將同源模建得到的半胱氨酸蛋白酶TsCL-1結構模型加入一個四面體的溶劑框,接著向溶劑框中加入SPC水分子和抗衡離子,隨后進行能量最小化處理(最陡下降和共軛梯度能量優化方法)以消除蛋白質與水分子間的立體化學沖突,兩次能量最小化所使用的力場為GROMOS96 43a1力場,長程靜電相互作用采用PME算法,非鍵項范德華相互作用的截斷半徑(cut-off)設定為1.4 nm,最陡下降能量優化的收斂力設定為1 000.0 kJ∕(mol·nm),共軛梯度能量優化的收斂力設定為100.0 kJ∕(mol·nm),體系分別在264步和484步達到收斂。然后,對系統進行400 ps的位置抑制性分子動力學模擬,長程靜電相互作用采用PME算法,非鍵項范德華相互作用的截斷半徑設定為1.0 nm,壓力保持在1標準大氣壓。最后,對體系進行生產分子動力學模擬。在生產分子動力學模擬中,長程靜電相互作用采用PME算法,非鍵項范德華相互作用的截斷半徑為0.9 nm,壓力保持在1標準大氣壓,溫度設置為300 K,消除質量中心平移和轉動的頻率為10步,記錄模擬軌跡的頻率為10 ps。生產分子動力學模擬時間為50 ns,模擬所得的軌跡將用于對半胱氨酸蛋白酶TsCL-1結構的動力學行為和結構柔性分析。
2 結果
2.1 模擬過程中TsCL-1的結構穩定性分析本文在分子動力學模擬軌跡的基礎上,通過分析蛋白質的動態結構特征或幾何性質來研究蛋白質的動力學行為和結構柔性特點。在動態結構幾何屬性的分析中,RMSD(root-mean-square deviation)是采樣結構與初始結構所有原子位置平均距離的均方根偏差,該值的大小反應了比較結構間構象差異程度的大小。RMSF(rootmean square fluctuation)是指原子的均方根波動,某個殘基或原子的RMSF值越高,表明該原子具有更強的構象柔性。本文所得到的RMSD和RMSF值分別通過Gromacs軟件包的g_rms和g_rmsf程序計算得到。見圖1。
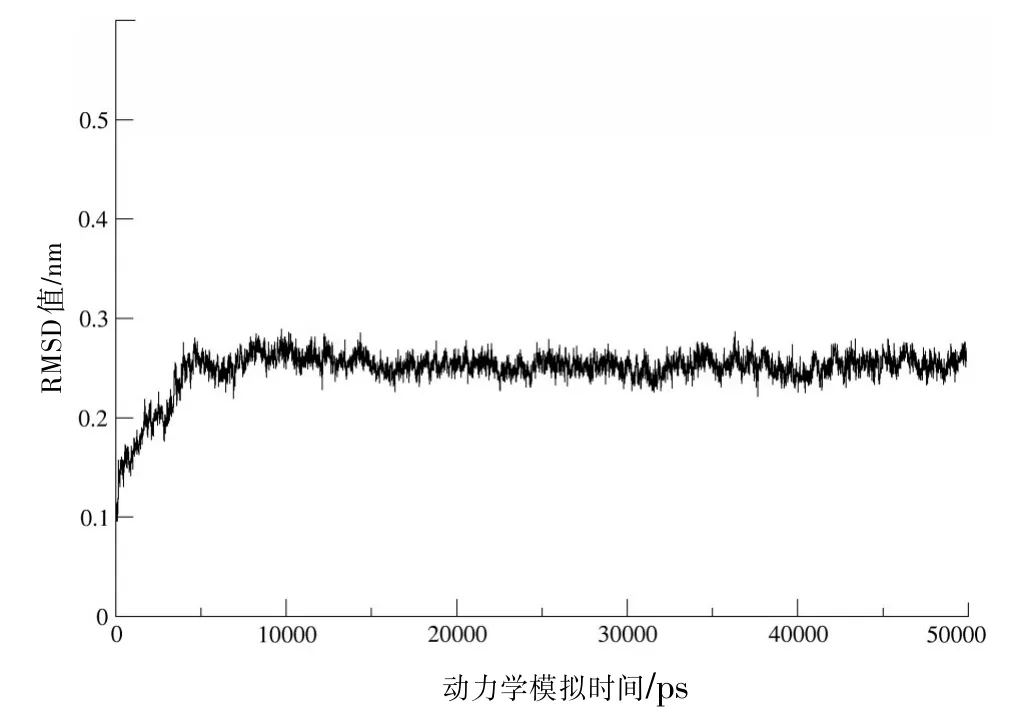
圖1 TsCL-1分子模擬過程中相對于初始結構的RMSD值
圖1中顯示了以模擬時間為函數的TsCL-1蛋白酶模擬過程中骨架原子相對于初始結構的RMSD值變化情況。從圖1中可以看出,RMSD值在前5 ns有一個明顯的上升過程,在10 ns以后RMSD值較為穩定。因此我們選取10~50 ns的模擬軌跡為平衡軌跡,用于之后的動力學特征和結構柔性分析。平衡軌跡較小的波動說明模擬過程中TsCL-1的結構與初始結構沒有較大的差異,模擬過程中TsCL-1的總體結構相對穩定。同時,經過計算,模擬過程中模擬結構的全原子(Allatom)、全骨架(Allbb)和二級結構骨架(SS bb)的RMSD平均值分別為3.32、2.49和1.94?,這一結果說明模擬過程中二級結構最為穩定,而結構的波動主要是由連接二級結構的環區運動引起的。
2.2 模擬過程中TsCL-1的局部構象柔性分析在對TsCL-1模擬過程中的總體結構性質進行分析的基礎上,我們通過分析計算TsCL-1在模擬過程中各氨基酸殘基Cα原子的RMSF值來研究局部構象的柔性。見圖2。從圖2中可以看出,具有最大構象柔性的區域(RMSF>0.15)有:殘基41-45,63-67,99-105,158-159和173-176。其中,殘基63-67是底物結合區域S1和S3的主要組成部分,殘基41-45是位于底物結合區域背面的環區,而殘基99-105,158-159,173-176均為暴露于蛋白質分子表面的環區。具有較大構象柔性的區域(0.10<RMSF<0.15)為:殘基1-3,10-12,19-22,91-98,109-111,138-147和215-216。殘基1-3和215-216主要是N-端和C-末端,殘基19-22是底物結合區域S1的主要組成部分,殘基138-147包含底物結合區域S1’及其附近的區域,殘基19-22,91-98和109-111是位于蛋白分子表面的環區。值得注意的是,在模擬過程中構成TsCL-1結構核心的主要二級結構元件具有相對較低的RMSF值(RMSF<0.10),也就是說在模擬過程中表現出了較高的結構剛性。
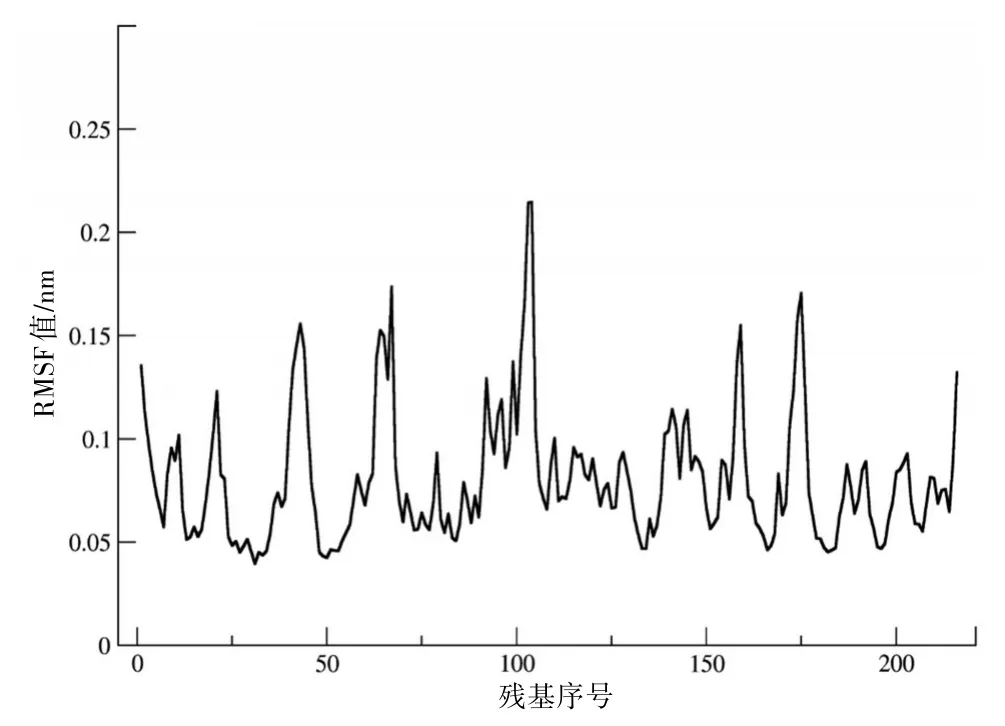
圖2 TsCL-1模擬過程中氨基酸殘基Cα原子的RMSF值
為了能更直觀地觀察出TsCL-1在模擬過程中,蛋白質分子不同結構部位波動的強弱,我們把模擬過程中各氨基酸殘基的CαRMSF值投射到TsCL-1的三維結構上,得到了TsCL-1在模擬過程中不同結構部位的波動差異圖〔16〕。見封三圖5。從封三圖5中可以直觀的看出,模擬過程TsCL-1構象柔性較強(波動較大)的部位主要是表面環區、N-端和C-末端、底物結合部位及與底物結合部位附近的環區。
3 討論
通過對同源模建構建的TsCL-1結構模型進行分析研究發現,TsCL-1為剛性相對較強的球狀蛋白,與其他半胱氨酸蛋白酶CA家族的成員一樣,TsCL-1具備半胱氨酸蛋白酶CA族典型的α∕β折疊模式,保持了家族保守的包含以α-螺旋為主要結構的L區結構域和以反平行β片層為主要結構的R區結構域的結構骨架。我們認為其具備的保守家族骨架折疊模式,保證了它具有堅固的球狀折疊構型。同時,對模擬過程中動力學軌跡的幾何性質計算和分析表明,在模擬過程中TsCL-1呈現出總體上相對穩定的結構狀態。RMSF分析結果表明,模擬過程中組成TsCL-1蛋白酶分子核心結構的二級結構元件具有較小的波動和較強的結構剛性,而暴露于表面的環區和底物結合區域則具有較大的波動的較強的構象柔性。因此,我們認為,在模擬過程中TsCL-1具備的整體結構剛性,特別是其構成折疊骨架核心結構的穩定性保證了它不易被自身或其他蛋白酶水解。同時,目前研究表明,蛋白酶底物結合區構象柔性越大,將可能越有利于底物對酶的構象選擇〔17〕或誘導契合過程〔18〕的發生,同時也有利于已結合底物的調節和定位,以便催化反應能順利進行。因此,我們認為模擬過程中TsCL-1底物結合區域的高構象柔性將有利于其生物學功能的發揮。本文的研究結果將有利于進一步在分子水平上研究和揭示豬囊尾蚴半胱氨酸蛋白酶結構和功能的關系。
〔1〕GARCíA H H,GONZALEZ A E,EVANS C A,et al.Taenia solium cysticercosis〔J〕.The Lancet,2003,362(9383):547-556.
〔2〕PRASAD K N,PRASAD A,VERMA A,etal.Human cysticercosis and Indian scenario:a review〔J〕.J Biosci,2008,33(4):571-582.
〔3〕WILLINGHAM A L,ENGELS D.Control of Taenia solium cysticercosis∕taeniosis〔J〕.Adv Parasitol,2006,61:509-566.
〔4〕BERASAíN P,GO?I F,MCGONIGLE S,etal.Proteinases secreted by Fasciola hepatica degrade extracellular matrix and basementmembrane components〔J〕.J Parasitol,1997,83(1):1-5.
〔5〕HOTEZ P,HAGGERTY J,HAWDON J,etal.Metalloproteases of infective Ancylostoma hookworm larvae and their possible functions in tissue invasion and ecdysis〔J〕.Infect Immun,1990,58(12):3883-3892.
〔6〕LI A H,MOON S U,PARK Y K,et al.Identification and characterization of a cathepsin L-like cysteine protease from Taenia solium metacestode〔J〕.Veterinary Parasitology,2006,141(3):251-259.
〔7〕楊琳,趙琴平,楊力權.豬囊尾蚴半胱氨酸蛋白酶TsCL-1結構的同源模建〔J〕.楚雄師范學院學報,2016,31(3):37-40.
〔8〕HENZLER-WILDMAN K,KERN D.Dynamic personalities ofproteins〔J〕.Nature,2007,450(7172):964-972.
〔9〕KARPLUS M,MCCAMMON J A.Molecular dynamics simulations of biomolecules〔J〕.Nat Struct Biol,2002,9(9):646-652.
〔10〕HANSSON T,OOSTENBRINK C,VAN GUNSTEREN W. Molecular dynamics simulations〔J〕.Curr Opin Struct Biol,2002,12(2):190-196.
〔11〕陳敏伯.計算化學:從理論化學到分子模擬〔M〕.北京:科學出版社,2009.
〔12〕DRENTH J,JANSONIUS J N,KOEKOEK R,et al.The structure of papain〔J〕.Adv Protein Chem,1971,25:79-115.
〔13〕LIGHT A,FRATER R,KIMMEL J R,etal.CurrentStatus of the Structure of Papain:The Linear Sequence,Active Sulfhydryl Group,and the Disulfide Bridges〔J〕.Proc Natl Acad SciUSA,1964,52:1276-1283.
〔14〕VAN DER SPOEL D,LINDAHL E,HESS B,et al.GROMACS:Fast,Flexible,and Free〔J〕.J Comput Chem,2005,26(16):1701-1718.
〔15〕LINDAHL E,HESS B,VAN DER SPOEL D.GROMACS 3.0:a package for molecular simulation and trajectory analysis〔J〕.Journalof Molecular Modeling,2001,7(8):306-317.
〔16〕PETTERSEN E F,GODDARD T D,HUANG C C,et al. UCSF Chimera:a visualization system for exploratory research and analysis〔J〕.J Comput Chem,2004,25(13):1605-1612.
〔17〕TOBID,BAHAR I.Structuralchanges involved in protein binding correlate with intrinsic motions of proteins in the unbound state〔J〕.Proc Natl Acad Sci USA,2005,102(52):18908-18913.
〔18〕KOSHLAND D E.Application of a Theory of Enzyme Specificity to Protein Synthesis〔J〕.Proc Natl Acad Sci USA,1958,44(2):98-104.
Study on the StructuralFlexibility of Cysteine Protease TsCL-1 from Cysticercus Cellulose
Yang Lin1,2,Zhu Yuexun3,Yang Yanchao3,Zhao Qinping1,Yang Liquan3*
(1.Schoolof Basic MedicalSciences,Wuhan University,Wuhan 430071,China;2.DaliInstitute of Schistosomiasis Prevention and Control,Dali,Yunnan 671000,China;3.College of Agriculture and Biological Science,Dali University,Dali,Yunnan 671003,China)
Using the Molecular Dynamics Simulation method,the dynamic structural stability and conformational flexibility of cysteine protease TsCL-1 from Cysticercus cellulose have been investigated.The results indicate that TsCL-1 contains an overall structural stability during the simulation.The results also indicate that the structural core of TsCL-1 shows a small fluctuation amplitude,whereas most of the surface-exposed loops and the substrate binding sites experience significant conformational fluctuations and exhibit a highly structural flexibility.The results of this article will provide a solid basis for further studying the relationship between dynamic structuralfeatures and functions ofTsCL-1.
Cysteine protease TsCL-1;molecular dynamics simu?lations;dynamic structural flexibility;structure and function relationship
Q6
A
2096-2266(2017)06-0055-04
10.3969∕j.issn.2096-2266.2017.06.013
(責任編輯李楊)
大學生科研基金資助項目(KYSX201615)
2016-12-07
2017-03-30
楊琳,主治醫師,主要從事寄生蟲診治研究.
*通信作者:楊力權,副教授,博士.

