嬰兒糞便長雙歧桿菌的分離與多樣性分析
張秋雪,劉曉嬋,朱宗濤,萬 峰,孫思睿,賈芳芳,孟祥晨*
嬰兒糞便長雙歧桿菌的分離與多樣性分析
張秋雪,劉曉嬋,朱宗濤,萬 峰,孫思睿,賈芳芳,孟祥晨*
(東北農業大學 乳品科學教育部重點實驗室,黑龍江 哈爾濱 150030)
本研究旨在分析嬰兒糞便長雙歧桿菌的多樣性,采用改良MRS培養基從哈爾濱地區健康嬰兒糞便中分離雙歧桿菌,采用生理生化實驗結合16S rDNA和熱應激蛋白60基因(heat-shock protein 60,hsp60)同源性分析鑒定分離株,利用同源性分析及隨機擴增多態性DNA(random amplified polymorphism DNA,RAPD)、多位點序列分型(multilocus sequence typing,MLST)技術進一步分析長雙歧桿菌多態性。實驗從11 個嬰兒體內分離得到18 株厭氧的細菌菌株,經形態學分析、生理生化實驗、16S rDNA測序及hsp60測序實驗發現,其中7 株為長雙歧桿菌長亞種,3 株為長雙歧桿菌嬰兒亞種。RAPD和MLST結果表明:7 株長雙歧桿菌長亞種為6 個基因型,3 株長雙歧桿菌嬰兒亞種為2 個基因型,上述結果說明不同人源嬰兒糞便長雙歧桿菌基因型差異較大。
長雙歧桿菌;熱應激蛋白60基因(hsp60);隨機擴增多態性DNA(RAPD);多位點序列分型(MLST)
雙歧桿菌(Bifidobacterium)作為腸道有益微生物在保持機體健康方面起重要作用[1-2],嬰兒出生后,來自出生環境、母乳、后天環境的微生物共同塑造了嬰兒腸道菌群[3-5],母乳喂養的嬰兒腸道中雙歧桿菌占優勢,但是不同個體雙歧桿菌種類差異較大,雙歧桿菌多樣性與嬰兒健康之間的關系目前還不十分清楚,因此對嬰兒腸道雙歧桿菌多樣性進行分析有利于深入了解雙歧桿菌在嬰兒健康中的作用。
人源長雙歧桿菌(B. longum)包括長雙歧桿菌長亞種(B. longum subsp. longum)和長雙歧桿菌嬰兒亞種(B. longum subsp. infantis)[6]。傳統的生理生化和16S rDNA同源性分析對親緣關系比較近的物種分辨率不高,而高度保守的熱應激蛋白60(heat-shock protein,hsp60)基因,在雙歧桿菌親緣物種鑒定方面則可以提供更為準確的信息[7],能彌補16S rDNA同源性分析的不足。隨機擴增多態性DNA(random amplified polymorphism DNA,RAPD)是研究多樣性經常采用的方法,與之相比多位點序列分型(multilocus sequence typing,MLST)技術可以更為準確地區分分離株的基因型,提供更多的基因信息[8]。一般選擇6~8 個編碼蛋白的管家基因進行測序,分析等位基因圖譜同時進行聚類分析,根據位點序列的不同賦予不同菌株序列類型(sequence typing,ST),達到對菌株進行多位點精確區分[9]。
本研究擬采集3~6 個月純母乳喂養嬰兒糞便,采用純培養技術分離其中的長雙歧桿菌,進一步利用16S rDNA、hsp60、RAPD和MLST技術分析分離得到的長雙歧桿菌多樣性,為深入研究長雙歧桿菌在嬰兒健康中的作用和功能提供理論支持。
1 材料與方法
1.1 材料與試劑
1.1.1 微生物菌株
長雙歧桿菌長亞種ATCC15707作為標準菌株,購自中國微生物菌種保藏中心。
1.1.2 培養基與試劑
改良MRS(modified MRS,mMRS)培養基(每升添加含0.05% L-半胱氨酸鹽酸鹽和5%乳糖的MRS培養基) 青島海博生物技術有限公司。
預還原液:L-半胱氨酸鹽酸鹽3 g/L,大豆蛋白胨2 g/L,121 ℃滅菌15 min[10]。
糞便稀釋液:Na2HPO46 g/L;KH2PO44.5 g/L;L-半胱氨酸鹽酸鹽0.5 g/L;吐溫80 0.5 g/L[11]。
TIANGEN細菌基因組DNA提取試劑盒、DNA Marker、dNTPs、Taq DNA聚合酶 天根生化科技有限公司;引物 吉林省庫美生物科技有限公司;L-半胱氨酸鹽酸鹽 天津光復精細化工研究所;乳糖 天津科密歐化學試劑有限公司;瓊脂糖 美國Sigma公司。
1.2 儀器與設備
LJQ-F2厭氧培養罐 自行設計;SPX-150B生化培養箱 上海佳勝實驗設備有限公司;BCN1360型生物潔凈工作臺 北京東聯哈爾儀器制造;微量臺式離心機美國Beckman公司;光學顯微鏡 麥克奧迪實業集團有限公司;DYY-10C型電泳儀 北京市六一儀器廠;凝膠成像系統 美國UVP公司。
1.3 方法
1.3.1 糞便采集與菌株的分離純化
采集哈爾濱地區3~6 個月母乳喂養的健康嬰兒的糞便,糞便采集前嬰兒均未服用過抗生素類藥物,無益生菌服用史,無胃腸病史。取新鮮嬰兒糞便立刻放入加有預還原液的無菌取樣管中,并放入冰盒內迅速帶回實驗室進行分離。
將1 g新鮮糞便用糞便稀釋液梯度稀釋,在10-4、10-5、10-6稀釋度下取0.1 μL涂布于mMRS培養基中,37 ℃厭氧培養48~72 h后挑取凸起、光滑、邊緣水潤的乳白色或白色菌落鏡檢。將呈V、Y或兩端鈍圓的桿狀形態菌落劃線于mMRS培養基中,進行有氧和厭氧培養,剔除需氧和兼性厭氧菌。選取僅厭氧條件生長并且革蘭氏染色為陽性的菌落,進一步劃線厭氧培養純化,直至鏡檢為純菌。
1.3.2 生理生化實驗
將1.3.1節中獲得的分離株分別進行過氧化氫酶實驗、明膠液化實驗、運動性實驗、氧化酶實驗、吲哚實驗、硝酸鹽還原實驗[12-13]。選擇過氧化氫酶、明膠液化、氧化酶實驗、吲哚和硝酸鹽還原實驗結果均為陰性,并且不運動的菌株進行基因同源性分析。
1.3.3 分離菌株基因同源性分析
1.3.3.1 基因組DNA提取
按照細菌基因組DNA提取試劑盒步驟提取分離株基因組DNA。
1.3.3.2 分離菌株16S rDNA同源性分析
16S rDNA擴增反應總體系50 μL:超純水35.5 μL;5×Taq DNA polymerase buffer(含Mg2+)5 μL;dNTP(2.5 mol/L)4 μL;27F(10 μmol/L)2 μL;1429R(10 μmol/L)2 μL;Taq DNA聚合酶0.5 μL;DNA模板1 μL。其中引物序列27F為5’-AGAGTTTGATCCTGGCTCAG-3’,1492R為5’-GGTTACCTTGTTACGACTT-3’[14]。
16S rDNA擴增反應條件:94 ℃預變性4 min;30 個循環包括:94 ℃變性30 s,53 ℃退火1 min,72 ℃延伸1 min;最后72 ℃延伸7 min。
1.3.3.3 分離菌株hsp60同源性分析
hsp60基因擴增反應體系同16S rDNA基因擴增體系。其中上游引物為hspF3(5’-ATCGCCAAGGAGATCGAGCT-3’),下游引物為hspR4(5’-ATCGCCAAGGAGATCGAGCT-3’)[15]。
hsp60基因擴增反應條件:94 ℃預變性5 min;第1階段4 個循環包括94 ℃變性60 s,62 ℃退火60 s,72 ℃延伸90 s;第2階段21 個循環包括94 ℃變性60 s,60 ℃退火60 s,72 ℃延伸90 s;第3階段15 個循環包括94 ℃變性60 s,58 ℃退火60 s,72 ℃延伸90 s;最后72 ℃延伸7 min[15]。
1.3.3.4 基因擴增產物分析
聚合酶鏈式反應(polymerase chain reaction,PCR)完畢之后,將PCR產物進行1%的瓊脂糖凝膠電泳。電泳后,將凝膠置于紫外燈下進行觀察。若16S rDNA擴增產物獲得約1 500 bp的條帶,hsp60擴增產物獲得約650 bp的條帶,且條帶單一清晰無雜帶,擴增產物送吉林省庫美生物科技有限公司進行測序。所得序列運用MEGA 6軟件,采用Neighbor-Joining方法,自展值分析設置1 000 次重復取樣,對菌株構建系統發育進化樹。
1.3.4 分離菌株多樣性分析
1.3.4.1 RAPD分析
RAPD分析時選擇4對引物:A18(5’-AGGTGACCGT-3’)、L16(5’-AGGTTGCAGG-3’)、E14( 5’-CCAAGCTTCC-3’) 、 OPU10(5’-ACCTCGGCAC-3’)。
RAPD反應總體系25 μL:超純水17 μL;5×Taq DNA polymerase buffer(含Mg2+)3 μL;dNTP(2.5 mol/L)2 μL;引物(10 μmol/L)1 μL;Taq DNA聚合酶1 μL;DNA模板1 μL。
RAPD反應條件:94 ℃預變性3 min;40個循環包括:94 ℃變性1 min,36 ℃退火30 s,72 ℃延伸70 s;最后72 ℃延伸10 min。將PCR產物于1.5%的瓊脂糖進行凝膠電泳,在紫外下觀察擴增條帶圖譜[16]。
1.3.4.2 MLST分析
等位基因擴增體系與16S rDNA基因擴增體系相同。其中每個等位基因所用上下游引物信息如表1所示。

表1 長雙歧桿菌MLST擴增引物Table 1 MLST primers for Bifidobacterium longum
等位基因擴增條件:95 ℃預變性5 min;30 個循環包括:95 ℃變性30 s,55 ℃或60 ℃退火30 s,72 ℃延伸1 min;最后72 ℃延伸10 min。其中對于等位基因ileS和rpoB退火溫度為55 ℃,等位基因clpC、fusA、gyrB、purF、rplB退火溫度為60 ℃[17-18]。
PCR完畢之后,將PCR產物進行1%瓊脂糖凝膠電泳。電泳后,將凝膠置于紫外燈下進行觀察。若clpC、fusA、gyrB、ileS、PurF、rplB、rpoB條帶單一清晰無雜帶,送檢測序。
為提高多位點分型效果的準確性,將等位基因進行雙向測序,采用Chromas軟件打開測序峰圖驗證序列的正確性,并去除測序起始端與引物結合不穩定的序列[18]。經測序及數據處理后,用于MLST分型分析的序列長度分別為:clpC:603 bp;fusA:666 bp;gyrB:627 bp;ileS:489 bp;purF:591 bp;rplB:357 bp;rpoB:501 bp[17-18]。
2 結果與分析
2.1 分離菌株形態及生理生化實驗結果
2.1.1 分離菌株的菌落菌體形態
從11 個健康母乳喂養的嬰兒糞便中分離得到18 株疑似菌株。菌落乳白色或白色、凸起、光滑且邊緣水潤。18 株菌均為革蘭氏染色陽性,菌體排列不規則,菌體形態為桿狀,且桿狀末端分叉,呈V或Y字、頓圓、啞鈴等形態。
2.1.2 生理生化實驗結果
18 株分離株均為專性厭氧菌,過氧化氫酶實驗、明膠液化實驗、氧化酶實驗、吲哚實驗、硝酸鹽還原實驗均為陰性,無運動性。
2.2 分離菌株的基因同源性分析
2.2.1 16S rDNA同源性

圖1 分離菌株16S rDNA 的PCR電泳圖譜Fig. 1 Electrophotogram of PCR-amplified 16S rDNA from the isolates
18 株分離株PCR擴增產物經瓊脂糖凝膠電泳后,在1500 bp左右處有明亮單一條帶(圖1),滿足測序要求。對18 株分離菌株進行測序,所得序列輸入國家國立生物技術信息中心(National Center for Biotechnology Information,NCBI)中進行比對,結果顯示有10 株菌與長雙歧桿菌的同源性均達到99%。其余8 株分別為兩歧雙歧桿菌2 株、假小連雙歧桿菌1 株、動物雙歧桿菌乳酸亞種4 株、短雙歧桿菌1 株。
選取16S rDNA同源性99%以上的模式菌株序列,以Micrococcus luteus DSM 20030(GenBank序列號:AJ536198)作為16S rDNA基因進化樹的根,對分離得到的長雙歧桿菌菌株構建系統發育樹(圖2)。發現有7 株菌(H3-R1、H9-5、H10-1、H16-2、H20-14、H27-10、H27-13)均與長雙歧桿菌長亞種ATCC15707 16S rDNA親緣關系最近,初步確定為長雙歧桿菌長亞種;3 株菌(H5-19、H5-21、H11)與長雙歧桿菌嬰兒亞種ATCC15697 16S rDNA親緣關系最近,初步確定為長雙歧桿菌嬰兒亞種。

圖2 基于16S rDNA基因序列構建長雙歧桿菌系統發育樹Fig. 2 Phylogenetic tree of B. longum based on 16S rDNA gene sequences
2.2.2 hsp60同源性分析
上述10 株長雙歧桿菌的hsp60基因PCR擴增后電泳,在650 bp左右處均有明亮單一條(圖3),將樣品進行測序。
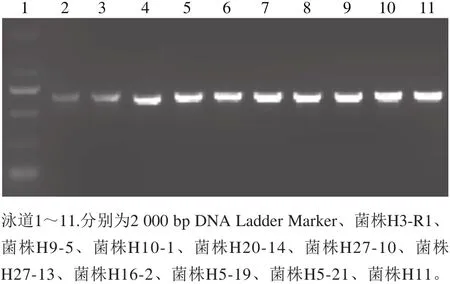
圖3 長雙歧桿菌hsp60基因的PCR電泳結果Fig. 3 Electrophotogram of PCR-amplified hsp60 gene from the B. longum strains
選取hsp60同源性99%以上的模式菌株序列,根據測序結果構建無根進化樹(圖4)。同樣發現,7 株菌(H3-R1、H9-5、H10-1、H16-2、H20-14、H27-10、H27-13)與長雙歧桿菌長亞種JCM1217的親緣關系最近,并結合以上結果判定為長雙歧桿菌長亞種;3 株菌(H5-19、H5-21、H11)與長雙歧桿菌嬰兒亞種JCM1222的親緣關系最近,并結合以上結果判定為長雙歧桿菌嬰兒亞種。
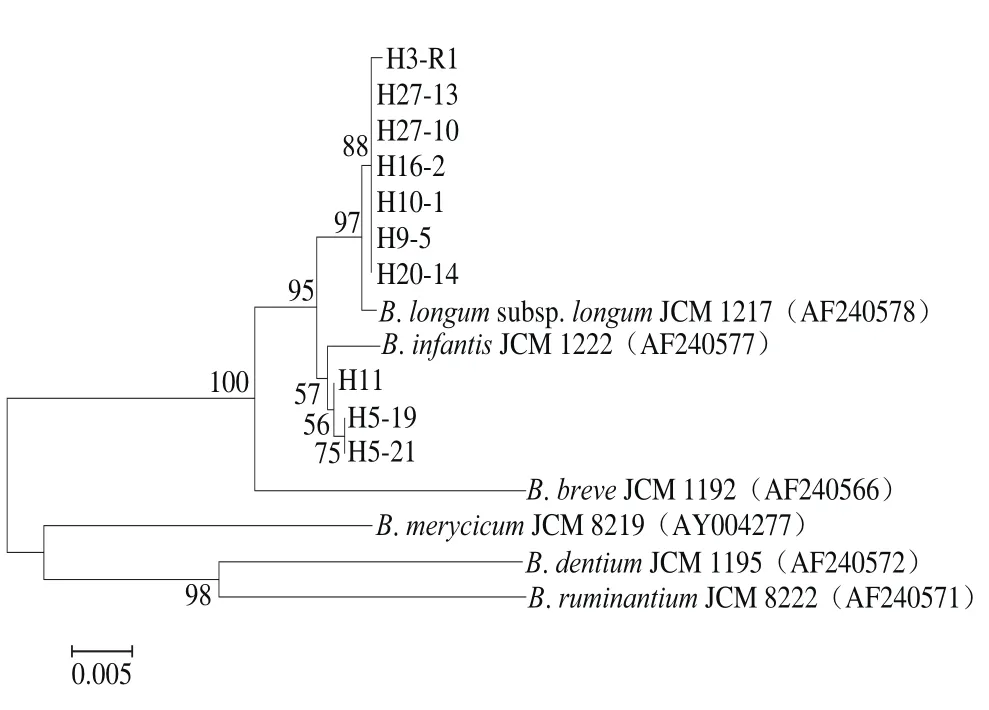
圖4 基于hsp60基因序列構建長雙歧桿菌系統發育樹Fig. 4 Phylogenetic tree of B. longum based on hsp60 gene sequences
2.3 長雙歧桿菌多樣性的分析
2.3.1 RAPD分析結果
本實驗采用4 個隨機引物對10株長雙歧桿菌的基因組DNA進行擴增,實驗重復3 次確保引物具有良好的重復性,以研究其遺傳多態性。擴增產物瓊脂糖凝膠電泳得到多態性圖譜(圖5),不同引物的擴增片段數目為1~7 條之間,片段分子質量大小在0~1 kb之間。綜合4 個隨機引物的擴增圖譜可知,長雙歧桿菌H3-1、H9-5、H10-1、H20-14、H16-2、H11分別具有差異條帶圖譜,屬于不同株型;H27-10和H27-13、H5-19和H5-21具有相同條帶圖譜,屬于同一株型。


圖5 基于引物A18(a)、L16(b)、E14(c)、OPU10(d)的長雙歧桿菌基因組的RAPD圖譜Fig. 5 RAPD profiles of B. longum genome based on primers A18 (a),L16 (b), E14 (c) and OPU10 (d)
2.3.2 MLST的分析結果
2.3.2.1 等位基因電泳及測序
選取7 個等位基因,運用設計好的等位基因引物對長雙歧桿菌等位基因擴增。10 株長雙歧桿菌的7 個等位基因均擴增成功,且有明亮單一條帶(圖6),滿足測序要求送樣品進行測序。

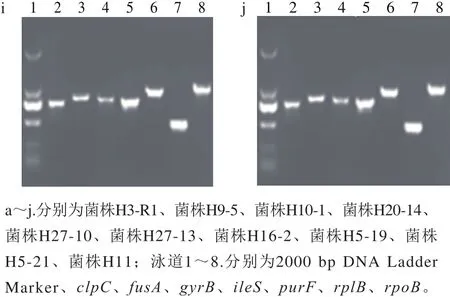
圖6 長雙歧桿菌菌株MLST基因PCR電泳圖Fig. 6 PCR electrophoresis bands of the MLST genes of B. longum
2.3.2.2 長雙歧桿菌的等位基因分型
以ATCC15707為參考菌株,運用7 個等位基因對10株長雙歧桿菌進行MLST分型。去除兩端誤差序列,在7 個等位基因的等位點上,賦予每個不同的等位基因以不同的數值,并指定7 個等位點中,每個不同的等位基因模式為一個ST型。最終將10 株長雙歧桿菌分為8 個ST型,且均與ATCC15707為不同的ST型。

表2 長雙歧桿菌7 個等位基因的MLST分型Table 2 MLST genotyping of B. longum strains based on seven allelic genes
7 個等位基因分型結果如表2所示,7 株長雙歧桿菌長亞種分為6 個基因型,其中菌株H3-R1、H9-5、H10-1、H20-14、H16-2分別屬于不同ST型,而菌株H27-10和H27-13屬于同一ST型。3 株長雙歧桿菌嬰兒亞種分為2個基因型,其中H5-19和H5-21屬于同一ST型,且與H11為不同ST型。
2.3.2.3 長雙歧桿菌等位基因系統發育樹構建
將10 株菌的7 個等位基因序列連接起來(clpC-fusA-gyrB-ileS-purF-rplB-rpoB),拼接長度約為3 834 bp,對菌株構建聯合基因系統發育進化樹,結果如圖7所示,其結果與RAPD結果一致,相對于RAPD技術,MLST可以通過進化樹體現不同株之間進化距離關系。
對所有長雙歧桿菌菌株等位基因序列分型結果可以看出,分離于H5嬰兒個體的H5-19和H5-21菌株為相同ST型,分離于H27嬰兒個體的H27-10和H27-13號菌株為相同ST型。

圖7 長雙歧桿菌菌株等位基因系統發育樹Fig. 7 Phylogenetic tree of B. longum based on allelic genes
3 討 論
本研究在分離菌株分子鑒定中,hsp60基因同源性分析結果與16S rDNA同源性分析結果一致,但hsp60基因同源性更進一步展示出分離株進化距離上的差異[19-20]。從圖4可以看出長雙歧桿菌長亞種H3-R1與其他分離菌株及JCM1217之間進化距離上存在差異;H5-19、H5-21、H11都在長雙歧桿菌嬰兒亞種聚類中,但存在進化距離的差異,且H5-19與H5-21進化距離無差異。此結果顯示hsp60基因序列分析優于16S rDNA基因分析。已有研究表明保守基因hsp60具有高度保守的結構,可以對雙歧桿菌在種或亞種水平上進行鑒定[21],更能有效地揭示出親緣關系較高菌株之間系統發育的差異[22-23]。
長雙歧桿菌RAPD多樣性分析發現,長雙歧桿菌菌株間擴增圖譜存在共有帶,也存在特異性條帶,擴增出的譜帶大小及條數差異可作為區分各長雙歧桿菌菌株的依據[24]。如引物L16擴增譜圖(圖5b)在4 000 bp左右所有長雙歧桿菌均有一條明亮條帶,該條帶可能是長雙歧桿菌種內特征性條帶,在1 000~2 000 bp之間泳道10和11均有一條其他泳道未出現的特異性條帶;引物E14擴增譜圖(圖5c)顯示:泳道6與7、泳道10與11條帶相同,除此之外的其余泳道條帶均不相同。
長雙歧桿菌MLST多樣性分析發現,單個等位基因的進化樹只能反映物種間進化關系,多個基因系統發育分析才能更好地表現物種之間的關系[25-28]。已有研究結果表明長雙歧桿菌在人體中定植的影響因素包括環境和宿主基因型[29],這為解釋同一宿主體內定植的長雙歧桿菌具有相同的ST型提供了理論依據。同時,不同嬰兒個體(H3、H5、H9、H10、H11、H16、H20、H27)的長雙歧桿菌形成了8 個不同的ST型,也表明長雙歧桿菌之間存在較大的遺傳多樣性[30]。
綜上所述,本研究從11 個健康嬰兒體內分離鑒定得到10 株長雙歧桿菌,7 株為長雙歧桿菌長亞種,3 株為長雙歧桿菌嬰兒亞種。多態性分析發現:7 株長雙歧桿菌長亞種為6 個基因型,3 株長雙歧桿菌嬰兒亞種為2 個基因型,說明嬰兒腸道中長雙歧桿菌存在較大的遺傳多樣性。
[1] BOTTACINI F, VENTURA M, VAN SINDEREN D, et al. Diversity,ecology and intestinal function of bifidobacteria[J]. Microbial Cell Factories, 2014, 13(1)∶ 1. DOI∶10.1186/1475-2859-13-S1-S4.
[2] SIVAN A, CORRALES L, HUBERT N, et al. Commensal Bifidobacterium promotes antitumor immunity and facilitates anti–PDL1 efficacy[J]. Science, 2015, 350(6264)∶ 1084-1089. DOI∶10.1126/science.aac4255.
[3] PATEL R M, DENNING P W. Intestinal microbiota and its relationship with necrotizing enterocolitis[J]. Pediatric Research, 2015,78(3)∶ 232-238. DOI∶10.1038/pr.2015.97.
[4] FLORES G E, CAPORASO J G, HENLEY J B, et al. Temporal variability is a personalized feature of the human microbiome[J].Genome Biology, 2014, 15(12)∶ 1. DOI∶10.1186/s13059-014-0531-y.
[5] SAKWINSKA O, MOINE D, DELLEY M, et al. Microbiota in breast milk of Chinese lactating mothers[J]. PLoS ONE, 2016, 11(8)∶e0160856. DOI∶10.1371/journal.pone.0160856.
[6] MATTARELLI P, BONAPARTE C, POT B, et al. Proposal to reclassify the three biotypes of Bifidobacterium longum as three subspecies∶ Bifidobacterium longum subsp. longum subsp.nov., Bifidobacterium longum subsp. infantis comb. nov. and Bifidobacterium longum subsp. suis comb. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(4)∶767-772. DOI∶10.1099/ijs.0.65319-0.
[7] JIAN W, ZHU L, DONG X. New approach to phylogenetic analysis of the genus Bifidobacterium based on partial HSP60 gene sequences[J].International Journal of Systematic and Evolutionary Microbiology,2001, 51(5)∶ 1633-1638. DOI∶10.1099/00207713-51-5-1633.
[8] MAIDEN M C J, VAN RENSBURG M J J, BRAY J E, et al. MLST revisited∶ the gene-by-gene approach to bacterial genomics[J].Nature Reviews Microbiology, 2013, 11(10)∶ 728-736. DOI∶10.1038/nrmicro3093.
[9] PéREZ-LOSADA M, CABEZAS P, CASTRO-NALLAR E, et al. Pathogen typing in the genomics era∶ MLST and the future of molecular epidemiology[J]. Infection, Genetics and Evolution, 2013,16∶ 38-53. DOI:10.1016/j.meegid.2013.01.009.
[10] BEVILACQUA L, OVIDI M, DI MATTIA E, et al. Screening of Bifidobacterium strains isolated from human faeces for antagonistic activities against potentially bacterial pathogens[J]. Microbiological Research, 2003, 158(2)∶ 179-185. DOI:10.1078/0944-5013-00192.
[11] 高鵬飛, 孫志宏, 麻士衛, 等. 蒙古族兒童源益生特性雙歧桿菌的篩選及鑒定[J]. 微生物學報, 2009, 49(2)∶ 210-216.
[12] TURRONI F, PEANO C, PASS D A, et al. Diversity of bifidobacteria within the infant gut microbiota[J]. PLoS ONE, 2012, 7(5)∶ e36957.DOI∶10.1371/journal.pone.0036957.
[13] 凌代文, 東秀珠. 乳酸菌的分離鑒定及實驗基礎[M]. 北京∶ 中國輕工業出版社, 1999∶ 142-143.
[14] MORITA H, NAKANO A, ONODA H, et al. Bifidobacterium kashiwanohense sp. nov., isolated from healthy infant faeces[J].International Journal of Systematic and Evolutionary Microbiology,2011, 61(11)∶ 2610-2615. DOI:10.1099/ijs.0.024521-0.
[15] MODESTO M, MICHELINI S, STEFANINI I, et al. Bifidobacterium aesculapii sp. nov., from the faeces of the baby common marmoset(Callithrix jacchus)[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(8)∶ 2819-2827. DOI:10.1099/ijs.0.056937-0.
[16] 王濤, 孟祥晨. 一株來源于健康成人糞便雙歧桿菌的分離及鑒定[J].東北農業大學學報, 2008, 39(7)∶ 107-111.
[17] DELéTOILE A, PASSET V, AIRES J, et al. Species delineation and clonal diversity in four Bifidobacterium species as revealed by multilocus sequencing[J]. Research in Microbiology, 2010, 161(2)∶ 82-90.DOI:10.1016/j.resmic.2009.12.006.
[18] DAN T, LIU W, SUN Z, et al. A novel multi-locus sequence typing(MLST) protocol for Leuconostoc lactis isolates from traditional dairy products in China and Mongolia[J]. BMC Microbiology, 2014, 14(1)∶ 1.DOI:10.1186/1471-2180-14-150.
[19] TSUCHIDA S, TAKAHASHI S, NGUEMA P P, et al. Bifidobacterium moukalabense sp. nov. isolated from the faeces of wild west lowland gorilla(Gorilla gorilla gorilla)[J]. International Journal of Systematic &Evolutionary Microbiology, 2014, 64(Pt 2)∶ 449-455. DOI:10.1099/ijs.0.055186-0.
[20] KIM M S, ROH S W, BAE J W. Bifidobacterium stercoris sp. nov.,isolated from human faeces[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60(12)∶ 2823-2827. DOI:10.1099/ijs.0.019943-0.
[21] CHOI J H, LEE K M, LEE M K, et al. Bifidobacterium faecale sp. nov., isolated from human faeces[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(9)∶ 3134-3139.DOI:10.1099/ijs.0.063479-0.
[22] ZHU L, LI W, DONG X. Species identification of genus Bifidobacterium based on partial HSP60 gene sequences and proposal of Bifidobacterium thermacidophilum subsp. porcinum subsp. nov[J].International Journal of Systematic and Evolutionary Microbiology,2003, 53(5)∶ 1619-1623. DOI:10.1099/ijs.0.02617-0.
[23] BAFFONI L, STENICO V, STRAHSBURGER E, et al. Identification of species belonging to the Bifidobacterium genus by PCR-RFLP analysis of a hsp60 gene fragment[J]. BMC Microbiology, 2013,13(1)∶ 1. DOI:10.1186/1471-2180-13-149.
[24] BUNESOVA V, KILLER J, VLKOVA E, et al. Isolation and characterization of bifidobacteria from ovine cheese[J]. International Journal of Food Microbiology, 2014, 188∶ 26-30. DOI:10.1016/j.ijfoodmicro.2014.07.001.
[25] VENTURA M, CANCHAYA C, DEL CASALE A, et al. Analysis of bifidobacterial evolution using a multilocus approach[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(12)∶2783-2792. DOI:10.1099/ijs.0.64233-0.
[26] UNDERWOOD M A, GERMAN J B, LEBRILLA C B, et al.Bifidobacterium longum subspecies infantis∶ champion colonizer of the infant gut[J]. Pediatric Research, 2014, 77(1/2)∶ 229-235.DOI∶10.1038/pr.2014.156.
[27] GADAGKAR S R, ROSENBERG M S, KUMAR S. Inferring species phylogenies from multiple genes∶ concatenated sequence tree versus consensus gene tree[J]. Journal of Experimental Zoology Part B∶ Molecular and Developmental Evolution, 2005, 304(1)∶ 64-74.DOI:10.1002/jez.b.21026.
[28] WU Y C, RASMUSSEN M D, BANSAL M S, et al. TreeFix∶ statistically informed gene tree error correction using species trees[J]. Systematic Biology, 2013, 62(1)∶ 110-120. DOI:10.1093/sysbio/sys076.
[29] ZHANG M, HANG X, TAN J, et al. The host genotype and environment affect strain types of Bifidobacterium longum subsp. longum inhabiting the intestinal tracts of twins[J]. Applied and Environmental Microbiology,2015, 81(14)∶ 4774-4781. DOI:10.1128/AEM.00249-15.
[30] MAKINO H, KUSHIRO A, ISHIKAWA E, et al. Transmission of intestinal Bifidobacterium longum subsp. longum strains from mother to infant determined by multilocus sequencing typing and amplified fragment length polymorphism[J]. Cancer Letters, 2011, 71(1/2/3)∶ 19-24.DOI:10.1128/AEM.05346-11.
Isolation and Diversity Analysis of Bifidobacterium longum from Infant Faeces
ZHANG Qiuxue, LIU Xiaochan, ZHU Zongtao, WAN Feng, SUN Sirui, JIA Fangfang, MENG Xiangchen*
(Key Laboratory of Dairy Science, Ministry of Education, Northeast Agricultural University, Harbin 150030, China)
The aim of this experiment was to study the diversity of Bifidobacterium longum isolated from infant feces.Using modified MRS medium, bifidobacterial strains were isolated from the feces of healthy infants in Harbin. To identify and analyze the diversity of B. longum strains, we used physiological and biochemical experiments, 16S rDNA homology analysis, heat-shock protein 60 gene (hsp60) homology analysis, random amplified polymorphism DNA (RAPD) and multilocus sequence typing (MLST). Eighteen anaerobic strains were isolated, among which, 7 were identified as B. longum subsp. longum, and 3 as B. longum subsp. infantis according to morphology, physical and biochemical properties, 16S rDNA homology analysis and hsp60 homology analysis. The results of RAPD and MLST showed that the 7 strains of B. longum subsp. longum belonged to 6 genotypes while the 3 strains of B. longum subsp. infantis belonged to 2 genotypes. Overall,these results showed that there were differences in the diversity of B. longum isolated from infant feces.
Bifidobacterium longum; heat-shock protein 60 gene (hsp60); random amplified polymorphism DNA (RAPD);multilocus sequence typing (MLST)
DOI∶10.7506/spkx1002-6630-201724002
TS201.3
A
1002-6630(2017)24-0008-07
張秋雪, 劉曉嬋, 朱宗濤, 等. 嬰兒糞便長雙歧桿菌的分離與多樣性分析[J]. 食品科學, 2017, 38(24)∶ 8-14. DOI∶10.7506/spkx1002-6630-201724002. http∶//www.spkx.net.cn
ZHANG Qiuxue, LIU Xiaochan, ZHU Zongtao, et al. Isolation and diversity analysis of Bifidobacterium longum from infant faeces[J]. Food Science, 2017, 38(24)∶ 8-14. (in Chinese with English abstract) DOI∶10.7506/spkx1002-6630-201724002.http∶//www.spkx.net.cn
2016-11-25
國家自然科學基金面上項目(31671917)
張秋雪(1992—),女,碩士研究生,研究方向為食品微生物與生物技術。E-mail:zqx823@163.com
*通信作者:孟祥晨(1970—),女,教授,博士,研究方向為食品微生物與生物技術。E-mail:xchmeng@163.com

