Diaphorobacter sp.PDB3菌好氧反硝化脫氮特性
許 濤,王國英,岳秀萍 (太原理工大學環境科學與工程學院,山西 太原 030024)
反硝化是氮循環的重要組成部分,環境中的氮元素通過生化作用進入到生物圈,再通過微生物反硝化作用以氮氣的形式釋放到空氣中[1].現代反硝化理論認為反硝化菌通常是一類兼性厭氧菌,當 O2充足時,反硝化菌氧化分解有機物是以分子態氧作為最終電子受體.在缺氧或無氧條件下(污水處理中通常在 DO≤0.5mg/L[2]),反硝化菌則利用有機物作為碳源和電子供體來提供能量并以硝酸鹽和亞硝酸鹽作為電子受體.
現已發現的好氧反硝化菌約 50多個屬,130多個種[3],其中,環境中最普遍存在的好氧反硝化細菌為不動桿菌屬(Acinetobacter)[4]、紅球菌屬(Rhodococcus)[5]、產堿桿菌屬(Alcaligenes)[6]和農桿菌屬(Agrobacterium)[7]等.這些細菌通過異養生長去除部分 NO3?-N同時利用硝酸鹽還原酶完成 NO3?-N 的反硝化.目前關于好氧反硝化菌的研究多集中于篩選、分離、鑒定和搖床內研究菌種性能,并未有在發酵罐內研究其特性的文章.發酵罐具有通氣量可控、轉速可調、在線監測等一般搖床不具備的優勢,因此探究好氧反硝化菌在發酵罐內的生化特征很有必要.
本課題組前期從太原市煤氣化焦化廠生物池好氧段污泥中篩選分離得到一株反硝化菌Diaphorobacter sp. PDB3[8],并在搖床內對該菌反硝化特征進行了研究,但尚未在發酵罐內研究該菌好氧反硝化特征.本文首先在 3L發酵罐內研究比較不同碳源對 PDB3菌好氧反硝化特性的影響,采用響應面法對影響 PDB3菌好氧反硝化條件(溫度、pH值、發酵罐轉速、碳氮比)進行優化.最后通過氮平衡法和 RT-qPCR法分析了PDB3菌好氧反硝化過程中的脫氮途徑和脫氮基因的表達水平,為 PDB3菌的實際應用提供一定的理論依據.
1 材料與方法
1.1 材料
1.1.1 菌株 好氧反硝化菌 Diaphorobacter sp.PDB3由本課題組篩選保藏[8].
1.1.2 培養基 LB培養基(g/L):NaCl 10.0,酵母浸粉5.0,胰蛋白胨10.0, pH值7.2,用于活化與富集菌株.
好氧反硝化培養基(DM,g/L):KH2PO41.0,Na2HPO42.0, MgSO4·7H2O 0.1, KNO31.0,微量元素溶液2.00mL[9],加蒸餾水溶解,實驗中所有的培養基和實驗器具均需在 121℃高壓蒸汽滅菌器中滅菌20min.
1.2 實驗方法
1.2.1 種子培養 將經甘油冷凍保藏的菌種常溫放置解凍,取1%量接種于100mL LB液態培養基中,培養基用250mL錐形瓶置于搖床內振蕩培養15h,培養條件為120r/min、30℃.
1.2.2 碳源影響 在 DM 培養基中使用葡萄糖、醋酸鈉、丙酮酸鈉、檸檬酸鈉、苯酚和蔗糖作為碳源.通過固定C/N比(W/W)為8和恒定硝態氮濃度(NO3--N,138.6mg/L)來確定每個碳源的添加量.按 3%接種量將菌液轉移到裝有 2L DM培養基的發酵罐(上海保興全自動發酵罐)中,在30℃、300r/min下培養.定期收集10mL試樣用于測定細胞密度和化學分析.
1.2.3 優化試驗條件 響應面法(RSM)[10]用于研究溫度、pH值、發酵罐轉速、碳氮比等4個因素對 PDB3菌好氧反硝化活動的影響.控制不同溫度、pH值、轉速、碳氮比等因素,分批發酵.根據Box-Behnken設計確定4個獨立變量的水平,進行30個實驗(表2).根據實驗數據擬合出響應面數學模型,最終確定 PDB3菌在發酵罐內好氧反硝化最佳條件,并進行實驗驗證.實驗數據分析借助Design Expert 8.0.6軟件.
1.2.4 氮平衡分析 在 RSM 分析獲得的優化條件下發酵培養 12h,發酵液離心后取上清液測TN、NH4+-N、NH2OH-N、NO3--N、NO2--N,取沉淀測胞內氮.
1.2.5 RT-qPCR分析 通過實時熒光定量聚合酶鏈式反應(RT-qPCR)測定PDB3菌株中脫氮基因 napA、nirS、nosZ、cnorB的表達水平.所有樣品總RNA提取和互補DNA(cDNA)合成分別用 RNAprep Bacteria Kit和 Fast Quant RT Kit進行.16S rRNA基因用基因組 DNA提取試劑盒(Tiangen Biotech Co,Ltd,China)提取 napA、nirS、cnorB、nosZ和V3[11-14]的引物序列和PCR擴增片段的長度見表1,16S rRNA的V3區被證明是合適的管家基因(housekeeping gene)[15],其作為內參基因來校正 cDNA量的差異帶來的表達量差異.對含有脫氮基因的質粒進行梯度稀釋并分別同時對內參基因和脫氮基因做標準曲線,同時對各個樣本中內參基因和脫氮基因進行擴增,然后從各自標準曲線上計算待測樣本中內參基因和脫氮基因的拷貝數.qPCR的總體積為50μL,含有 2μL DNA 模板,15μL Taq PCR Master Mix(Tiangen Biotech Co,Ltd,China),正向和反向引物各 2μL(10μmol/L)和 29μL ddH2O.qPCR 系統(Applied Biosystems 7300)反應在以下條件進行[15]: 95℃下預變性10min,接著完成40個循環,每個循環包括 95℃下變性 30s,56℃下(nosZ和nirS)或 60℃下(napA、cnorB 和 V3)退火 30s,最后在72℃下延伸30s.40個循環過后在72℃下延伸10min.

表1 引物序列和PCR擴增片段的長度Table 1 Sequence of the primers and length of the PCR amplification segment
1.2.6 測定與分析方法[16]菌液濃度的測定采用可見分光光度法,取發酵液測定菌液的吸光度(OD600)值;根據細胞干重標準曲線將吸光度值轉換為細胞干重;TOC和 TN用 TOC/TN分析儀(TOC-VCpH/TNM-1,日本島津)測量;胞內氮用元素分析儀(EA3000,意大利歐維特)測量;有機氮等于TN減去NO3--N、NO2--N、NH4+-N 之和;NH2OH-N測定參照文獻[17]測定采用納氏試劑分光光度法;NO3?-N 測定采用酚二磺酸分光光度法;測定采用N-(1-萘基)-乙二胺光度法.
吸光度(OD)值看成是時間的函數,細胞比生長速率(μ)通過對生長曲線線性階段的擬合來確定.由于硝化產物的脫氮是不可避免的并且難以分別檢測它們的活性[7],故用總氮去除活性來表示在不同條件下的好氧反硝化脫氮比速率(ν),方程式如下:

式中:s是 TN的濃度,mg/L; x為微生物濃度,mg/L;t是反應時間,h.
好氧反硝化過程中 4種脫氮基因(napA、nirS、nosZ、cnorB)的表達水平進行歸一化處理來校正cDNA量的差異帶來的表達量差異.

2 結果與分析
2.1 碳源影響
PDB3菌在利用不同碳源時對總氮去除率和細胞比生長速率的影響如圖 1所示,對于好氧反硝化菌來說,菌株對不同碳源的利用率差別很大.實驗的 6個碳源中,PDB3菌在利用苯酚和蔗糖時總氮去除率和細胞比生長速率相對較低,表明它們均不能被該細菌很好利用.PDB3菌最初是以苯酚為底物從焦化廢水中篩選出來的[8,25],發現 PDB3菌以其他碳源為底物均比以苯酚為底物時的反硝化效果要好.PDB3菌在利用丙酮酸鈉和檸檬酸鈉時總氮去除率均相對較高,但細胞比生長速率存在明顯差異,相比之下,檸檬酸鈉作為 PDB3菌的碳源更有利于菌株生長,這與王曉靜等[18]研究確定檸檬酸鈉為一株異養硝化-好氧反硝化菌脫氮的最佳碳源的結果一致.

圖1 不同碳源對PDB3菌總氮去除率和細胞比生長速率的影響Fig.1 Effects of various carbon sources on the specific growth rate and total nitrogen removal by strain PDB3
2.2 響應面分析法確定影響因素的最佳條件
按照響應面分析法中的 Box-Behnken中心組合設計原理,對影響菌株 PDB3好氧反硝化的4個因素即溫度、pH值、轉速、碳氮比(C/N)進行優化,以菌種 PDB3好氧反硝化脫氮比速率(ν)為響應值,進行四因素三水平的響應面設計,結果見表 2.利用軟件對實驗數據進行二次多項回歸擬合得到模型方程:
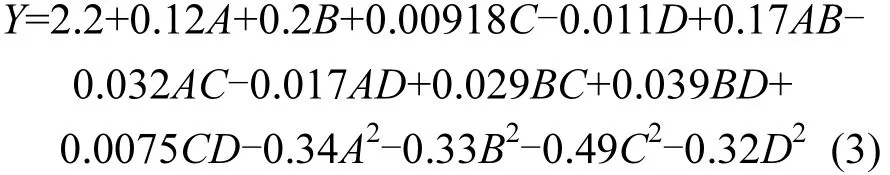
式中:Y為菌種 PDB3好氧反硝化脫氮比速率值;A、B、C、D分別為pH值、碳氮比、溫度、轉速的值.

表2 Box-Behnken試驗設計及結果Table 2 Box-Behnken design and experimental results
從Box-Behnken實驗回歸分析結果(表3)可得出,模型的相關系數 R2=0.9975,表明實驗值與預測值之間高度相關.校正決定系數R2adj=0.9952,表明僅有 0.01%的好氧反硝化比速率值的變異不能被本模型解釋.失擬項 0.3633,差異不顯著;因此實測值能夠被本回歸模型很好的擬合,即可以用本模型來對 PDB3菌在發酵罐內反硝化效果進行分析和預測.

表3 Box-Behnken實驗回歸分析結果Table 3 Results of regression analysis of the Box–Behnken design
由表3可知,模型方程的一次項A、B、C、D(P<0.05)均影響顯著;二次項 A2、B2、C2(P<0.0001)影響均極顯著;交互項 AB、AC、BC(P<0.05)影響也均極顯著;交互項AD(P>0.05)影響不顯著;可見各因素對響應值影響并不是單純的線性關系,且對響應值的影響存在某一極值點,這一極值點恰是要尋找的最佳條件.根據模型方程可預測,當pH值、碳氮比、溫度、轉速分別為7.3、8.1、30.1℃、299.9r/min時,PDB3菌在發酵罐內好氧反硝化比速率達到2.25h-1.這與Alcaligenes sp.W1在pH值為7左右反硝化效果最佳[17]的研究結果一致.同時也與 Zhang等[19]和Huang等[20]在研究溶解氧對反硝化影響的結果接近.
根據模型方程作響應面圖,分析pH值、碳氮比、溫度、轉速對PDB3菌好氧反硝化比速率的影響,圖2為4個影響因素相互之間的響應面,每個圖分別代表 2個獨立變量之間的相互作用,此時另外2個變量保持在中心點水平[21].等高線的形狀反映出交互作用的強弱,圓形表示兩者交互作用不顯著,橢圓形則表示兩者交互作用顯著[7].圖2中的(a)、(b)、(d)、(e)、(f)等高線呈橢圓形,而圖2(c)等高線呈近似圓形,表明pH值和C/N、溫度和pH值、溫度和C/N、轉速和C/N及溫度和轉速之間交互作用顯著,而轉速和 pH值之間交互作用不顯著.這與表2中AB、AC、BC、BD、DC值<0.05,而AD值>0.05相對應.

圖2 不同影響因素對PDB3菌好氧反硝化比速率影響的響應面Fig.2 Response surface diagram of the effect of different influencing factors on the rate of aerobic denitrification of strain PDB3
2.3 模型驗證
為了驗證本模型的有效性和準確性,在預測的最佳條件下進行初始 NO3--N 濃度為138.6mg/L的定量實驗.如圖3所示,通過將OD600值與 Logistic模型擬合和 NO3--N濃度與ExpDec1模型擬合來分別繪制PDB3菌株生長曲線和NO3--N 降解曲線.相關系數(R2)分別為0.995、0.981.表明模型方程均能夠充分描述PDB3菌株生長曲線和NO3--N降解特性.在接種前2h, OD600值增加緩慢,NO3--N濃度變化很小.這主要歸因于 PDB3菌株需要時間來調整其新陳代謝以適應新環境.隨后在 2~10h時間內OD600值迅速增加,細胞比生長速率在2~4h內達到最大,在4~7h內又急劇下降,4h時菌株細胞比生長速率達到最大,μmax為 0.42h-1.對于一些反硝化菌,其最大細胞比生長速率約為 0.013~0.208h-1[22],表明PDB3菌株在最優化條件下比這些反硝化菌生長更快.2~10h內NO3--N濃度迅速下降.表明快速生長的菌株需要大量的氮源來維持自身代謝需要.最終在 12h,測得 PDB3菌好氧反硝化比速率為 2.12h-1,誤差僅為 5.8%,故驗證實驗值與預測值極為接近,可以認為本模型準確有效.

圖3 PDB3菌株生長曲線和降解NO3--N濃度曲線Fig.3 Growth curve of strain PDB3 and degradation curve of NO3--N
2.4 氮平衡分析
在 RSM 分析獲得的優化條件下發酵培養12h,發酵液離心后取上清液測量TN、NH4+-N、NH2OH-N、NO3--N及NO2--N,取沉淀測胞內氮.PDB3菌好氧反硝化發酵過程中各物質氮含量如表4所示.最初NO3--N是氮源的唯一形式,故TN為277.2mg.隨著PDB3菌好氧反硝化及細胞同化作用的進行,氨氮、羥胺、亞硝酸鹽氮及有機氮出現少量積累,王弘宇等[23]在研究兩株異養硝化細菌的脫氮特性時發現有少量亞硝酸鹽積累,而張樹松等[24]在研究 Diaphorobacter polyhydroxybutyrativorans SL-205菌的好氧反硝化特性時發現沒有亞硝酸鹽積累,可見不同菌株的脫氮中間產物并不一樣.胞內氮由初始2.04mg增長到78.3mg,增長量占去除總氮量的29.0%.存在 67.3%的氮損失,這部分氮最終以氮氣形式被去除[25].此外,PDB3菌在好氧脫氮過程中胞內氮和氮損失共計占初始氮總量的96.3%,可見PDB3菌主要是通過好氧反硝化作用和細胞同化作用脫氮,這與其他好氧反硝化菌的研究結果一致[26-27].
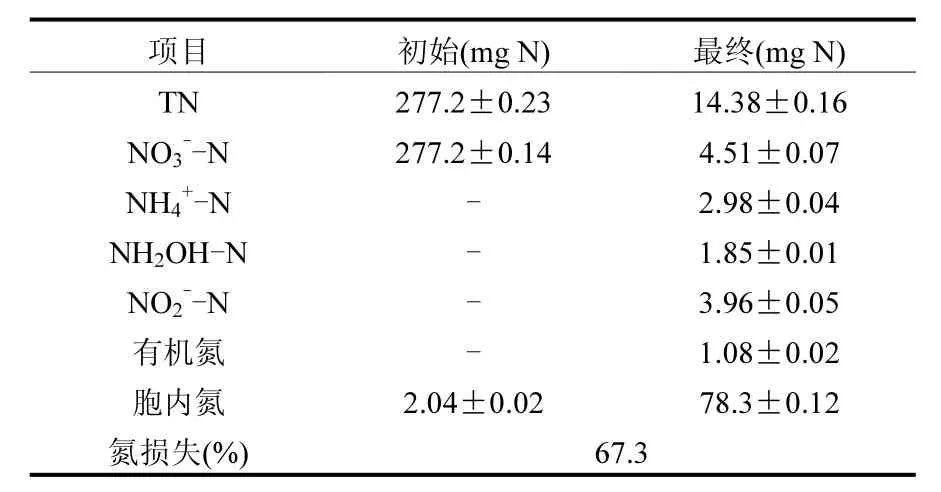
表4 PDB3菌好氧反硝化過程中氮平衡分析Table 4 Analysis of nitrogen balance during aerobic denitrification by strain PDB3
2.5 RT-qPCR分析
在 RSM 分析獲得的優化條件下發酵培養,測定PDB3菌好氧反硝化過程中脫氮基因napA、nirS、cnorB、nosZ的表達水平,以進一步解釋脫氮特征.如圖4所示,圖中4種脫氮基因的表達水平為進行歸一化處理后的相對表達量.napA基因表達迅速,在 1~3h保持較高水平,這可能與初始培養基中含硝酸鹽氮有關.nirS、cnorB、nosZ的表達與napA基因的表達之間存在一個時間差.原因可能是這 3個基因的表達需要亞硝酸鹽[28].7h后亞硝酸鹽耗盡導致了nirS表達的快速降低.3~6h時cnorB的表達達到最高水平.cnorB、nosZ基因表達量比napA、nirS基因表達量約高一個數量級,這就保證胞內有足夠的 NO還原酶和N2O還原酶合成以將NO和N2O濃度維持在較低水平.

圖4 PDB3菌好氧反硝化過程中4種脫氮基因的表達量Fig.4 Expression of four nitrogen removal genes in aerobic denitrification of strain PDB3
3 結論
3.1 得出檸檬酸鈉為PDB3菌好氧反硝化的最佳碳源.
3.2 利用Box-Behnken實驗構建出PDB3菌好氧反硝化的模型方程.模型預測結果表明 PDB3菌發酵罐內反硝化最佳條件:pH值、碳氮比、溫度、轉速分別為 7.3、8.1、30.1℃、299.9r/min;根據方程計算得到最佳好氧反硝化比速率為2.25h-1.驗證實驗表明預測值與實驗值極為接近,故可以認為本模型準確有效.
3.3 氮平衡分析表明,胞內氮增長量占總氮去除量的29.0%.67.3%的氮以含氮氣體的形式被去除.此外,PDB3菌在好氧脫氮過程中胞內氮和氮損失共計占初始總氮量的96.3%,PDB3菌主要是通過好氧反硝化作用和細胞同化作用脫氮.
3.4 研究了PDB3菌好氧反硝化過程中脫氮基因的表達水平.napA 基因表達迅速,并與 nirS、cnorB、nosZ基因的表達之間存在一個時間差.此外cnorB、nosZ基因表達量比napA、nirS基因表達量約高一個數量級,保證了體系中有足夠的NO還原酶和N2O還原酶合成以將NO和N2O的積累保持在低水平.
[1]Walter G, Zumft K. Cell biology and molecular basis of denitrification [J]. Microbiology and Molecular Biology Reviews,1997,61(4):533-542.
[2]辛明秀,趙 穎,周 軍,等.反硝化細菌在污水脫氮中的作用[J]. 微生物學通報, 2007,34(4):773-776.
[3]周石磊,黃廷林,白士遠,等.貧營養好氧反硝化菌的分離鑒定及其脫氮特性 [J]. 中國環境科學, 2016,36(1):238-248.
[4]Yao S, Ni J R. Heterotrophic nitrification and aerobic denitrification at low temperature by a newly isolated bacterium Acinetobacter sp. HA2 [J]. Bioresource Technology, 2013,45(139):80-86.
[5]Chen P, Li J, Li Q X, et al. Simultaneous heterotrophic nitrification and aerobic denitrification by bacterium Rhodococcus sp CPZ24 [J].Bioresource Technology, 2012,32(116):266-270.
[6]周石磊,黃廷林,張春華,等.基于 Miseq的好氧反硝化菌源水脫氮的種群演變 [J]. 中國環境科學, 2016,36(4):1125-1135.
[7]Chen Q, Ni J R. Ammonium removal by Agrobacterium sp. LAD9 capable of heterotrophic nitrification-aerobic denitrification [J].Bioscience and Bioengineering, 2012,20(113):619-623.
[8]葛啟隆,岳秀萍,王國英,等.好氧反硝化苯酚降解菌的分離鑒定及動力學 [J]. 環境工程學報, 2014,8(6):2605-2610.
[9]鄒艷艷,張 宇,李明智,等.一株異養硝化-好氧反硝化細菌的分離鑒定及脫氮活性研究 [J]. 中國環境科學, 2016,36(3):887-893.
[10]葛啟隆,岳秀萍,王國英,等.一株苯酚降解菌的分離鑒定及響應面法優化其固定化 [J]. 中國環境科學, 2014,34(2):518-525.
[11]Muyzer G, De Waal E C, Uitterlinden A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA [J]. Applied and environmental microbiology, 1993,59(12):695-700.
[12]Throback N, Enwall K, Jarvis A, et al. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE [J]. FEMS Microbiology Ecology, 2004,49(25):401-417.
[13]Dandie C E, Miller M N, Burton D L, et al. Nitric oxide reductase-targeted real-time PCR quantification of denitrifier populations in soil [J]. Applied and Environmental Microbiology,2007,54(45):4250-4258.
[14]Kapoor V, Li X, Elk M, et al. Impact of heavy metals on transcriptional and physiologicalactivity of nitrifying bacteria [J].Environmental Science & Technology, 2015,49(22):13454-13462.
[15]Gui M Y, Chen Q, Ni J R. Effect of NaCl on aerobic denitrification by strain Achromobacter sp. GAD-3 [J]. Applied Microbiology and Biotechnology, 2017,50(101):5139-5147.
[16]國家環境保護總局《水和廢水監測分析方法》編委會.水和廢水監測分析方法 [M]. 4版.北京:中國環境科學出版社, 2002:315-318.
[17]Frear D S, Burrell R C. Spectrophotometric method for determining hydroxylamine reductase activity in higher plants [J].Analytical Chemistry, 1955,27(10):1664-1665.
[18]王驍靜,于德爽,李 津,等.海洋異養硝化-好氧反硝化菌 y6同步脫氮除碳特性 [J]. 中國環境科學, 2017,37(2):686-695.
[19]Zhang Q L, Liu Y, Ai G M, et al. The characteristics of a novel heterotrophic nitrification-aerobic denitrification bacterium,Bacillus methylotropHicus strain L7 [J]. Bioresource Technology,2012,108(3):35-44.
[20]Huang X, Li W, Zhang D, et al. Ammonium removal by a novel oligotrophic Acinetobacter sp. Y16capable of heterotrophic nitrification-aerobic denitrification at low temperature [J].Bioresource Technology, 2013,146(10):44-50.
[21]李 鋒,溫順華,黃庶冰,等.響應面法優化甘露醇產丁醇的發酵條件 [J]. 化工進展, 2013,32(7):1629-1634.
[22]Shrimali M, Singh K P. New methods of nitrate removal from water [J]. Environmental pollution, 2001,35(112):351-359.
[23]王弘宇,馬 放,楊 開,等.兩株異養硝化細菌的氨氮去除特性[J]. 中國環境科學, 2009,29(1):47-52.
[24]張 樹 松,樊 月 婷,孫 興 濱,等.菌 株 Diaphorobacter polyhydroxybutyrativorans SL-205的反硝化特性 [J]. 中國環境科學, 2017,37(9):3532-3539.
[25]王國英,崔 杰,岳秀萍,等.異養硝化-好氧反硝化菌脫氮同時降解苯酚特性 [J]. 中國環境科學, 2015,35(9):2644-2649.
[26]Ren Y X, Yang L, Liang X. The characteristics of a novel heterotrophic nitrifying and aerobic denitrifying bacterium,Acinetobacter junii YB [J]. Bioresource Technology, 2014,30(171):1-9.
[27]Shi Z, Zhang Y, Zhou J, et al. Biological removal of nitrate and ammonium under aerobic atmosphere by Paracoccus versutus LYM [J]. Bioresource Technology, 2013,35(148):144-148.
[28]Mao S Z, Da H.Reducing NO and N2O emission during aerobic denitrification by newly isolated Pseudomonas stutzeri PCN-1 [J].Bioresource Technology, 2014,20(162):80-88.

