氧化石墨烯與淀粉樣短肽相互作用的理論研究
謝羅剛
(鄭州輕工業學院 物理與電子工程學院,鄭州 450001)
1 引 言
蛋白質是生物體內功能的執行者,通常在生物大分子環境中進行折疊并行使生物學功能,正確的折疊是其行使生物功能的前提和條件. 但是在適當的條件下,蛋白質會發生錯誤折疊并導致病理性聚集而形成淀粉樣纖維[1]. 與蛋白質聚集相關的疾病大約有20多種,像阿茲海默癥(Alzheimer’s disease,AD)、帕金森氏癥、II型糖尿病等[2-5]. 阿茲海默癥,俗稱老年癡呆癥,是與我們最密切相關的神經性疾病,它的病理性特征是在病人的腦組織里面出現老年斑,這些斑塊主要由Aβ(Amyloid-β)組成[6]. Aβ通常具有39-42個氨基酸的縮氨酸,它的病理性聚集將導致不溶于水的淀粉樣纖維的形成,進而導致疾病的發生. Aβ16-22片段對全長Aβ蛋白形成β-sheet結構起著關鍵作用,因為該片段包括中心疏水核(序列為LVFF)[7]. 同時,該片段也是形成與Aβ纖維類似結構的最短片段[8]. 因此,Aβ16-22是研究Aβ聚集的理想蛋白片段.
阿茲海默癥等淀粉樣變性病與淀粉樣肽的錯誤折疊和聚集有關,為了解決淀粉樣肽的聚集,人們一直致力于找尋抑制蛋白以及淀粉樣肽聚集的抑制劑. 碳納米顆粒容易通過血腦屏障進入人體,因而對淀粉樣肽聚集的調控研究受到人們的關注. 最近的研究表明,碳納米顆粒(富勒烯,碳納米管和石墨烯及其衍生物)能夠影響淀粉樣肽的聚集以及纖維化的形成[9-14]. 氧化石墨烯(graphene oxide,GO)是典型的二維納米材料,是石墨烯的衍生物. 氧化石墨烯的結構遵從C10O1(OH)1(COOH)0.5模式,也就是說,每20個碳原子包含2個環氧基、2個羥基、1個羧基. 其中羥基和環氧基位于GO表面上;羧基主要位于GO的邊緣部分. 氧化石墨烯能夠調控淀粉樣肽的聚集已有諸多報道,像國家納米中心的王琛研究組報道了在GO表面吸附病毒蛋白片段Vpr13-33,該片段結構會發生從α螺旋到β片層的轉變,是誘導聚集的[15]. 但是,另外一些研究工作表明氧化石墨烯是抑制蛋白或淀粉樣肽的聚集. 譬如,Mahmoudi等提出了氧化石墨烯將會對Aβ蛋白的纖維化有強烈的抑制作用[12]. 他們認為GO片層可以吸附淀粉樣肽單體,能夠降低溶液中蛋白濃度,進而抑制蛋白聚集. 另外,Li[16]以及Zhou[17]等研究組也相繼提出了氧化石墨烯以及氧化石墨烯的衍生物能夠強烈抑制Aβ33-42蛋白片段以及全長的hIAPP蛋白的淀粉樣聚集. 從上面的研究結果可以看出,GO能夠調控淀粉樣肽的聚集,但是GO對蛋白質聚集的影響結果不相同.
分子動力學(molecular dynamics,MD)和副本交換分子動力學(replica exchange molecular dynamics,REMD)方法能夠跟蹤系統中各原子的運動軌跡,獲得許多納米尺度上的細節[18-21]. 目前,該方法被廣泛應用于研究淀粉樣肽的聚集以及蛋白質與生物小分子的相互作用[13, 14]. GO調控淀粉樣肽聚集的研究主要集中在實驗工作方面,相關的理論工作還比較少,GO與蛋白質間相互作用的分子機制仍不清楚,需要利用計算機模擬作進一步研究. 在本文中,我們將選擇Aβ16-22為模型片段,利用REMD模擬對比研究Aβ16-22在加入石墨烯以及氧化石墨烯前后的聚集情況,找出氧化石墨烯影響淀粉樣肽聚集的物理機制,為實驗研究提供理論基礎.
2 模型與方法
我們的模擬體系包括:Aβ16-22以及加入氧化石墨烯后的Aβ16-22+GO體系;為了對比研究,我們還考慮加入石墨烯(graphene,G)后的Aβ16-22+G體系. Aβ16-22的氨基酸序列為Ac-KLVFFAE-NH2,兩端被乙酰基和氨基保護. 我們模擬的體系是中性pH值,Lys和Glu兩個氨基酸的側鏈分別帶上正負電荷. 我們選擇8條氨基酸來模擬其聚集,初始結構呈無序狀態. 石墨烯和氧化石墨烯每個體系加入3片,每片碳原子選擇60個(如圖1所示),為了方便與之前的研究富勒烯C60調控淀粉樣肽聚集的結果作對比[14]. 氧化石墨烯還包括3個羧基、6個羥基和6個環氧基,如圖1所示. 每個體系都放在大小(6.8×6.8×6.8 nm3)的立方體的tip3p水模型的盒子里面,蛋白到水盒子壁的最近距離為0.9 nm. 這三個體系(Aβ16-22、Aβ16-22+G和Aβ16-22+GO)的總原子數分別為:29710,29536和29578.

圖1 氧化石墨烯的結構圖. (A)頂視圖,(B)側視圖. 銀色代表C原子,紅色代表O原子,白色代表H原子Fig. 1 The chemical structure of GO molecule: (A) top view and (B) side view, carbon atoms are in silver, oxygen atoms in red, and hydrogen atoms in white.
三個體系均采用REMD[22]進行模擬,時長均為200 ns. 我們采用的是GROMACS-4.5.3程序包[23, 24],amber99sb-ildn力場以及NPT系綜. 每個REMD模擬用48個副本,溫度范圍是從308 K到410 K之間呈指數增長. 溫度耦合采用速度標度耦合方法,耦合時間為0.1 ps. 壓強耦合采用Parrinello-Rahman方法,耦合時間為1.0 ps. 靜電相互作用采用的是particle mesh Ewald方法,截斷半徑為1.0 nm. GO的力場參數平移于amber99sb-ildn力場.
分析方法主要基于GROMACS軟件包里面的程序以及我們自己開發的一些程序. 結果分析時,去掉前面100 ns的數據,因此我們三個體系的數據分析都是基于后100 ns的REMD模擬. 分析的數據是在310 K溫度下的副本. 利用DSSP方法,我們將對3個體系中氨基酸鏈的二級結構進行分析. 利用Daura團簇分析方法,對基于REMD模擬得到的構型進行分類分析,其中Cα-RMSD截斷半徑我們選取0.3 nm. 我們計算了Aβ16-22氨基酸鏈間以及Aβ16-22與GO之間的氫鍵相互作用以及氨基酸鏈間主鏈和主鏈以及側鏈和側鏈的相互接觸幾率,主要考慮兩類原子對:Cα原子對之間要小于0.54 nm,其它原子對要小于0.46 nm. 本文所有的結構圖都是用VMD軟件畫出[25].
3 結果與討論
我們首先比較了100-150 ns和150-200 ns兩個時間段內Aβ16-22、Aβ16-22+G和Aβ16-22+GO三個體系中各氨基酸二級結構(coil和β-sheet)的幾率. 根據我們對二級結構的計算(如圖2所示),發現coil和β-sheet是最主要的二級結構,因此在這里只給出該兩種二級結構的幾率,如表1所示. 通過對比我們發現,在這兩個時間段內除了端部2個氨基酸外,其他各二級結構的幾率變化不大,差別在0.1 % ~ 10.8 %之間. 這些數據表明我們的模擬已基本收斂.
表1 氨基酸在100-150 ns和150-200 ns兩個時間段內二級結構的幾率. 括號內為150-200 ns時段二級結構的幾率
Table 1 The calculated secondary structure probability of each residue in the REMD runs within the time intervals of 100-150 ns and 150-200 ns (in bracket) for Aβ16-22

氨基酸Coil (%)β-sheet (%)Aβ16-22Aβ16-22+GAβ16-22+GOAβ16-22Aβ16-22+GAβ16-22+GOK160(0)0(0)0(0)0(0)0(0)0(0)L1747.90 (42.86)49.50 (46.35)70.13 (69.33)44.83 (40.77)36.43 (37.54)21.84 (21.61)V1827.48 (23.52)30.21 (28.37)48.85 (52.56)60.14 (65.51)50.33 (51.42)26.23 (24.71)F1921.70 (20.27)22.41 (24.58)45.81 (43.97)63.82 (67.30)54.21 (55.49)29.68 (29.67)F2031.90 (32.96)33.23 (32.95)55.71 (56.03)53.13 (55.35)47.55 (45.34)22.25 (25.22)A2161.46 (61.33)62.73 (61.89)80.91 (80.20)30.53 (30.57)30.21 (30.78)12.03 (14.05)E220(0)0(0)0(0)0(0)0(0)0(0)
3.1 GO對Aβ16-22八聚體二級結構的影響
圖2給出了Aβ16-22、Aβ16-22+G和Aβ16-22+GO三個體系中Aβ16-22八聚體二級結構的幾率,該幾率是對所有氨基酸做了平均. 從圖中我們可以看出,coil和β-sheet是兩種最主要的二級結構,β-bridge、bend、turn和helix的幾率都比較低. 純的Aβ16-22蛋白片段,初始結構是無定型的、無規卷曲的八聚體,可以形成富含β-sheet的聚集體結構,與我們之前的結果一致[14],我們得到β-sheet的平均幾率為51.19 %. 當我們在體系中添加石墨烯時,二級結構幾率略有下降,β-sheet的幾率由51.19 %降到43.59 %,下降了14.85 %. 當在體系中添加氧化石墨烯時,β-sheet的幾率出現明顯的降低,從51.19 %降到22.73 %,降低了將近56%;而coil結構的幾率則由37.14 %增加到60.35 %. 值得注意的是,我們之前研究了C60對Aβ16-22聚集的影響,發現β-sheet的幾率由45.20 %降到25.70 %,降低了50 %[14]. 相同的碳原子數,氧化石墨烯對Aβ16-22聚集的抑制作用略強于C60. 我們的研究結果表明,氧化石墨烯的加入對Aβ16-22中β-sheet的形成有著明顯的抑制作用. 另外,β-bridge、bend、turn和helix的幾率在加入石墨烯或者氧化石墨烯前后變化并不明顯.

圖2 Aβ16-22、Aβ16-22+G和Aβ16-22+GO三個體系中Aβ16-22八聚體二級結構幾率分布圖.Fig. 2 Calculated secondary structure probabilities of Aβ16-22 octamers in the absence and presence of graphene and graphene oxide nanoparticles.
我們進一步計算了Aβ16-22、Aβ16-22+G和Aβ16-22+GO三個體系中各個氨基酸的β-sheet幾率,如圖3所示. 我們發現,在Aβ16-22體系中,中心疏水區域L17-V18-F19-F20-A21各氨基酸的β-sheet幾率為30.55 ~ 65.56 %,其中以V18和F19的幾率最高,分別為62.82 %和65.56 %. 在Aβ16-22+G體系中,CHC區域各個氨基酸的β-sheet幾率為30.72 ~ 54.42 %,L17-F20的幾率略有下降,A21的幾率不變. 當體系中加入GO后,CHC區域氨基酸的β-sheet幾率下降為13.04 ~ 29.68 %,下降非常顯著,降低了將近50 %. 這些結果進一步表明GO能夠顯著降低Aβ16-22中β-sheet形成的幾率,這對淀粉樣肽的聚集是不利的.
3.2 GO對Aβ16-22八聚體三級結構的影響
為了研究GO對Aβ16-22形成的聚集體結構影響,我們對50,000個構象做了基于RMSD的結構類聚法分析,其中,Cα-RMSD截斷半徑取0.3 nm. 對這三個體系進行分析,我們得到Aβ16-22、Aβ16-22+G和Aβ16-22+GO體系分別有201、227和272個類結構. 圖4分別給出了三個體系前六個幾率最高的團簇的中心結構以及他們的幾率. 這六個團簇分別占據了這三個體系所有構型的32.5 %,46.5 %和34.3 %. 從圖中我們可以看到,在純的Aβ16-22體系中,團簇主要呈現各種有序以及無序的富含β-sheet的結構.

圖3 Aβ16-22、Aβ16-22+G和Aβ16-22+GO三個體系中各個氨基酸的β-sheet幾率分布圖.Fig. 3 Calculated β-sheet probability of each residue of Aβ16-22 octamers in the absence and presence of graphene and graphene oxide nanoparticles.

圖4 Aβ16-22八聚體在加入石墨烯和氧化石墨烯前后平衡態下的構型,取前6個幾率最高的團簇構型并給出相應的幾率.Fig. 4 Representative conformations of the top six most-populated clusters for the Aβ16-22 octamers in the absence and presence of graphene and graphene oxide nanoparticles.
有趣的是,當體系中加入石墨烯后,石墨烯初始是無序地分布在水盒子里面,當進行200 ns的副本交換分子動力學模擬后,3片石墨烯則面對面對齊排列在一起,周圍包裹著Aβ16-22聚集體,如圖4中間所示. 我們知道,純的石墨烯是高疏水性分子,能夠與蛋白片段Aβ16-22中的疏水氨基酸(L17-A21)發生疏水相互作用. 但是需要注意的是,高疏水的石墨烯片層結構在水溶液中自身容易發生團聚,這樣就會減少與Aβ16-22蛋白片段氨基酸的接觸面積,進而會弱化與Aβ16-22蛋白的相互作用. 因此,石墨烯對Aβ16-22蛋白聚集的影響很有限,與我們前面對二級結構的分析結果一致. 當體系中加入GO后,我們發現Aβ16-22主要以無定型狀態存在,從圖4下可以看出聚集體主要包含無序的氨基酸鏈以及一些二聚體和三聚體. 這些結果表明,GO能夠使Aβ16-22八聚體從富含β-sheet的結構向無序狀聚集體結構轉變,從而影響淀粉樣肽的聚集.
3.3 GO與Aβ16-22蛋白片段間的氫鍵相互作用
通過對二級結構以及三級結構分析,我們發現石墨烯對淀粉樣肽Aβ16-22片段聚集的影響很有限. 因此,在這里我們將主要研究Aβ16-22+GO體系中GO與Aβ16-22-氨基酸鏈間的相互作用. 相比石墨烯,氧化石墨烯表面有3類極性基團,包括羥基,羧基和環氧基,很容易與Aβ16-22蛋白片段的主鏈以及部分側鏈形成氫鍵. 圖5給出了Aβ16-22和Aβ16-22+GO體系中,氨基酸鏈間(Aβ-Aβ)以及氨基酸鏈與GO之間(Aβ-GO)的氫鍵數目(包括主鏈和側鏈)的幾率密度分布. 我們發現和Aβ16-22體系相比,氨基酸鏈間Aβ-Aβ的氫鍵數目在Aβ16-22+GO體系中有著明顯的降低,從27降到14. 其中很重要的原因就是Aβ16-22氨基酸鏈與GO形成多達20個氫鍵. Aβ-GO之間的氫鍵作用很明顯將與氨基酸鏈間的氫鍵相互作用產生競爭,進而破壞Aβ16-22聚集體的氫鍵網絡,最終達到抑制淀粉樣肽Aβ16-22聚集的目的.
我們進一步計算了Aβ16-22和Aβ16-22+GO體系中不同氨基酸鏈間主鏈-主鏈以及側鏈-側鏈間的接觸幾率,如圖6所示. 我們發現兩個體系呈現不同的鏈間作用模式,表明氧化石墨烯對鏈間相互作用有重要的影響. 圖6(A和C)給出了Aβ16-22體系中八聚體各氨基酸主鏈和側鏈的接觸幾率圖. 主鏈間主要相互作用對包括:V18-V18、V18-F19和F19-F19,接觸幾率分別是8.92 %、8.94 %和7.83 %. 側鏈間的主要相互作用對包括:L17-F19、F19-F19和F19-F20,接觸幾率分別是11.19 %、11.68 %和10.64 %. 圖6(A)中,接觸幾率比較高的地方是沿左對角線方向,該結果表明氨基酸鏈間排列將主要以反平行堆積為主,該結果與之前實驗報道的Aβ16-22氨基酸鏈形成反平行對齊的堆積模式的結論一致[8]. 當體系中加入GO納米顆粒時,我們發現盡管氨基酸鏈間仍然采用反平行的排列模式,但是氨基酸間的接觸幾率明顯降低,如圖6(B)和(D)所示. 穩定主鏈間相互作用的氨基酸對的接觸幾率不高于4.75 %,穩定側鏈間相互作用的氨基酸對的接觸幾率不超過6.5 %,與Aβ16-22體系相比,接觸幾率下降了50 %. 我們的計算結果表明,氧化石墨烯顆粒極大地弱化了氨基酸間的相互作用,特別是對β-sheet的形成以及Aβ16-22的纖維化形成有重要作用的氨基酸對,進而抑制Aβ16-22淀粉樣肽的聚集.
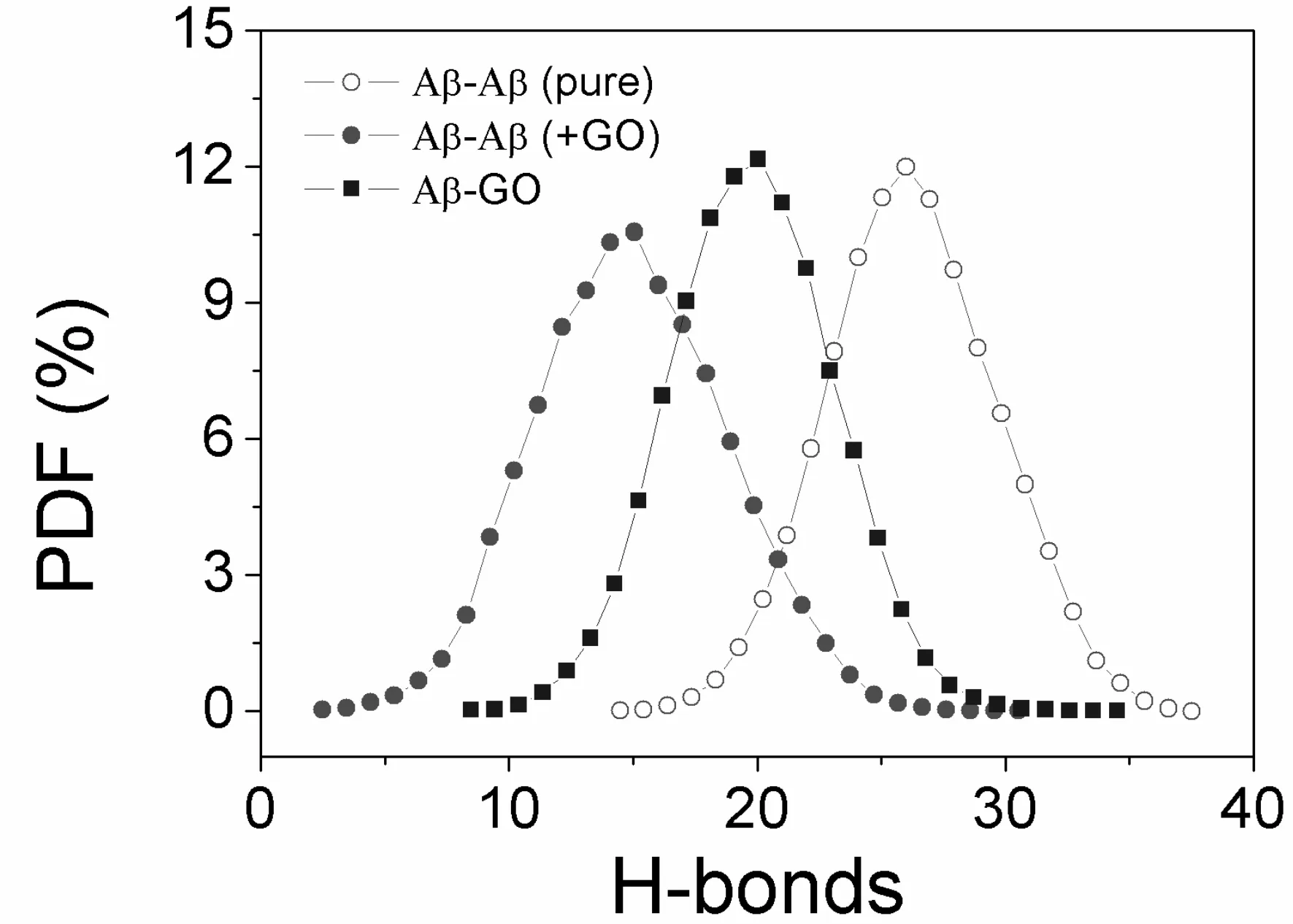
圖5 加入GO前后,氨基酸鏈間(Aβ-Aβ)以及氨基酸鏈與GO間(Aβ-GO)氫鍵數目的幾率密度分布.Fig. 5 Probability distribution functions (PDF) of the number of H-bonds for Aβ16-22 octamers (Aβ-Aβ) and between Aβ16-22 and GO (Aβ-GO) in the absence and presence of GO nanoparticles.

圖6 Aβ16-22(A和 C)和Aβ16-22+GO(B和 D)體系中各氨基酸主鏈以及側鏈間接觸幾率圖.Fig. 6 Main chain - main chain and side chain - side chain contact probability maps for Aβ16-22 octamers in the absence (A and C) and presence (B and D) of GO nanoparticles.
4 結 論
利用副本交換分子動力學模擬方法,我們研究了Aβ16-22八聚體在加入石墨烯和氧化石墨烯前后的聚集情況. 我們的研究結果包括如下:(1)純的Aβ16-22八聚體會形成富含β-sheet的聚集體結構,與我們之前的模擬結果一致;(2)石墨烯對淀粉樣肽Aβ16-22片段聚集的影響很有限,主要原因是石墨烯在水溶液中容易發生團聚,這將大大減少與蛋白片段中氨基酸的接觸面積,弱化了對淀粉樣肽聚集的影響;(3)氧化石墨烯對Aβ16-22片段的聚集具有很強的抑制作用,主要原因是氧化石墨烯與Aβ16-22蛋白片段中的氨基酸間的疏水相互作用以及氧化石墨烯表面的極性基團與Aβ16-22形成的氫鍵作用將大大地弱化了Aβ16-22氨基酸鏈間的相互作用,最終抑制Aβ16-22的纖維化. 我們的研究結果將為找尋抑制淀粉樣肽聚集的抑制劑提供理論幫助.

