基于lncRNA-mRNA共表達網絡的HBV相關肝癌生物靶點篩選及綜合分析
農順強 陳曉昊 許桂丹 韋武均 彭彬 周律 鄧益斌
【摘要】?目的?對公共基因表達數據庫(GEO)中的HBV相關肝癌數據進行生物信息學分析,探討長鏈非編碼RNA(lncRNA)與mRNA在HBV相關肝癌中的作用。方法?從GEO數據庫下載HBV相關肝癌數據集,利用相應R包對數據集的差異表達基因進行計算與比較,獲取差異基因并構建lncRNA-mRNA共表達網絡、蛋白質相互作用(PPI)網絡和模塊分析;繼而對共表達網絡中的mRNA進行基因本體論(GO)功能注釋和京都基因與基因組百科全書(KEGG)路徑分析;并分析與關鍵lncRNA高度相關的mRNA與患者生存曲線的相關性。結果?通過基因分析獲得HBV相關肝癌高度相關的差異lncRNA 30個,mRNA 676個。共表達的mRNA主要參與的GO通路有細胞黏附、細胞運動的負調控、生物黏附、酶聯受體蛋白信號通路、細胞趨化性;KEGG通路主要有類固醇激素生物合成、視黃醇代謝、PI3K-Akt信號通路、ECM-受體相互作用、化學致癌作用、黏著斑、Rap1信號通路等。與關鍵lncRNA(DNM3OS、HAND2-AS1、HELLPAR、AC126118.1、FAM230E、LINC01139、LINC01943、AL022344.1和PCAT7)高度相關的mRNA(GGT5、CEP55、DPT、TEK、TRIP13、STAB2、ESM1和MS4A1)與患者生存曲線有顯著相關性(P<0.01)。結論?lncRNA (DNM3OS、HAND2-AS1、HELLPAR、AC126118.1、FAM230E、LINC01139、LINC01943、AL022344.1和PCAT7)對HBV相關肝癌的發生發展及預后具有重要作用,為HBV相關HCC發生發展的機制研究、預后指標、藥物治療靶點的選擇等提供參考。
【關鍵詞】?乙肝病毒;肝細胞癌;lncRNA-mRNA共表達網絡;生物標志物
中圖分類號:R735.7?文獻標志碼:A?DOI:10.3969/j.issn.1003-1383.2021.01.004
【Abstract】?Objective?To perform bioinformatics analysis on HBV related liver cancer data in public gene expression omnibus (GEO), so as to investigate the role of long non-coding RNA (lncRNA) and mRNA in HBV related hepatocellular carcinoma(HCC).Methods?Data set of HBV related HCC were downloaded from GEO, and differentially expressed genes in the data set were calculated and compared by corresponding R packets to obtain differentially expressed genes and construct lncRNA-mRNA co-expression network, protein-protein interaction (PPI) network and module analysis. And then, gene ontology (GO) functional annotation and Kyoto encyclopedia of genes and genomes (KEGG) pathway analysis were performed on mRNA in co-expression networks, and correlation between mRNA highly related to key lncRNA and patient survival curve was analyzed.Results?By genetic analysis, 30 highly correlated differential lncRNAs and 676 mRNAs were obtained for HBV related HCC. Main GO pathways involved in co-expressed mRNA were cell adhesion, negative regulation of cell movement, bioadhesion, enzyme-linked receptor protein signaling pathway and chemotaxis. KEGG pathways included steroid hormone biosynthesis, retinol metabolism, PI3K-Akt signaling pathway, ECM-receptor interaction, chemical carcinogenesis, focal adhesion and Rap1 signaling pathway, etc. mRNAs (GGT5, CEP55, DPT, TEK, TRIP13, STAB2, ESM1 and MS4A1) that highly co-expressed with the key lncRNAs (DNM3OS, HAND2-AS1, HELLPAR, AC126118.1, FAM230E, LINC01139, LINC01943, AL022344.1 and PCAT7) were significantly correlated with the survival curve of HBV related HCC patient(P < 0.01).Conclusion?lncRNA (DNM3OS, HAND2-AS1, HELLPAR, AC126118.1, FAM230E, LINC01139, LINC01943, AL022344.1 and PCAT7) plays a crucial role in the occurrence, development and prognosis of HBV-related HCC, and provide reference for the study of mechanism, prognostic index, and selection of drug therapeutic targets, etc.
【Key words】?HBV; HCC; lncRNA-mRNA co-expression network; biomarker
肝細胞癌(hepatocellular carcinoma,HCC)是常見的惡性腫瘤之一,且惡性程度較高,其發病率居全球第六位、病死率居第四位[1]。慢性乙型肝炎病毒(HBV)感染是大多數高危HCC地區的主要危險因素。HBV參與肝細胞的癌變、侵襲和轉移,在肝癌的發生發展中起著至關重要的作用[2~4]。由于缺乏特征性臨床表現,HCC的大部分診斷多處于晚期,預后較差。盡管部分文獻報道了一些用于早期診斷的血清生物標志物,但結果并不十分令人滿意[5~7]。因此,探求新的早期發現與早期干預標志物,對于改善肝癌患者預后和提高其長期生存率尤為重要。
長鏈非編碼RNA(lncRNA)一般是指長度大于200個核苷酸(nucleotide,nt),缺乏或者僅有微弱蛋白編碼能力的RNA[8]。lncRNA以多種方式調節表觀遺傳、轉錄及轉錄后、翻譯和翻譯后水平的基因表達[9~10],積極參與腫瘤生長、轉移和復發等多方面的調節[11~12]。lncRNA已經被證實與肝癌細胞的增殖、凋亡、侵襲和轉移、血管生成及預后密切相關[13~14],參與肝癌進展過程中的各種生物學過程。例如,肝癌中表達上調的ZEB1-AS1通過調節上皮-間質轉化誘導標志物的表達水平,參與HCC中的腫瘤生長和轉移,且高表達ZEB1-AS1的患者與高轉移復發和預后不良相關[15]。HULC在HCC中上調,與HBV感染及HCC患者腫瘤生長相關,并與其腫瘤分級、轉移和耐藥性相關[11]。雖然與HCC的lncRNA相關研究在逐日增多,但lncRNA在HBV相關肝癌發生發展進程中的作用及機制研究仍舊十分局限。
芯片技術、二代測序和其他基因檢測技術及生物信息學的發展,為疾病的診斷和治療靶點的發掘提供了新的技術手段[16]。本研究利用生物信息學方法,從GEO數據庫中下載獲得HBV相關肝癌基因數據,構建lncRNA-mRNA共表達網絡,獲取關鍵lncRNA,通過基因本體論(gene ontology,GO)和京都基因與基因組百科全書(kyoto encyclopedia of genes and genomes,KEGG)分析HBV相關肝癌共表達mRNA所富集的信號通路,分析與關鍵lncRNA共表達的mRNA及HBV相關肝癌患者生存時間的相關性,探究關鍵lncRNA在肝癌發生、發展進程中的作用機制,從而為lncRNA在作為HBV相關肝癌的診斷、預后和治療靶點的潛在生物標志物提供新的見解及有價值的信息。
1?資料與方法
1.1?數據材料
從美國國立生物技術信息中心(national center for biotechnology information,NCBI)的GEO數據庫中,檢索HBV相關肝癌芯片表達數據集。選取GSE55092、GSE19665芯片表達數據集行后續分析。所選數據集均基于Affmetrix公司[HG-U133_Plus_2]平臺,其中GSE55092包括49例肝癌和91例癌旁正常樣本,GSE19665包括肝癌和癌旁正常樣本各5例。以GSE14520的218例HBV相關肝癌數據做共表達mRNA的生存分析。
1.2?篩選差異表達基因
首先,我們從Affymetrix官方網站(www.affymetrix.com)下載芯片探針序列FASTA格式文件。然后,使用SeqMap工具將HG-U133_Plus_2芯片的探針序列與GENCODE的人類基因組(GRCh38)(https://www.gencodegenes.org/)(release 30)和lncRNA基因序列進行比對,獲取非編碼RNA(non-coding RNA)和信使RNA(mRNA)探針信息。使用R語言中的limma[17]包標準化數據,將HBV肝癌組與HBV正常組比較,以差異倍數2倍(|logFC|>1),P<0.05為標準,獲取差異基因;使用R語言中Robust Rank Aggreg包[18],根據log Fold Change值對基因進行排序,并選出兩個數據集中都存在差異表達的基因。
1.3?基因共表達分析
選取在2個數據集中,HBV肝癌和HBV正常患者中均差異表達的lncRNA和mRNA,納入共表達分析網絡。根據基因表達值,計算所有篩選出的差異表達lncRNA與mRNA兩兩間的皮爾森相關系數,選取相關系數絕對值|r|>0.75,且校正后P<0.05的lncRNA和mRNA對,導入Cytoscape 3.72構建共表達網絡。
1.4?差異表達基因功能和通路富集分析
利用DAVID(https://david.ncifcrf.gov/)對共表達的差異基因進行GO富集分析及KEGG分析,以P<0.05為富集標準。
1.5?蛋白質相互作用網絡構建
將共表達的mRNA,用在線數據String(https://string-db.org/)進行蛋白質互作(PPI)分析及MCODE模塊識別,將結果網絡引入Cytoscape進行可視化,選取基因模塊,利用DAVID網站進行信號通路分析。
1.6?生存分析
使用R語言的“Survival”包行Kaplan-Meier生存分析,分析與lncRNA存在共表達關系的差異mRNA和HBV肝癌患者生存時間相關性,檢驗方法為Log-rankχ2,以α=0.05為檢驗水準。
2?結?果
2.1?差異基因篩選
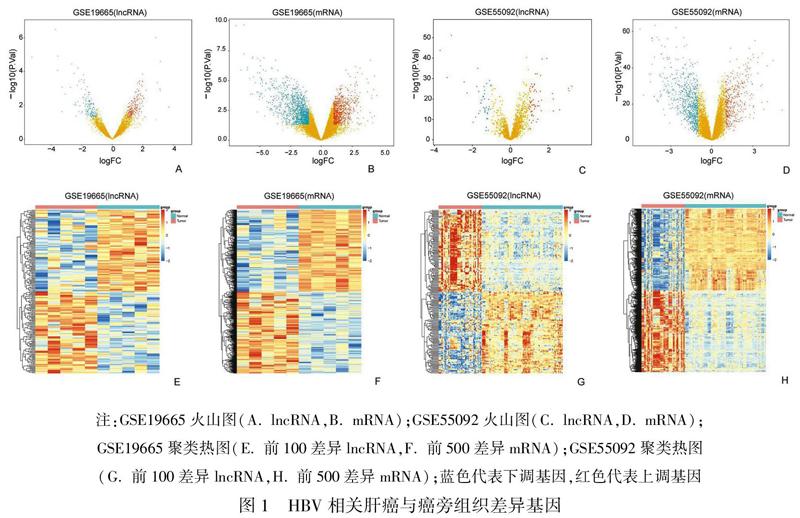
根據篩選條件,在數據集GSE55092中得到差異mRNA 1182個,上調基因412個,下調基因770個;差異lncRNA 103個,上調基因59個,下調基因44個。在數據集GSE19665中得到差異mRNA 2147個,上調基因833個,下調基因1314個;差異lncRNA 333個,上調基因195個,下調基因138個(圖1)。根據log Fold Change值,對2個數據集差異基因進行排序,然后對2個數據集進行Robust Rank Aggreg分析,得到HBV肝癌相關可信度高的差異lncRNA 30個、mRNA 676個。
2.2?LncRNA-mRNA共表達網絡分析
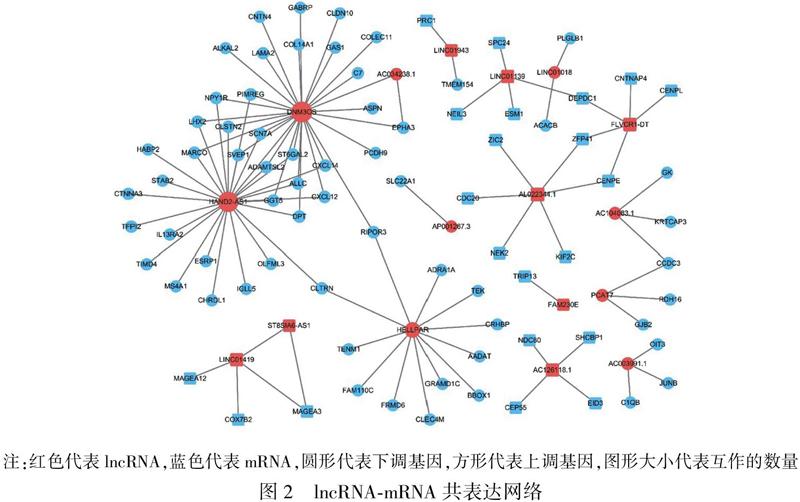
通過共表達網絡構建(共表達相關系數絕對值|r|>0.75,且校正后P<0.05),發現有17個lncRNA及82個mRNA具有高度共表達關系。其中DNM3OS、HAND2-AS1、HELLPAR居于共表達網絡核心位置,與眾多mRNA均具有共表達關系(圖2)。
2.3?差異基因功能富集及通路富集分析
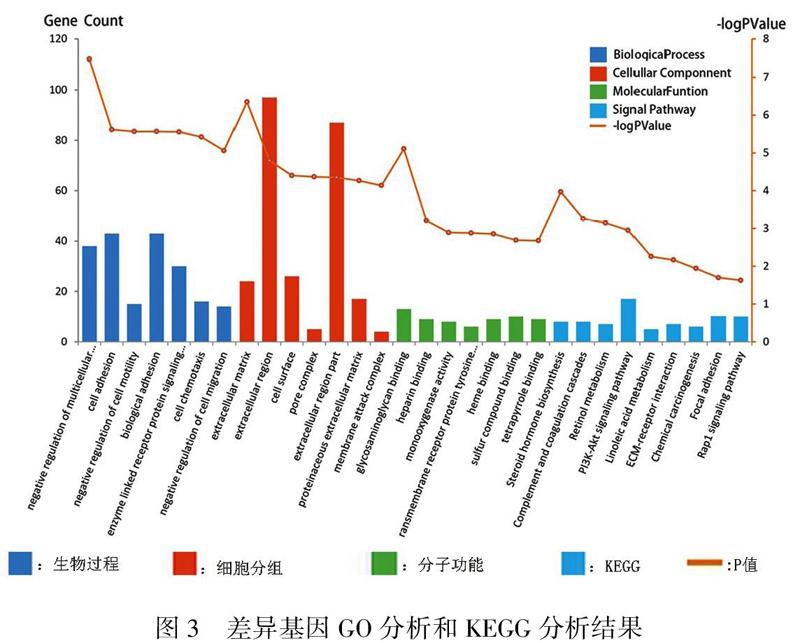
對共表達的mRNA,通過DAVID進行GO分析發現,差異基因主要參與多細胞生物過程的負向調控、細胞黏附、細胞運動的負調控、生物黏附、酶聯受體蛋白信號通路、細胞趨化性、單加氧酶活性跨膜受體蛋白酪氨酸激酶活性等。KEGG分析發現,共表達差異基因主要參與類固醇激素生物合成、補體和凝血級聯、視黃醇代謝信號通路、ECM-受體相互作用、化學致癌作用、黏著斑、Rap1信號通路等(圖3)。
2.4?共表達差異mRNA的蛋白質互作網絡分析
將PPI網絡引入Cytoscape進行可視化,使用MCODE插件進行模塊識別,模塊1包含28個基因及357連線,模塊2包含10個基因及29連線(圖4)。對模塊進行KEGG通路富集,結果顯示模塊1主要與卵母細胞減數分裂、細胞周期相關;模塊2主要與視黃醇代謝、化學致癌作用、藥物代謝-細胞色素P450、代謝途徑、類固醇激素生物合成、細胞色素P450對異生素的代謝、亞油酸代謝、藥物代謝-其他酶等信號通路相關(表1)。
2.5?與關鍵lncRNA共表達的mRNA與HCC患者生存曲線相關性
為了進一步探討上述關鍵lncRNA在HBV相關肝癌發生、發展中的作用,使用GSE14520數據分析與關鍵lncRNA存在高相關性的mRNA與HBV相關肝癌患者生存預后關系。K-M曲線結果顯示,與9個關鍵lncRNA(DNM3OS、HAND2-AS1、HELLPAR、AC126118.1、FAM230E、LINC01139、LINC01943、AL022344.1和PCAT7)共表達的16個mRNA與HBV相關肝癌患者存在顯著相關性(P<0.05),其中8個mRNA存在高度相關性(P<0.01)。見表2、圖5。
3?討?論
肝癌是一種發病率及病死率高的、世界范圍的、常見的消化系統腫瘤。雖然有多種治療方法,但其臨床預后仍很差;鑒于早期診斷對生存的重要性,急需更準確和更特異的診斷及預后標志物。越來越多的研究報道lncRNA在HBV相關肝細胞癌發生中起著至關重要的作用[19],然而,關于lncRNAs作為預測性生物標志物和治療靶點的研究仍然非常有限。
LncRNA通過表觀遺傳修飾、轉錄調控等多種方式影響其靶基因mRNA的表達[12]。LncRNA-mRNA共表達分析是鑒定lncRNA潛在靶基因和進一步研究lncRNA在疾病中生物學功能的常用方法。本文運用生物信息學技術,對NCBI-GEO數據庫中HBV相關肝癌的數據進行綜合分析,篩選出HBV相關肝癌可信度高的差異lncRNA及mRNA表達譜,并對差異表達基因行lncRNA-mRNA共表達分析。GO功能富集分析發現共表達網絡中的mRNA主要影響多細胞生物過程的負向調控、細胞黏附、細胞運動的負調控、生物黏附、細胞周期進程、酶聯受體蛋白信號通路、細胞趨化性、單加氧酶活性、跨膜受體蛋白酪氨酸激酶活性等。以上結果提示,共表達差異mRNA在HBV相關肝癌的細胞增殖和轉移中起重要作用。KEGG分析發現,共表達差異基因主要參與類固醇激素生物合成、補體和凝血級聯、視黃醇代謝、PI3K-Akt信號通路、ECM-受體相互作用、化學致癌作用、黏著斑、Rap1信號通路等。以上顯著富集的與代謝相關的信號通路,包括類固醇激素生物合成、視黃醇代謝、化學致癌作用等信號通路主要為涉及細胞色素P450酶系統(CYP)的蛋白編碼RNA;CYP在藥物代謝中起著重要作用,參與內源性和外源性物質的代謝,包括藥物和環境化合物[20],其通過化學致癌物的生物活性催化、癌癥治療藥物的激活、作為癌癥治療的靶點和代謝酶等多種機制參與多種癌癥[21]。黏著斑是一種膜相關的高分子集合體,將肌動蛋白、細胞骨架和整合素與細胞外基質連接起來,其在維持細胞張力和細胞生存的信號轉導中起著重要作用,參與調節腫瘤細胞的EMT過程,調控腫瘤的侵襲和轉移[22~23]。PI3K-Akt通路調節腫瘤細胞的增殖和存活,其活性異常不僅能導致細胞惡性轉化,而且與腫瘤細胞的遷移、黏附、腫瘤血管生成以及細胞外基質的降解等相關[24~25]。有研究表明,HBV感染通過促進NF-κB與IAP家族成員細胞凋亡蛋白2(cIAP2)啟動子的結合激活PI3K/AKT/NF-κB信號通路,在肝臟中誘導cIAP2表達而致肝癌[26]。細胞外基質(ECM)由基底膜細胞間質組成,可影響細胞分化、增殖、黏附、形態發生和表型表達等,在腫瘤的轉移中起重要的組織屏障作用。腫瘤細胞通過與ECM中組分黏附后激活或分泌降解酶來消耗基質,形成局部溶解的腫瘤細胞轉移通道。膠原在ECM影響腫瘤細胞生物學行為的功能密切相關[27],膠原降解是腫瘤細胞侵襲和轉移的關鍵環節之一,ECM中膠原蛋白及其他蛋白主要被基質金屬蛋白酶(MMPs)降解,如肝細胞癌組織MMP-2高于癌旁組織,可用于判斷肝癌的侵襲轉移。Ras蛋白是原癌基因ras的表達產物,Ras相關蛋白1(Rap1)為一種小的GTPase,通過調節整合素和其他黏附分子在不同細胞類型中的功能,在細胞-細胞和細胞-基質相互作用中起主導作用,它控制著細胞增殖、黏附、細胞間連接形成和細胞極性等多種過程;Rap1信號通路的異常激活,可促進肝癌細胞的增殖和侵襲轉移[28]。這進一步說明本研究中發現的lncRNA 及其共表達差異mRNA在乙肝相關肝癌的發生、發展中起重要作用,研究這些通路將有助于闡明HBV相關肝癌增殖和侵襲的機制,并有助于預測腫瘤的進展和病人的預后。
本研究中發現的關鍵lncRNA在肝癌或其他腫瘤中有不同程度的報道。如DNM3OS在口腔癌、胃癌及其細胞系中表達上調,與細胞活力及侵襲轉移相關[29~30],本研究發現DNM3OS在HBV相關肝癌中表達下調,其機理還有待進一步研究。HAND2-AS1在肝癌組織及細胞系中均呈低表達,其過表達降低了HCC細胞的活力和增殖[31]。有研究發現,HELLP綜合征相關長鏈非編碼RNA(HELLPAR/LINC-HELLP)可激活大量與細胞周期有關的基因,通過改變增殖速率或退出細胞周期而對滋養層細胞分化產生負面影響,減少增殖和侵襲,減少孕早期絨毛外滋養層細胞從增生型到浸潤型的分化,且阻斷HELLP突變位點可降低絨毛外滋養層細胞的侵襲能力[32~33];與本研究中預測HBV相關肝癌中表達下調的HELLPAR可能通過細胞周期的調控影響HCC進程一致。LINC01139在HCC組織和細胞系中過表達,并且與晚期TNM分期、淋巴結轉移和HCC患者臨床預后不良有關,敲低LINC01139可抑制HCC細胞的增殖、轉移;相關機制研究顯示,LINC01139可能充當miR-30的海綿來上調MYBL2表達而表現出HCC的致癌作用[34]。PCAT7在前列腺癌、非小細胞肺癌和鼻咽癌等惡性腫瘤中高表達,與腫瘤的侵襲轉移相關[35~37],本研究發現PCAT7在HBV相關肝癌中表達下調,其機理還有待進一步研究。
本研究發現另4個lncRNA可能在HBV相關肝癌中有重要作用,并預測其功能。反義lncRNA可通過多種機制在轉錄水平或轉錄后水平調控相應正義的mRNA,從而發揮生物學功能。AC126118.1是LRRFIP2的反義鏈lncRNA,LRRFIP2基因編碼的蛋白質與MYD88一起結合胞質尾部toll樣受體4(TLR4),其導致NF-κB信號傳導的激活;NF-κB在細胞增殖和凋亡的相關基因調控中起關鍵作用,與腫瘤的發生發展密切相關[38]。FAM230E是基因間長鏈非編碼RNA基因家族成員,其序列相似性為230,該家族成員基因通過相似基因序列復制形成,特別是在人類染色體低拷貝重復序列(LCR)或節段性復制中,其功能尚未見相關報道[39~40]。另外本研究發現的長鏈非編碼RNA LINC01943和AL022344.1也未見有相關文獻的報道,其在HBV相關肝癌中的功能及機制有待進一步研究。
生存分析發現,共表達網絡中16個mRNA與HBV相關肝癌患者的生存率顯著相關,其中8個mRNA具有高度相關性。如細胞分裂蛋白調節因子1(PRC1)編碼微管相關蛋白,在有絲分裂和細胞周期調控中起重要作用,敲低PRC1與微管相關因子可抑制胞質分裂,并顯著減輕HCC的發展和肝損傷,高表達PRC1增加了HCC細胞化療耐藥性,并與不良預后相關[41~42]。CEP55在細胞質分裂中有重要作用,其可通過調節JAK2/STAT3/MMPs信號傳導來促進HCC細胞遷移和侵襲[43]。DPT編碼一種細胞外基質蛋白,該蛋白在細胞-基質相互作用和基質組裝中發揮作用;DPT在肝癌中低表達且其表達水平與癌癥轉移和患者預后密切相關,其過表達顯著抑制了體外HCC細胞遷移和體內肝內轉移[44];DPT可能通過與TGF-β1相互作用或其他的潛在機制影響HCC的發生和進展[45]。TEK編碼受體蛋白屬蛋白酪氨酸激酶Tie2家族,為內皮細胞特異性受體,在調節血管生成和重塑中起重要作用;有研究顯示,TEK低表達的透明細胞腎細胞癌患者預后較差并具有良好的診斷區分性,有望成為新的腫瘤標志物[46],這與我們在HBV相關HCC中的發現基本一致。STAB2基因編碼跨膜受體蛋白,其可作用于血管生成、細胞黏附或受體清除等生物過程;在腫瘤細胞和淋巴管內皮細胞共培養的mRNA譜分析中,透明質酸受體STAB2、血管生成因子apelin受體(APLNR)和糖基化酶MAN1A1呈低表達,其參與腫瘤進程機制可能為促進腫瘤的淋巴轉移[47]。研究表明,內皮細胞特異性分子-1(ESM1)在肝癌中過表達,可作為預測HCC生長和死亡的獨立危險因素[48]。以上結果進一步證實了本研究發現的lncRNA在HBV相關肝癌的發生、發展中有著重要作用。
生物信息學作為一門新興學科,目前已成為尋找癌癥診斷及治療靶點的一種分析方法。本研究通過生物信息學方法,綜合分析了lncRNA及其共表達mRNA在HBV相關肝癌發生發展中的作用,為HBV相關HCC發生發展的機制研究、預后指標、藥物治療靶點的選擇等提供參考。
參?考?文?獻
[1]?BRAY F,FERLAY J,SOERJOMATARAM I,et al.Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA:A Cancer J Clin,2018,68(6):394-424.
[2]?LIU Y C,LU L F,LI C J,et al.Hepatitis B virus X protein induces RHAMM-dependent motility in hepatocellular carcinoma cells via PI3K-Akt-oct-1 signaling[J].Mol Cancer Res,2020,18(3):375-389.
[3]?ZHENG S,WU H,WANG F,et al.The oncoprotein HBXIP facilitates metastasis of hepatocellular carcinoma cells by activation of MMP15 expression[J].Cancer Manag Res,2019,11:4529-4540.
[4]?ZHA Y, YAO Q, LIU J S, et al. Hepatitis B virus X protein promotes epithelial-mesenchymal transition and metastasis in hepatocellular carcinoma cell line HCCLM3 by targeting HMGA2[J]. Oncol Lett, 2018,16(5):5709-5714.
[5]?ALI O M, EL AMIN H A, SHARKAWY Y L, et al. Golgi Protein 73 versus Alpha-Fetoprotein as a New Biomarker in Early Diagnosis of Hepatocellular Carcinoma[J]. Int J Gen Med, 2020,13:193-200.
[6]?CHO H J,BAEK G O,SEO C W,et al.Exosomal microRNA-4661-5p-based serum panel as a potential diagnostic biomarker for early-stage hepatocellular carcinoma[J].Cancer Med,2020,9(15):5459-5472.
[7]?MA X L, TANG W G, YANG M J, et al. Serum STIP1, a Novel Indicator for Microvascular Invasion, Predicts Outcomes and Treatment Response in Hepatocellular Carcinoma[J]. Front Oncol, 2020,10:511.
[8]?IYER M K, NIKNAFS Y S, MALIK R, et al. The landscape of long noncoding RNAs in the human transcriptome[J]. Nat Genet, 2015,47(3):199-208.
[9]?ZHANG X P, WANG W, ZHU W D, et al. Mechanisms and Functions of Long Non-Coding RNAs at Multiple Regulatory Levels[J]. Int J Mol Sci, 2019,20(22):5573.
[10]?PANDEY R R, KANDURI C. Transcriptional and Posttranscriptional Programming by Long Noncoding RNAs[J]. Prog Mol Subcell Biol, 2011,51:1-27.
[11]?KLEC C, GUTSCHNER T, PANZITT K, et al. Involvement of long non-coding RNA HULC (highly up-regulated in liver cancer) in pathogenesis and implications for therapeutic intervention[J]. Expert Opin Ther Targets, 2019,23(3):177-186.
[12]?HUANG Z, ZHOU J K, PENG Y, et al. The role of long noncoding RNAs in hepatocellular carcinoma[J]. Mol Cancer, 2020,19(1):77.
[13]?HUANG J F, GUO Y J, ZHAO C X, et al. Hepatitis B virus X protein (HBx)-related long noncoding RNA (lncRNA) down-regulated expression by HBx (Dreh) inhibits hepatocellular carcinoma metastasis by targeting the intermediate filament protein vimentin[J]. Hepatology, 2013,57(5):1882-1892.
[14]?XU D, YANG F, YUAN J H, et al. Long noncoding RNAs associated with liver regeneration 1 accelerates hepatocyte proliferation during liver regeneration by activating Wnt/β-catenin signaling[J]. Hepatology, 2013,58(2):739-751.
[15]?LAN T, CHANG L, WU L, et al. Downregulation of ZEB2-AS1 decreased tumor growth and metastasis in hepatocellular carcinoma[J]. Mol Med Rep, 2016,14(5):4606-4612.
[16]?魏超, 張曉, 高杰. 長鏈非編碼RNA與mRNA在胰腺癌中的差異表達及其預后價值[J]. 癌變·畸變·突變, 2019,31(2):119-126,132.
[17]?RITCHIE M E, PHIPSON B, WU D, et al. limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Res, 2015,43(7):e47.
[18]?BOLSTAD B M, IRIZARRY R A, STRAND M, et al. A comparison of normalization methods for high densityoligonucleotide array data based on variance and bias[J]. Bioinformatics, 2003, 19(2): 185-193.
[19]?CHAUHAN R, LAHIRI N. Tissue-?and Serum-Associated Biomarkers of Hepatocellular Carcinoma[J]. Biomark Cancer, 2016,8(Suppl 1):37-55.
[20]?TORNIO A, BACKMAN J T. Cytochrome P450 in Pharmacogenetics: An Update[J]. Adv Pharmacol, 2018,83:3-32.
[21]?ALZAHRANI A M, RAJENDRAN P. The Multifarious Link between Cytochrome P450s and Cancer[J]. Oxid Med Cell Longev, 2020,2020:3028387.
[22]?LIU C Y,LIN H H,TANG M J,et al.Vimentin contributes to epithelial-mesenchymal transition cancer cell mechanics by mediating cytoskeletal organization and focal adhesion maturation[J].Oncotarget,2015,6(18):15966-15983.
[23]?SHAH P P,FONG M Y,KAKAR S S.PTTG induces EMT through integrin αVβ3-focal adhesion kinase signaling in lung cancer cells[J].Oncogene,2012,31(26):3124-3135.
[24]?MAYER I A,ARTEAGA C L.The PI3K/AKT pathway as a target for cancer treatment[J].Annu Rev Med,2016,67:11-28.
[25]?RUIZ-SAENZ A, DREYER C, CAMPBELL M R, et al. HER2 Amplification in Tumors Activates PI3K/Akt Signaling Independent of HER3[J]. Cancer Res, 2018,78(13):3645-3658.
[26]?LIAN J P,ZOU Y H,HUANG L,et al.Hepatitis B virus upregulates cellular inhibitor of apoptosis protein 2 expression via the PI3K/AKT/NF-κB signaling pathway in liver cancer[J].Oncol Lett,2020,19(3):2043-2052.
[27]?JABOńSKA-TRYPU A, MATEJCZYK M, ROSOCHACKI S. Matrix metalloproteinases (MMPs), the main extracellular matrix (ECM) enzymes in collagen degradation, as a target for anticancer drugs[J]. J Enzyme Inhib Med Chem, 2016,31(sup1):177-183.
[28]?MO S J, HOU X, HAO X Y, et al. EYA4 inhibits hepatocellular carcinoma growth and invasion by suppressing NF-κB-dependent RAP1 transactivation[J]. Cancer Commun (Lond), 2018,38(1):9.
[29]?FANG X,TANG Z,ZHANG H,et al.Long non-coding RNA DNM3OS/miR-204-5p/HIP1 axis modulates oral cancer cell viability and migration[J].J Oral Pathol Med,2020,49(9):865-875.
[30]?WANG S, NI B, ZHANG Z, et al. Long non-coding RNA DNM3OS promotes tumor progression and EMT in gastric cancer by associating with Snail[J]. Biochem Biophys Res Commun, 2019,511(1):57-62.
[31]?BI H Q,LI Z H,ZHANG H.Long noncoding RNA HAND2-AS1 reduced the viability of hepatocellular carcinoma via targeting microRNA-300/SOCS5 axis[J].Hepatobiliary Pancreat Dis Int,2020,19(6):567-574.
[32]?VAN DIJK M,VISSER A,BUABENG K M,et al.Mutations within the LINC-HELLP non-coding RNA differentially bind ribosomal and RNA splicing complexes and negatively affect trophoblast differentiation[J].Hum Mol Genet,2015,24(19):5475-5485.
[33]?VAN DIJK M,THULLURU H K,MULDERS J,et al.HELLP babies link a novel lincRNA to the trophoblast cell cycle[J].J Clin Invest,2012,122(11):4003-4011.
[34]?LI Z B, CHU H T, JIA M, et al. Long noncoding RNA LINC01139 promotes the progression of hepatocellular carcinoma by upregulating MYBL2 via competitively binding to miR-30 family[J]. Biochem Biophys Res Commun, 2020,525(3):581-588.
[35]?LANG C, DAI Y, WU Z, et al. SMAD3/SP1 complex-mediated constitutive active loop between lncRNA PCAT7 and TGF-β signaling promotes prostate cancer bone metastasis[J]. Mol Oncol, 2020,14(4):808-828.
[36]?LIU Q, WU Y, XIAO J, et al. Long Non-Coding RNA Prostate Cancer-Associated Transcript 7 (PCAT7) Induces Poor Prognosis and Promotes Tumorigenesis by Inhibiting mir-134-5p in Non-Small-Cell Lung (NSCLC)[J]. Med Sci Monit, 2017,23:6089-6098.
[37]?LIU Y, TAO Z, QU J, et al. Long non-coding RNA PCAT7 regulates ELF2 signaling through inhibition of miR-134-5p in nasopharyngeal carcinoma[J]. Biochem Biophys Res Commun, 2017,491(2):374-381.
[38]?VERZELLA D, PESCATORE A, CAPECE D, et al. Life, death, and autophagy in cancer: NF-κB turns up everywhere[J]. Cell Death Dis, 2020,11(3):210.
[39]?DELIHAS N.Formation of a family of long intergenic noncoding RNA genes with an embedded translocation breakpoint motif in human chromosomal low copy repeats of 22q11.2—some surprises and questions[J].NcRNA,2018,4(3):16.
[40]?DELIHAS N. A family of long intergenic non-coding RNA genes in human chromosomal region 22q11.2 carry a DNA translocation breakpoint/AT-rich sequence[J]. PLoS One, 2018,13(4):e0195702.
[41]?WANG Y, SHI F, XING G H, et al. Protein Regulator of Cytokinesis PRC1 Confers Chemoresistance and Predicts an Unfavorable Postoperative Survival of Hepatocellular Carcinoma Patients[J]. J Cancer, 2017,8(5):801-808.
[42]?LIU X, LI Y, MENG L, et al. Reducing protein regulator of cytokinesis 1 as a prospective therapy for hepatocellular carcinoma[J]. Cell Death Dis, 2018,9(5):534.
[43]?LI M,GAO J,LI D,et al.CEP55 promotes cell motility via JAK2dSTAT3dMMPs cascade in hepatocellular carcinoma[J].Cells,2018,7(8):99.
[44]?FU Y,FENG M X,YU J,et al.DNA methylation-mediated silencing of matricellular protein dermatopontin promotes hepatocellular carcinoma metastasis by α3β1 integrin-Rho GTPase signaling[J].Oncotarget,2014,5(16):6701-6715.
[45]?LI X,FENG P,OU J,et al.Dermatopontin is expressed in human liver and is downregulated in hepatocellular carcinoma[J].Biochemistry:Mosc,2009,74(9):979-985.
[46]?HA M, SON Y R, KIM J, et al. TEK is a novel prognostic marker for clear cell renal cell carcinoma[J]. Eur Rev Med Pharmacol Sci, 2019,23(4):1451-1458.
[47]?OLIVEIRA-FERRER L,MILDE-LANGOSCH K,EYLMANN K,et al.Mechanisms of tumor-lymphatic interactions in invasive breast and prostate carcinoma[J].Int J Mol Sci,2020,21(2):602.
[48]?VILLA E,CRITELLI R,LEI B,et al.Neoangiogenesis-related genes are hallmarks of fast-growing hepatocellular carcinomas and worst survival.Results from a prospective study[J].Gut,2016,65(5):861-869.
(收稿日期:2020-10-26?修回日期:2020-12-04)
(編輯:潘明志)

