RobustDEA:一種快速魯棒的RNA-Seq數據尋找差異表達基因方法
張 禮, 王嘉瑞, 吳東洋
(南京林業大學 信息科學與技術學院,南京 210016)
下一代高通量DNA測序技術已經得到快速發展和普及,轉錄組測序技術(RNA-Sequencing,RNA-Seq)具有高通量,高信噪比,可重復性好,所需樣本少等特點,被廣泛應用的轉錄組學的各個研究領域中[1].尋找差異基因表達分析作為RNA-Seq數據分析中最基本應用之一,其目的是在不同對照組和控制組樣本中,比如正常狀態和藥物治療,健康和疾病個體,不同生物組織或者不同發育階段等,識別出具有差異表達的基因.從RNA-Seq測序實驗中獲得海量讀段數據后,首先需要將讀段數據定位到參考基因組或者轉錄組上,從而統計出每個基因或者外顯子上的讀段數目.一旦獲得基因或者外顯子的讀段數目,就可以進行差異表達分析.盡管RNA-Seq技術相比傳統基因芯片具有很多優勢,但是由于RNA-Seq測序本身技術上限制產生了很多困難,比如多源讀段映射,測序偏好和小樣本等問題,導致差異表達分析仍然是一個極具挑戰的任務.
針對尋找差異表達基因所面臨的種種挑戰,提出了大量統計方法,通常可以分為兩類:基于讀段數目的方法和基于表達水平的兩步法.基于讀段數目的方法是比較在不同樣本條件中基因上映射的讀段數目.由于在不同樣本之間,基因上讀段數目變異性較大,若采用泊松分布會導致讀段分布存在過散步(overdispersion)問題.因此,基于讀段數目方法通常采用負二項(Negative binomial)分布,比如DESeq[2],DESeq2[3],baySeq[4],edgeR[5]等.相比泊松分布的單參數,負二項分布增加一個參數,能單獨描述數據方差特性,從而能解決讀段數據的過散步問題.此外,Voom使用正太線性模型來擬合讀段數據,并且嵌入了數據分布的均值和方差的相關性信息[6].SAMSeq[7],NOISeq[8]以及NPEBSeq[9]等非參數模型,無需假設基因讀段分布服從任何概率分布.但這類方法都忽略了讀段的多源映射和數據偏差等問題,因此提出了基于表達水平的兩步法:第一步利用現有表達水平估計方法計算出基因的表達水平,比如Cufflinks[10],PGSeq[11]等,這些方法在計算過程中考慮了讀段多源映射和數據偏差等問題,從而獲得更為準確的基因表達水平;第二步是利用獲得的基因表達水平進行差異分析,比如CuffDiff2與Cufflinks,BDSeq[12]與PGSeq.由于考慮了更多的數據特征,基于表達水平的兩步法通常能獲得較好的結果.但由于兩步法需要預先計算基因表達水平,其時間效率要比基于讀段數目的方法低很多.因此,在面向尋找差異表達基因的單一任務時,目前主流做法是選擇基于讀段數目的方法,在較短的時間內獲得較高的準確度[13].
針對RNA-Seq數據分析發現,同一個數據集中的基因,采用不同的尋找差異表達基因方法中會得到差異較大的結果[14].采用DESeq,baySeq,edgeR和Voom 4個方法來尋找MAQC數據集中的差異表達基因,表1展示了兩個基因在不同方法中的P值和差異重要性排序,說明不同方法在分析同一個數據集是存在不一致性.導致這種現象除了數據集本身的生物特異性差異,以及不同研究者發表的基因表達數據的質量差異外,不同模型的假設不同或特點數據集偏好性,如NOISeq比較適合小樣本數據集[15].此外,不同方法的結果報告格式不一致通常會導致結果理解和推斷的困難.

表1 不同方法下基因載MAQC數據集上的結果
為了克服上述的問題,一種潛在可行方法是將當前算法結合起來,以獲得更魯棒的差異基因列表,其特點在于具有更高的統計能力,更少的假陽性和假陰性差異基因.研究人員通常是結合不同方法在同一個數據集上分析結果,從而推斷出更穩定的結論.IDEAMEX是一個在線差異表達基因檢測軟件[16],該軟件首先預設閾值得到每個方法所識別出的差異表達基因,在采用簡單的投票方式選出4個方法都公認差異表達基因列表.consensusDE[17]和MultiRankSeq[13]方法都采用了基因排名求和的思想.首先不同方法根據p-value值大小對基因分別進行排序,然后將基因在不同方法中的排名求和,從而得到基因最終排名序列,排名越靠前的基因其越差異表達的可能性就越大.不同之處在于,MultiRankSeq方法結合了DESeq,edgeR和baySeq 3個方法,數據驗證顯示,DESeq和edgeR的差異基因集很接近,而baySeq找到的差異基因卻和上述兩個方法重疊的部分較少.圖1是4個方法前1 000個最有可能的差異基因的Venn圖,可以發現baySeq方法與其他3個方法的重疊基因很少,這將導致MultiRankSeq的結果易受到baySeq的影響.因此consensusDE方法采用了DESeq2,edgeR和Voom 3個方法,并在數據預處理步驟考慮消除不變基因的影響.上述方法通過投票或者排名求和的結合方式,找到被所結合方法共同認為最有可能的差異基因,但這些方法容易受到結合方法中單個方法的影響,反而降低了方法的準確性和魯棒性.為了減輕單個方法對融合結果的影響,PANDORA方法對DESeq,edgeR,Vomm,NBPSeq,NOISeq和baySeq 6個方法進行加權融合[18].但是PANDORA方法需要預先產生與測試數據集特征相近的模擬數據集,通過模擬數據集計算出每個方法的權重值,此過程極其耗時.此外,PANDORA和MltiRankSeq都采用了DESeq,而不是性能更優越的DESeq2.
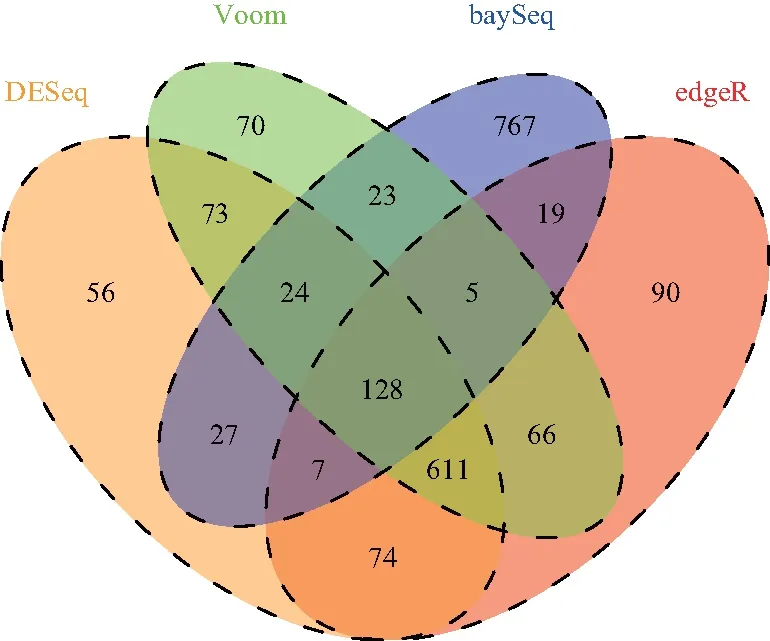
圖1 DESeq, edgeR, Voom和baySeq
針對上述方法存在的問題,文中提出了一種魯棒的尋找差異表達基因表達方法(Robust differential expression analysis based on RNA-Seq data,RobustDEA).RobustDEA方法選擇了不同數據集中表現較好且穩定的4個方法:DESeq2, Voom, edgeR和NOISeq,通過加權方式結合多個尋找差異表達基因方法,其權值可快速從數據集中學習獲到,并能有效的反映數據集的特點,從而使得RobustDEA方法可以在不同數據集上都能獲得更穩定的結果.通過多個真實RNA-Seq測序數據集的驗證表明,RobustDEA方法能在不同數據集上獲得更加魯棒的差異基因集.
1 方法
1.1 常見結合方法
基因芯片數據和RNA-Seq數據的差異表達基因分析中,多種結合方法被廣泛應用到對不同方法檢測結果的融合.目前,應用最為廣泛的兩種常規統計方法和專門針對RNA-Seq數據的最流行 PANDORA方法.首先,假設基因i使用尋找差異表達基因方法j獲得其對應的P值pij,然后可以使用下面幾種結合方法來進行不同方法結果的融合.
1.1.1 Fisher方法
根據Fisher方法,基因i通過m個方法估計的P值合并為:
(1)
合并后得到的統計量符合自由度為2m的卡方分布.χ2值越大,P值就越小,表示對應的基因其差異性就越明顯.但是Fisher方法設計目的是合并具有相同零假設的統計檢驗方法在不同數據集中產生的P值,并不是合并同一個數據集上由不同統計檢驗方法產生的P值.
1.2.2 Stouffer方法
針對每個基因,標準的Stouffer方法首先將pj值轉換為Zj=Φ-1(1-pj),其中Φ是標準正態分布的累積分布函數,每個基因的Z值為:
(2)
根據Z值大小對基因差異表達排序.標準Stouffer方法的優點易于為不同方法賦予不同權重值,因此提出改進的加權Stouffer方法:
(3)
式中:ωj為方法j的權重,其值可根據具體問題來選擇,比如數據集的樣本規模等.Z值越大,對應P值就越小,表示該基因的差異性就越顯著.Stouffer方法和Fisher方法密切相關,其設計目的的假設都是合并具有相同零假設的統計檢驗方法在不同數據集中產生的P值.但加權Stouffer方法能便于對不同方法進行加權,而Fisher方法的卡方統計量,存在加權困難問題.
1.2.3 PANDORA方法
PANDORA方法的模型為:
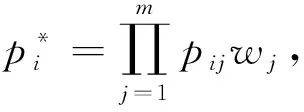
(4)
式中:ωj為方法j的權重,其值自動從模擬數據集中獲得[18].此模擬數據集是基于正研究的數據集而產生,因此其權值能更為貼近研究的數據集.但模擬數據集的產生極其耗時,與結合方法的個數,測序樣本數,基因個數等諸多因素有直接關系.
1.2 RobustDEA方法
針對不同尋找差異表達基因方法檢測得到的差異基因集存在不一致情況,基于統計學中元分析的思想,文中提出一個快速魯棒的RNA-Seq數據尋找差異表達基因方法RobustDEA,結合多個不同的差異表達基因方法,其模型為:
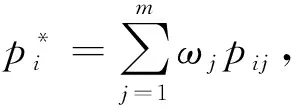
(5)
式中:ωj為方法j的權重值.RobustDEA方法采用自動加權方式,其權重值從數據集中自動計算得到.結合的每個方法先分別對數據集進行分析,獲得每個基因的P值,可計算出每個方法在此數據集上的接收者操作特征曲線(receiver operating characteristic curve,ROC)下的面積(area under curve,AUC)值,從而自動估計出每個方法所對應的權重值為:
(6)
式中:AUCj為方法j的AUC值.通過自動加權方式,可以獲得與當前分析數據集最合適的權重值,有利于提高RobustDEA方法的精確度和魯棒性.
在RNA-Seq數據集,計算各個方法的AUC值需要大量基準基因(即已知差異表達的基因),基準基因的差異表達值需要通過實時熒光是量聚合酶鏈反應技術(quantitative real-time PCR,qRT-PCR)生物實驗來確定.而現實中包含PCR數據的RNA-Seq數據集是極其少的.因此,RobustDEA方法采用了一種相互交叉差異基因集的驗證方式來計算各個方法的AUC值,從而避開對PCR數據的依賴.針對每個方法,先計算并排序每個基因的P值,選取前N個基因(N通常選擇2 000),這部分基因被認為最有可能的差異表達基因.再計算多個方法的差異基因的交集.交集中基因是被共同認可的差異基因,以此作為該數據集的基準基因,用來計算自身的AUC值,其算法的具體過程如算法1.

算法1:相互交叉差異基因集的驗證AUC值計算方法輸入:m個方法對差異基因列表(根據P值排序)Repeat:對于方法j 1. 分別選取其它方法的前N個差異表達基因; 2. 前N個差異表達基因的交集; 3. 以交集中基因作為基準基因; 4. 計算AUCj值Until:所有方法的AUC值計算完畢輸出:根據各個方法的AUC值計算權重大小
通過AUC值計算的相互交叉驗證,可避免PANDORA方法中需要預先使用模擬數據集去計算各個方法權重的過程,不僅可大幅度的提高權重的計算效率,同時也能保留數據集的特點.
1.3 評估準則
選取了多種評估準則來評估不同方法和驗證RobustDEA方法的性能.當RNA-Seq數據集包含PCR驗證基因,或者將結合方法共同認可的差異基因作為基準基因,選擇F1值來評估方法性能.F1值是對精確率(Precision)和召回率(Recall)的加權調和平均,其定義為:
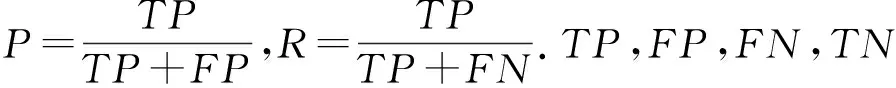
此外,進一步采用ROC曲線和AUC值評估按方法的預測準確性.ROC曲線圖是反映橫坐標假陽性率(false positive rate, FPR)與縱坐標真陽性率(true positive rate, TPR)之間關系的曲線,其中:
AUC值表示ROC曲線下面積,其值越大表示正樣本越有可能被排在負樣本之前,即方法的預測性越好.
1.4 RobustDEA實現
為了便于用戶使用,RobustDEA方法采用R語言實現,且選擇的結合方法都在Bioconductor中提供了R語言包,避免了用戶額外安裝其它基于Unix的軟件,比如Cuffdiff2等.
RobustDEA方法的R語言包和詳細文檔可以從網址https://github.com/BIO-LAB/RobustDEA下載,供全球生物學家免費使用.
2 實驗結果與討論
2.1 數據集
為了評估RobustDEA方法,文中選擇了ReCount數據庫中的3個真實RNA-Seq數據集[19],分別是人類大腦數據集MAQC,老鼠數據集Katz和小鼠數據集Hammer.
人類大腦MAQC數據集來自美國藥品監督局(FDA)聯合全球多所研究機構進行的“生物芯片質量控制”項目[20].MAQC數據集包含2個條件,正常大腦組織(HBR)和病變大腦組織(UHR),每個條件下包含7個重復試驗樣本,總計包含52 580個基因.過濾掉在所有樣本中讀段數目都是零的基因,獲得11 907個具有表達的基因.此外,MAQC項目提供了1 000個qRT-PCR驗證基因.通過篩選具有顯著變化的基因(FC<=0.5或FC>=2.0),獲得305個驗證基因作為標準基因,用來評估不同方法尋找差異表達基因的準確性.
大鼠數據集Katz包含兩個實驗條件,正常成肌細胞和敲掉CUGBP1的成肌細胞,每個條件包含2個生物性重復實驗,總計包含36 536個基因.過濾掉在所有樣本中讀段數目都是零的基因,獲得9 501個具有表達的基因.
小鼠數據集Hammer是關于小鼠脊柱神經發育的研究,包含胚胎發育后2周和2月兩個時間點.每個時間點包含2個條件,正常脊柱神經細胞和進行脊柱神經結扎的神經細胞.每個條件包含2個生物性重復樣本,總計包含29 516個基因,過濾掉在不同時間點時樣本中讀段數目都是零的基因,兩個時間點分別獲得17 125和18 463個具有表達的基因.
2.2 結合方法的選擇
目前,提出了大量尋找差異基因表達方法,結合方法的選擇會對RobustDEA方法的穩定性和性能影響較大.因此,在不同數據集中性能穩定且時間效率高的方法作為RobustDEA的結合方法的最有選擇.根據多個綜述性評估不同方法的精確度和時間效率(如表2),文中選擇DESeq2(v1.26.0) ,edgeR(v3.28.1),NOISeq(v2.30.0) 和Voom(v3.42.2)4種方法作為RobustDEA的原始方法.PANDORA方法來自R包metaseqR(v1.26.0).
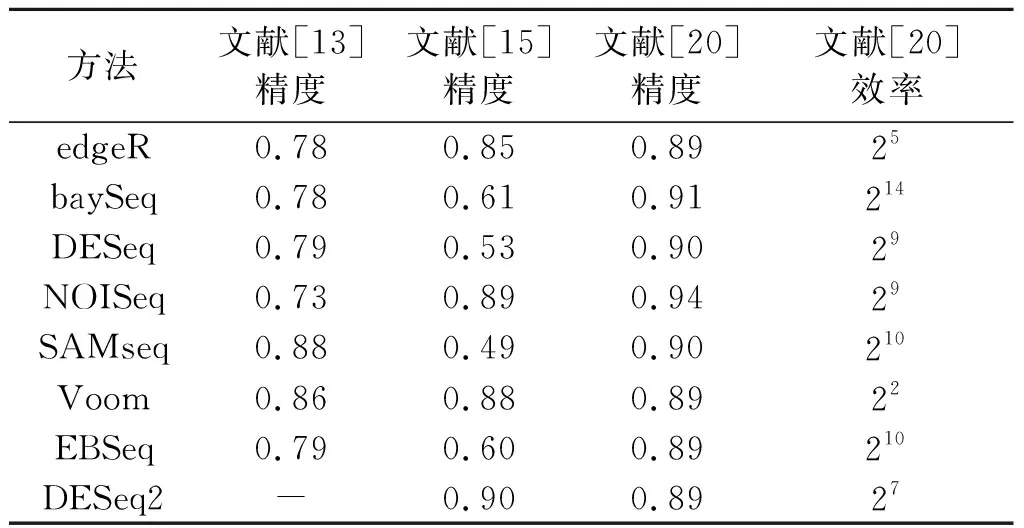
表2 尋找差異表達基因方法在不同數據集上的性能
2.3 MAQC數據集上qRT-PCR標準基因驗證
經過qRT-PCR生物實驗驗證過的基因往往被當做標準基因,用來評估生物信息學中各種方法的準確度.通過注釋文件和倍數比率的篩選,MAQC數據集獲得了305個qRT-PCR基因,被用來評估不同尋找差異方法的準確性.圖2顯示不同方法在MAQC數據集上的ROC曲線.其中圖2(a)為RobustDEA方法和其他4個原始方法的比較.結果表明,RobustDEA方法和Voom方法的ROC曲線接近重合,但是要明顯要好于DESeq2,edgeR和NOISeq 3個方法.這表明在RobustDEA方法在結合不同方法的結果之后,至少可獲得不低于原始方法中最優性能.圖2(b)為RobustDEA方法與其他3種結合方法的比較結果.從圖中可以看出,RobustDEA方法的ROC曲線要明顯高于其他3個結合方法.Fisher,Stouffer和PANDORA方法得到比原始方法更差的結果,說明這些方法不夠穩定,容易受到原始方法中較差結果的影響.
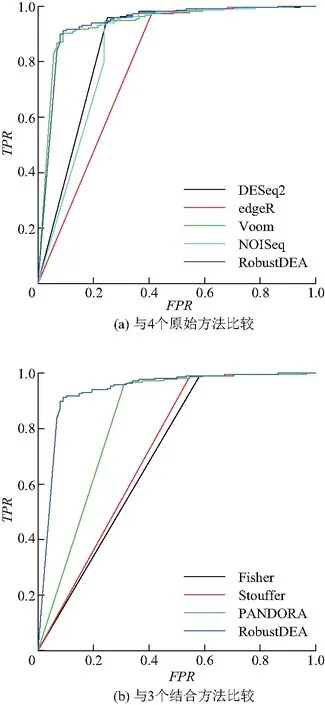
圖2 不同方法在MAQC數據集上的ROC曲線
圖3為原始方法和結合方法在MAQC數據集上的AUC值和F1值,從這兩個值均可說明RobustDEA方法獲得了最好準確度,表明該方法相比于其他結合方法,不易受到較差性能的原始方法影響,具有更好的魯棒性.
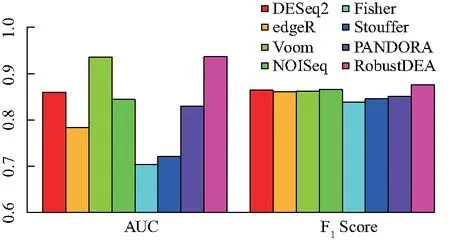
圖3 不同方法在MAQC數據集上的AUC值和F1值
2.4 Katz和Hammer數據集的大規模基因驗證
MAQC數據集的僅提供了305個qRT-PCR驗證基因,缺少大量基因的評估.但是由于qRT-PCR實驗的費用問題,現實中缺少包含大量qRT-PCR驗證基因的數據集.為了進一步在大規模基因上評估RobustDEA方法的性能,采用交互驗證方式構建大規模差異基因數據集[11].首先分別選擇4個原始方法各自認為最可能是差異表達的前2 000個基因.將這些基因合并成一個驗證基因集,其中4個方法共同認定為差異表達基因作為“真實“差異表達基因,其余為非差異表達基因.
在Katz數據集中,通過交互驗證方式構建的驗證基因集包含2 501個基因,其中1 517個基因被共同認定為差異表達基因,其余基因即被劃分為非差異基因.通過此驗證基因集的評估,不同方法的ROC曲線如圖4.
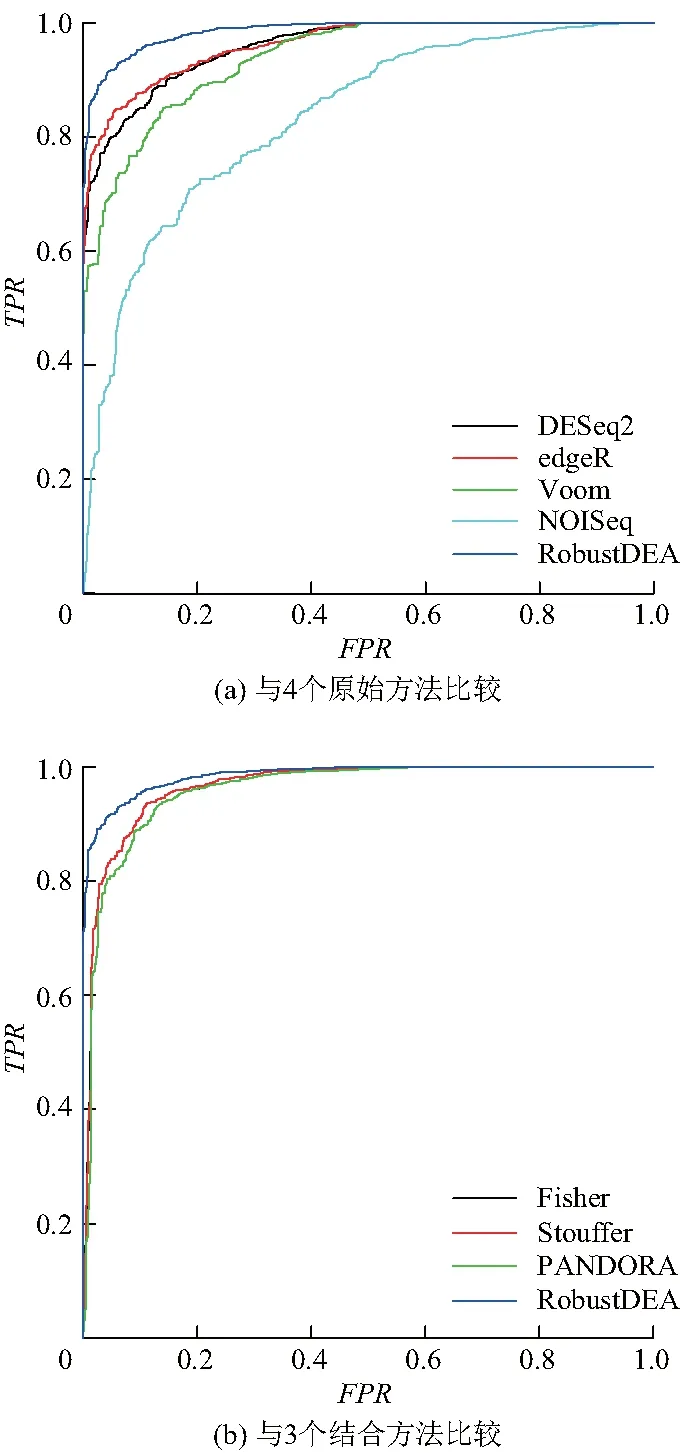
圖4 不同方法在Katz數據集中的ROC曲線
從圖4(a)中可以看出,RobustDEA方法比4個單獨原始方法的性能都要好.從圖4(b)可以獲得4個結合方法都獲得較高的準確度(圖4(b)中,PANDORA和Fisher的ROC曲線高度重合).而相比3個結合方法,RobustDEA方法同樣表現出最好的性能.
Hammer數據集包含兩周后和兩月后兩個時間點,通過交互驗證方式分別構建驗證基因集.兩周后時間點的驗證基因集包含2 540個基因,其中1 484個基因被共同認定為差異表達基因.兩月后時間點的驗證基因集包含2 995個基因,其中1 088個基因被認定為差異表達基因.在驗證基因集中其余基因為非差異表達基因.通過兩個驗證基因集的評估,圖5(a)和(c)顯示RobustDEA方法的ROC曲線要高于其他4個單獨原始方法,表明RobustDEA方法通過結合不同方法能進一步提升性能.圖5(b)和(d)中與其他3個結合方法進行比較,可以看出RobustDEA和PANDORA方法表現穩定,而Fisher和Stouffer方法性能有大幅度下降.
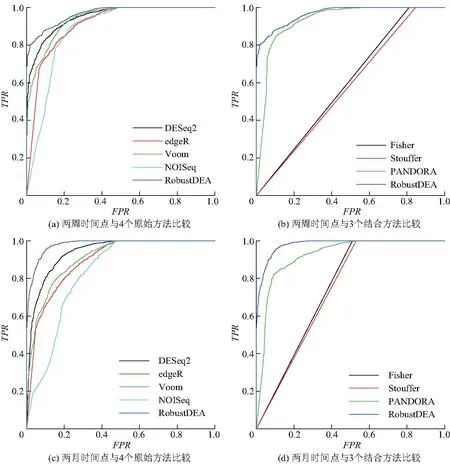
圖5 不同方法在Hammer數據集中2個時間點的ROC曲線
表3為不同方法在Katz和Hammer數據集上AUC值,結果表明在3個大規模基因數據集上,RobustDEA方法均可獲得最高的性能.雖然PANDORA方法在不同數據集中會比其中某個單獨原始方法性能要略低,但是在整體上能獲得較為穩定的結果,不會受到性能較差的NOISeq方法的影響.Fisher和Stouffer方法表現極為不穩定,在Katz數據集中能獲得極高的準確度,但是在Hammer數據集的2個時間點上性能都大幅度下降.導致此問題的原因在于Fisher方法對不同原始方法賦予相同的權重,容易受到性能較差方法的干擾.而RobustDEA和PANDORA方法對根據原始方法的性能進行加權,可以降低性能較差方法的影響,從而使性能更加表現更加穩定,具有較高的魯棒性.
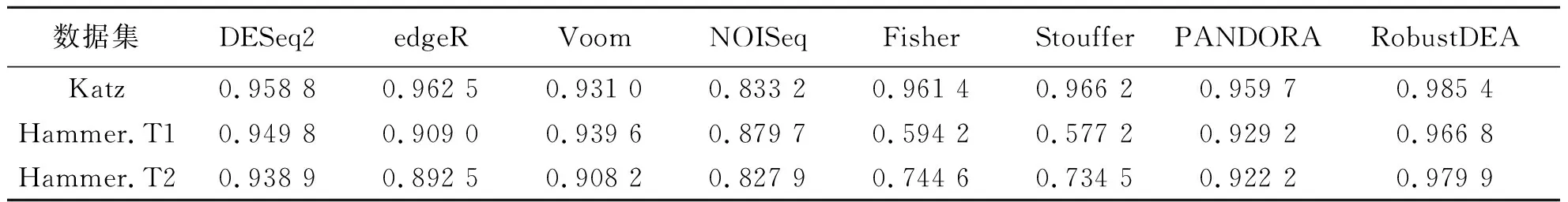
表3 不同方法在Katz和Hammer數據集上AUC值
2.5 時間效率的分析
結合方法的評估過程分為原始方法計算,權重計算和結合評估3個部分.對于所有結合方法來說,原始方法計算所消耗時間都是一致,區別主要在于權重計算部分.PANDORA方法的權重估計是根據現有數據特點去生成模擬數據集,然后再通過計算不同原始方法在模擬數據集上的精度作為權重.這部分的計算需要花費大量時間,導致PANDORA方法的時間效率大幅度下降.并且PANDORA方法的時間消耗與原始方法個數,模擬數據集中樣本量,基因個數等參因數有直接影響.RobustDEA方法的權重估計是基于原始方法計算的結果,因此能快速獲得不同方法的權重值,極大了提高了計算效率.表4為4個結合方法在MAQC數據集上的計算效率.PANDORA方法設置模擬數據集為4個測序樣本,1 000個基因.RobustDEA方法選擇N為1 000,其時間為同一工作站的CPU工作時間,性能為2.9 GHz雙核和8 G內存,采用單核計算.從表中結果可以看出PANDORA方法花費了整個時間894.6 s中的95%時間進行權重計算,而RobustDEA方法只需要花費5.5 s的時間估計權重值,大幅度提高了效率.雖然RobustDEA方法計算效率僅略高于Fisher和Stouffer方法,但其計算精度和穩定性要明顯高于這兩個方法.
表4 不同方法在MAQC數據集上計算效率
3 結論
針對不同尋找差異表達基因方法檢測得到的差異基因集不一致問題,提出了一種快速魯棒的RNA-Seq數據尋找差異表達基因的結合方法.通過人類大腦MAQC數據集和多個老鼠數據集的評估,得出如下結論:
(1) RobustDEA方法結合現有尋找差異表達基因方法,通過全新的權重評估方式,可快速獲得與當前分析數據集最合適的權重值.
(2) 相比于單個差異表達基因方法和現有結合方法,RobustDEA方法能獲得更加準確和穩定的結果.這表明該方法具有較好的魯棒性,受到性能較差的原始方式的影響較小.
(3) 相比PANDOR方法,RobustDEA方法采用全新的權重估計方式,可大幅度提高計算效率.
今后工作將RobustDEA方法實現為一個可視化在線平臺,用戶只需導入基因讀段數據,平臺可快速計算出差異基因集,并提供相應的分析報告,以及更加直觀的圖表展示,從而讓用戶使用更加便利.

