凡納濱對蝦進口親蝦子一代群體的遺傳變異分析
徐煜 黃小帥 胡曉娟 徐武杰 蘇浩昌 文國樑 曹煜成 楊鏗





摘要:【目的】明確凡納濱對蝦(Litopenaeus vannamei)進口親蝦直接繁殖子一代種苗群體的遺傳變異情況,旨在評估遺傳特征與養殖生產性能的關聯性,為我國養殖對蝦優質種苗大數據建設及質量評估體系的完善提供基礎數據支持。【方法】以來源于正大神灣蝦苗場的Stock 1~Stock 5群體、正大漳浦蝦苗場的Stock 6群體、海尚種苗培育基地的Stock 7和Stock 8群體及中海水產種苗科技有限公司的Stock 9群體等9個不同批次凡納濱對蝦進口親蝦子一代種苗為研究對象,利用11個微衛星分子標記(M1、Pvan1758、Pvan1815、HLJN-008、HLJN-023、TUMXLv7.121、TUMXLv8.256、TUMXLv9.43、TUMXLv10.312、TUYFLvL16.1a和TUDGLv1-3.224)對不同凡納濱對蝦種苗群體進行遺傳變異分析。【結果】9個凡納濱對蝦種苗群體的等位基因數(Na)、有效等位基因數(Ne)、觀測雜合度(Ho)、期望雜合度(He)、Shannon’s信息指數(I)和多態信息含量(PIC)的平均值分別在5.5455~8.8182、3.2795~5.4735、0.3769~0.5182、0.6189~0.6948、1.2823~1.6092和0.5863~0.6646,即各凡納濱對蝦種苗群體均具有較高的遺傳多樣性,具體排序為Stock 3>Stock 2>Stock 4>Stock 5>Stock 7>Stock 6>Stock 9>Stock 8>Stock 1。在9個凡納濱對蝦種苗群體中有91.82%的遺傳變異源自群體內,僅有8.18%的變異源自群體間。Stock 2、Stock 3、Stock 4和Stock 5等4個群體間及Stock 7群體與Stock 8群體間的遺傳分化系數(Fst)均小于0.05(0.0079~0.0371),即群體間無遺傳分化,而其他群體間表現為輕度遺傳分化(0.05<Fst <0.15)。基于Nei’s遺傳距離,9個凡納濱對蝦種苗群體大致可劃分為三大分支,第一分支包括Stock 1~Stock 6群體,第二分支由Stock 7群體和Stock 8群體組成,而Stock 9群體獨立為第三分支。【結論】不同批次凡納濱對蝦進口親蝦子一代種苗群體的遺傳特征存在明顯差異,各群體間的遺傳距離隨著采樣時間和地點的改變而發生明顯變化,且這些遺傳背景差異可能會造成不同群體在養殖生產性能方面產生差異。
關鍵詞: 凡納濱對蝦;進口親蝦;子一代群體;遺傳變異;微衛星分子標記
中圖分類號: S945.49? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2021)11-3139-08
Genetic variation analysis among first filial generation of introduced Litopenaeus vannamei stocks
XU Yu1,2, HUANG Xiao-shuai1, HU Xiao-juan1,2, XU Wu-jie1,2, SU Hao-chang1,2,
WEN Guo-liang1, CAO Yu-cheng1,2*, YANG Keng1*
(1South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences/Key Laboratory of South China Sea Fishery Resources Exploitation & Utilization, Ministry of Agriculture and Rural Affairs/Guangdong Provincial Key Laboratory of Fishery Ecology and Environment, Guangzhou? 510300, China; 2 Shenzhen Base of South China Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Shenzhen, Guangdong? 518121, China)
Abstract:【Objective】The main purpose of this study was to analyze and assess the genetic diversity of first filial ge-neration of Litopenaeus vannamei, in order to evaluate the correlation between its genetic characteristics and aquaculture production performance, and provide basic data support for the big data construction and establishment of quality evaluation system of high-quality post-larvae in China. 【Method】The objects were L. vannamei Stock 1-Stock 5 from Zhengda Shenwan shrimp larvae farm, Stock 6 from Zhengda Zhangpu shrimp larvae farm, Stock 7 and Stock 8 from Haishang shrimp larvae farm, Stock 9 from Zhonghai aquatic larvae culture company. The genetic variation of nine first filial gene-ration stocks were investigated using eleven microsatellite DNA markers(M1, Pvan1758, Pvan1815, HLJN-008, HLJN-023, TUMXLv7.121, TUMXLv8.256, TUMXLv9.43, TUMXLv10.312, TUYFLvL16.1a and TUDGLv1-3.224). 【Result】This paper showed that the average values of the number of alleles (Na), number of effective alleles (Ne), observed heterozygosity (Ho), expected heterozygosity (He), Shannon’s diversity index (I), and polymorphism information content (PIC) were among 5.5455-8.8182, 3.2795-5.4735, 0.3769-0.5182, 0.6189-0.6948, 1.2823-1.6092 and 0.5863-0.6646, respectively. That presented a high genetic diversity of these filial generation of introduced L. vannamei stocks, and according to the richness, they ranked as follows: Stock 3>Stock 2>Stock 4>Stock 5>Stock 7>Stock 6>Stock 9>Stock 8>Stock 1. AMOVA analysis indicated that the genetic variation within stock was 91.82% and the remaining 8.18% of the variation occurred among stocks. The genetic differentiation among Stocks 2-5 and among Stocks 7-8 showed a low level based on their Fst values (0.0079-0.0371), which were lower than 0.05, whereas the other stocks had no genetic differentiation (0.05<Fst <0.15). Based on Nei’s genetic distance, the nine stocks could be roughly divided into three branches: the first branch including Stock 1 - Stock 6 stocks, the second branch being composed of Stock 7 and Stock 8, and the third branch only included Stock 9. 【Conclusion】These findings indicated that different batches of first filial generations have distinct genetic characteristics. Dramatically, genetic distance of first filial generations are clearly fluctuated with the breeding farm and breeding time. Changes of genetic background might directly affect the shrimp traits in subsequent farming.
Key words: Litopenaeus vannamei; exotic parents; first filial generation; genetic variation; microsatellite molecular marker
Foundation item: Central Public-interest Scientific Institution Basal Research Fund of Chinese Academy of Fishery Sciences(2020TD54); China Agriculture Research System of MOF and MARA(CARS-48); Guangdong Provincial Special Fund for Modern Agriculture Industry Technology Innovation Teams (2019KJ149)
0 引言
【研究意義】凡納濱對蝦(Litopenaeus vannamei)又稱南美白對蝦,隸屬于十足目(Decapoda)游泳亞目(Natantia)對蝦科(Penaeidae),是當今最主要的對蝦養殖品種。凡納濱對蝦自1988年引入后逐步發展成為我國對蝦養殖產業的主導品種,近年來我國的凡納濱對蝦養殖產量穩居世界首位(代平等,2018)。凡納濱對蝦為熱帶型種類,原產自中南美洲太平洋沿岸水域,其優良種質資源長期受到國外育種機構的壟斷(王興強等,2004;代平等,2018;彭敏等,2020)。當前全球凡納濱對蝦種蝦資源主要分布在美國的夏威夷、邁阿密和關島,以及泰國、新加坡和印尼,我國每年需從境外進口大量親蝦,由進口親蝦直接繁育子一代種苗具有生長速度快、養成群體個頭均勻等優點,在我國對蝦養殖業中占有較高的市場份額。但受種質資源退化及養殖環境惡化等因素的影響,對蝦養殖業面臨病害頻發、養殖風險劇增等突出問題(王麗花等,2012;文國樑等,2015;Qiu et al.,2017)。從不同批次進口親蝦子一代種苗的養殖實踐來看,蝦苗的成活率、抗逆性及生長周期等表現良莠不齊,嚴重影響了養殖經濟效益。因此,針對凡納濱對蝦進口親蝦直接繁殖子一代種苗群體開展遺傳變異分析,對評估其遺傳特征與養殖生產性能的關聯性具有重要意義。【前人研究進展】微衛星分子標記分析具有簡便、快捷及穩定性好等優點,已廣泛應用于種質資源評價和遺傳育種等研究領域(孫效文等,2008;趙志英等,2018;周大顏等,2019)。近年來,微衛星分子標記也常應用于水生生物遺傳多樣性分析。劉連為等(2014)利用8個微衛星位點對北太平洋柔魚(Ommastrephes bartramii)的6個群體進行分析,結果發現群體間具有較高的遺傳多樣性。毛守康等(2016)利用14個微衛星位點對4個野生梭魚(Liza haematocheila)地理群體進行遺傳多樣性分析,結果表明,4個群體遺傳多樣性處于中等多態水平,且按其地理位置由北向南依次聚為一類。Wang等(2016)利用8個微衛星位點分析中國對蝦親本和回捕群體的遺傳多樣性,發現這2個種群擁有豐富遺傳多樣性,但存在較高程度的近親繁殖,且在中國對蝦種群中呈現一定的近交衰退。黃小芳等(2020)利用8對微衛星引物對廣西不同地區克氏原螯蝦群體的遺傳多樣性進行分析,結果發現各克氏原螯蝦群體具有一定的遺傳多樣性,但其雜合子缺失現象普遍存在,應通過適當引種及加強不同地區群體間的基因交流,保護好優良種質資源。在凡納濱對蝦方面,童馨等(2007,2009)在研究不同世代凡納濱對蝦養殖群體生長性狀變異的基礎上,利用微衛星分子標記分析種群遺傳多樣性的變化,發現隨著繁育世代增加,凡納濱對蝦的生長性狀逐代分化,生長速度變緩,群體遺傳多樣性降低,種質發生退化;吳怡迪等(2016)基于微衛星分子標記對凡納濱對蝦選育系間雜交的生長性狀及遺傳多樣性進行分析,結果表明,凡納濱對蝦的生長性能與群體遺傳多樣性水平呈顯著正相關,選育系間雜交可使雜交后代的生長性狀和遺傳多樣性水平得到改善;李強勇等(2020)基于第三代高通量測序技術——單分子實時(SMRT)測序開發出凡納濱對蝦微衛星分子標記,有助于開展凡納濱對蝦遺傳育種研究工作;彭敏等(2020)從GenBank中篩選11對能有效擴增的微衛星引物對2016—2018年桂海1號凡納濱對蝦連續3個世代選育群體進行遺傳多樣性分析,監測相鄰2個世代選育群體間的遺傳變異情況,結果顯示桂海1號凡納濱對蝦各世代選育群體尚具有較豐富的遺傳變異,但群體遺傳結構處于不穩定狀態。【本研究切入點】遺傳變異是影響對蝦養殖性能表現的重要因素,但近年來鮮見從遺傳變異水平研究進口親蝦子一代種苗養殖性能差異的問題。【擬解決的關鍵問題】選取不同批次凡納濱對蝦進口親蝦子一代種苗群體,利用微衛星分子標記分析不同群體的遺傳變異及其多樣性水平,旨在評估遺傳特征與養殖生產性能的關聯性,為我國養殖對蝦優質種苗大數據建設及質量評估體系的完善提供基礎數據支持。
1 材料與方法
1. 1 試驗材料
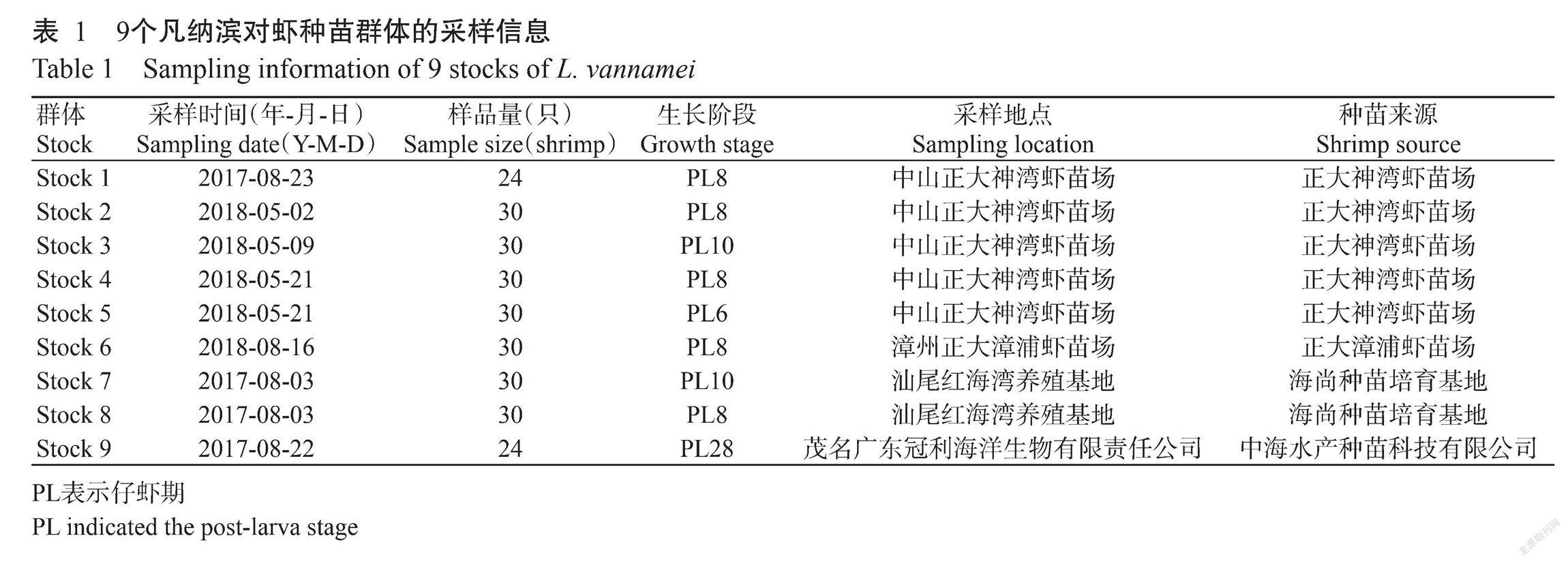
供試凡納濱對蝦種苗群體為2017—2018年收集于廣東中山、茂名、汕尾及福建漳州等地的蝦苗場或養殖場,共9個群體,分別命名為Stock 1~Stock 9。各凡納濱對蝦種苗群體樣品具體信息如表1所示。
1. 2 DNA提取
取凡納濱對蝦種苗腹部組織10~30 mg,參照E.Z.N.A. Tissue DNA Kit (OMEGA Bio-tek)試劑盒說明提取各樣品基因組DNA。采用1.0%瓊脂糖凝膠電泳檢測DNA完整性,以NanoDrop 2000紫外分光光度計測定其濃度,-80 ℃保存備用。
1. 3 微衛星位點
根據相關文獻報道,選取凡納濱對蝦中多態性高且擴增效果好的11個微衛星位點:M1(Garcia et al.,1996;Alcivar-Warren et al.,2007a)、Pvan1758(Cruz et al.,2002)、Pvan1815(Cruz et al.,2002)、HLJN-008(Jia et al.,2006)、HLJN-023(Jia et al.,2006)、TUMXLv7.121(Meehan et al.,2003)、TUMX Lv8.256(Meehan et al.,2003;Alcivar-Warren et al.,2007b)、TUMXLv9.43(Meehan et al.,2003;Alcivar-Warren et al.,2007b)、TUMXLv10.312(Meehan et al.,2003;Alcivar-Warren et al.,2007b)、TUYFLvL16.1a(Alcivar-Warren et al.,2007b)和TUDGLv1-3.224(Alcivar-Warren et al.,2007b;Garcia et al.,2007),在各位點上游引物5'端分別進行6-FAM、ROX、HEX或TAMRA熒光標記,引物序列及熒光標記類型詳見表2,并委托北京睿博興科生物技術有限公司合成。
1. 4 PCR擴增及產物檢測
PCR反應體系15.0 μL:DNA模板(50 ng/μL)1.0 μL,Premix TaqTM Mix(TaKaRa)7.5 μL,上、下游引物(0.5 μmol/L)各0.5 μL,雙蒸水5.5 μL。擴增程序:95 ℃預變性3 min;95 ℃ 30 s,50 ℃ 30 s,72 ℃ 20 s,進行32個循環;72 ℃延伸6 min。以1.0%瓊脂糖凝膠電泳檢測PCR擴增產物,并委托北京睿博興科生物技術有限公司,根據產物熒光特性進行毛細管電泳混檢,測序結果使用Gene Marker 2.2.0讀取。
1. 5 數據分析
根據各凡納濱對蝦種苗群體在各微衛星位點的基因型,利用GENE Stock 1.32進行統計分析(Yeh and Boyle,1997),計算各微衛星位點的等位基因數(Na)、有效等位基因數(Ne)、觀測雜合度(Ho)、期望雜合度(He)、Shannon’s信息指數(I)及Nei’s遺傳距離;采用PIC_CALC 0.6(https://github.com/luansheng/PIC_CALC)計算多態信息含量(PIC);運用Arlequin 3.1計算群體間遺傳分化系數(Fst)及進行分子變異分析(AMOVA) (Excoffier et al.,2005);同時基于Nei’s遺傳距離,通過MEGA 5.0以UPGMA法進行聚類分析(Tamura et al.,2011)。
2 結果與分析
2. 1 微衛星位點多態性分析結果
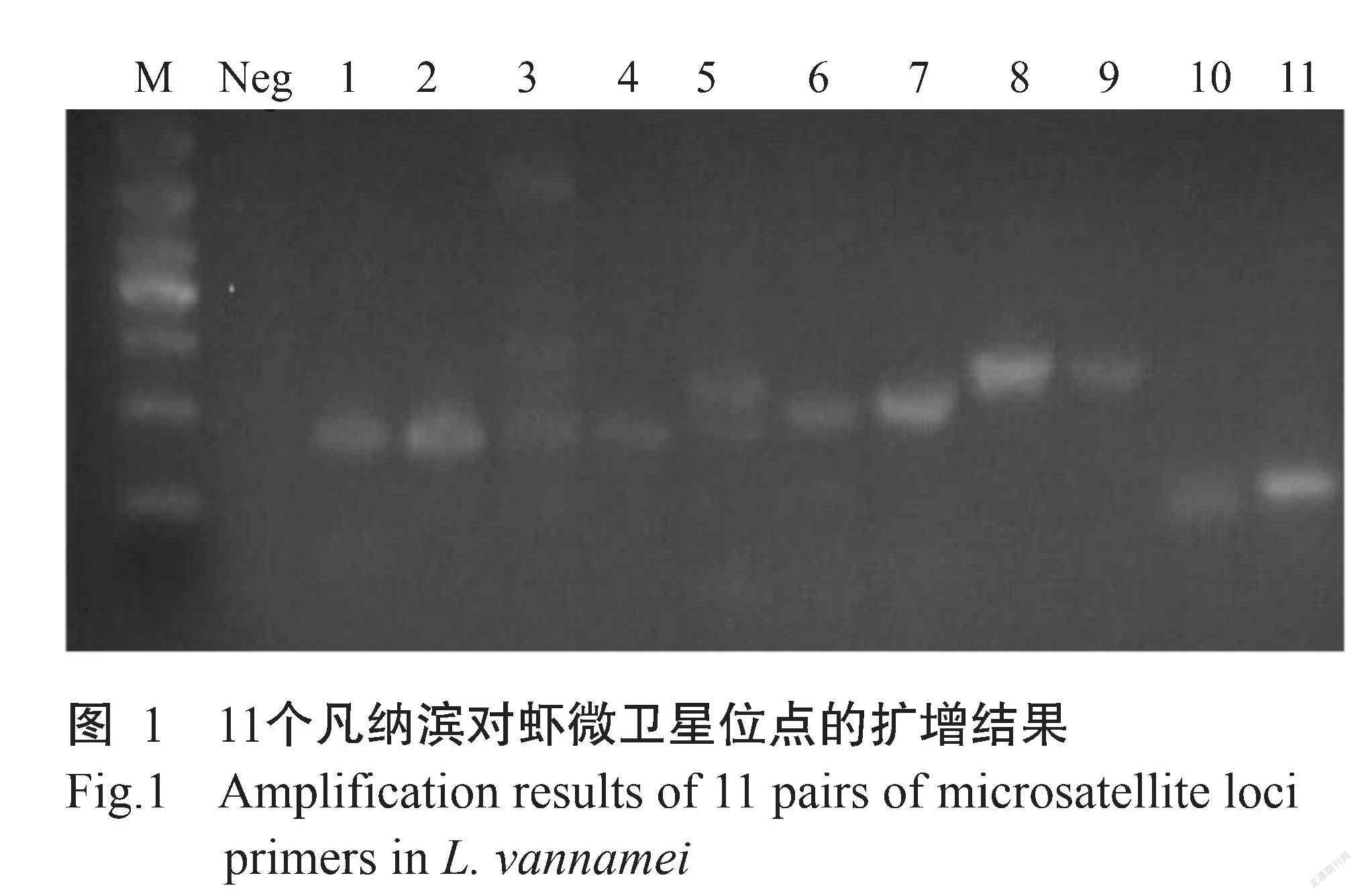
利用11對微衛星引物對9個凡納濱對蝦種苗群體共計258個個體的基因組進行擴增,結果(圖1和圖2)顯示,11個微衛星位點均能進行有效擴增,其Na、Ne、Ho、He、I及PIC等信息見表3。各微衛星位點的Na為5~39,Ne為1.6582~14.5320,Ho為0.2023~0.6357,He為0.3969~0.9312,I為0.8357~3.0296,PIC為0.3789~0.9274。其中,TUMXLv8.256、TUMXLv10.312和TUYFLvL16.1a位點的PIC分別為0.3821、0.4605和0.3789,而其余8個微衛星位點的PIC均大于0.6000。
2. 2 凡納濱對蝦種苗群體遺傳多樣性分析結果
9個凡納濱對蝦種苗群體的Na、Ne、Ho、He、I和PIC平均值分別在5.5455~8.8182、3.2795~5.4735、0.3769~0.5182、0.6189~0.6948、1.2823~1.6092和0.5863~ 0.6646(表4)。其中,Stock 3群體的Na、Ne和I平均值最高,Stock 2群體的He和PIC平均值最高,而Stock 1群體的Na、Ne、Ho、PIC和I平均值均最低。在9個凡納濱對蝦種苗群體中,He平均值以Stock 6群體最低,而Ho平均值以Stock 7群體最高。可見,各凡納濱對蝦種苗群體均具有較高的遺傳多樣性,遺傳多樣性水平排序為Stock 3>Stock 2>Stock 4>Stock 5>Stock 7>Stock 6>Stock 9>Stock 8>Stock 1。
2. 3 凡納濱對蝦種苗群體遺傳分化及遺傳距離分析結果
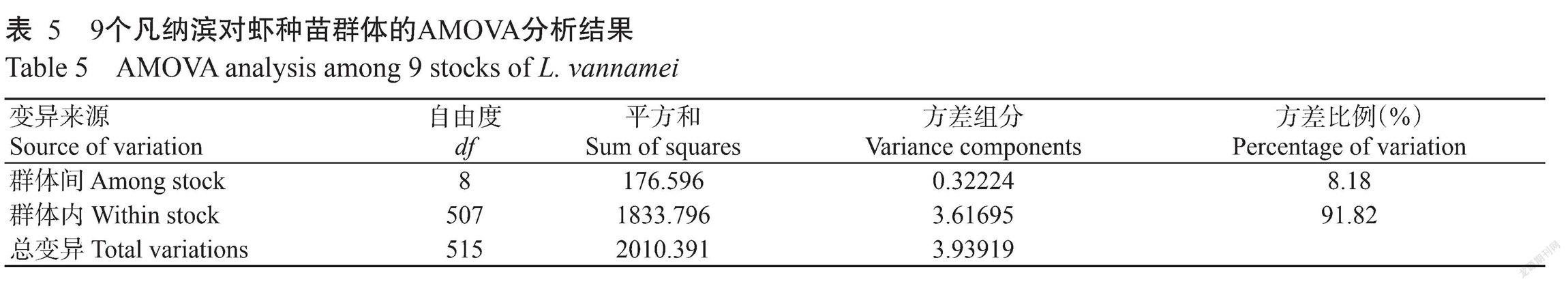
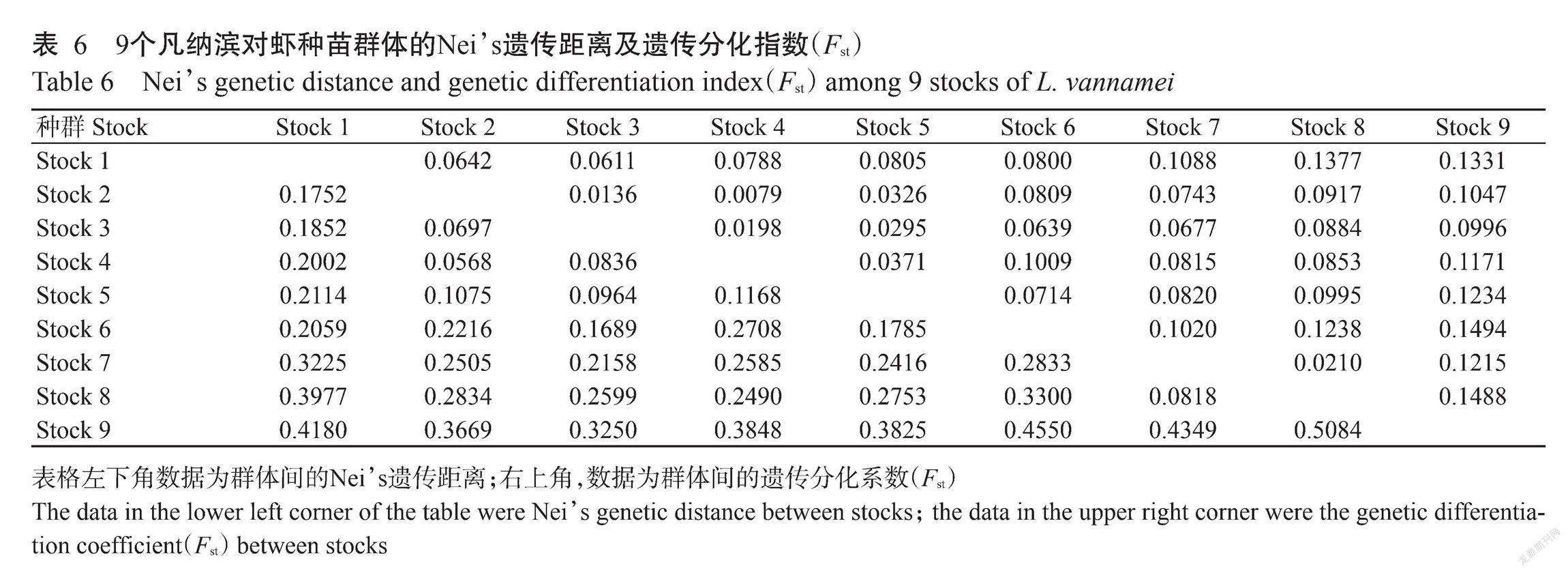
9個凡納濱對蝦種苗群體的AMOVA分析結果如表5所示,有91.82%的變異來自于群體內,僅有8.18%的變異來自于群體間。兩兩群體間的Fst范圍在0.0079~0.1494(表6),即整體遺傳分化表現為中低水平。其中,Stock 6群體與Stock 9群體間的Fst最高(0.1494),而Stock 2群體與Stock 4群體間的Fst最低(0.0079)。Stock 2、Stock 3、Stock 4和Stock 5等4個群體間及Stock 7群體與Stock 8群體間的Fst均小于0.05(0.0079~0.0371),即群體間無遺傳分化,而其他群體間表現為輕度遺傳分化(0.05<Fst <0.15)。
9個凡納濱對蝦種苗群體間的Nei’s遺傳距離為0.0568~0.5084(表6),以Stock 2群體與Stock 4群體的Nei’s遺傳距離最近,而Stock 8群體與Stock 9群體的Nei’s遺傳距離最遠。根據Nei’s遺傳距離構建的UPGMA聚類圖(圖3)顯示,9個凡納濱對蝦種苗群體大致可劃分為三大分支,第一分支包括Stock 1~Stock 6群體,其中Stock 2群體和Stock 4群體先聚為一簇,然后依次同Stock 3、Stock 5、Stock 1和Stock 6等4個群體聚為一支;第二分支由Stock 7群體和Stock 8群體組成,而Stock 9群體獨立為第三分支。
3 討論
微衛星分子標記是物種群體遺傳多樣性分析的重要手段,而基因型分析是微衛星分子標記研究的關鍵環節。本研究采用基于毛細管電泳的熒光標記引物進行基因分型,選取11個具有多態性的微衛星位點,針對不同批次的凡納濱對蝦進口親蝦一代苗群體開展遺傳多樣性分析,結果顯示各微衛星位點的PIC為0.3789~0.9274。PIC能反映等位基因的多態性水平,PIC在0.25~0.50為中度多態,在0.50之上則為高度多態(Shete et al.,2000;Nagy et al.,2012)。說明本研究選取的11個微衛星分子標記均呈中高度多態,是評估群體遺傳多樣性的有效分子標記。
遺傳多樣性是種質資源評價的重要依據,Na、Ho、He和PIC等均是衡量群體遺傳多樣性的重要指標。Zhang等(2014)選用7個微衛星分子標記對由新加坡和美國引進的7個親蝦群體遺傳多樣性進行分析,結果顯示,各群體的Na平均值為4.286~11.143,Ne平均值為3.090~6.407,Ho平均值為0.526~0.754,He平均值為0.600~0.802。本研究中,由引進親蝦直接繁育的9個子一代苗群體的Na、Ne和He平均值均在以上相應范圍內,僅Ho平均值小于上述指標,推測是由于各凡納濱對蝦群體幼體來源相同,其遺傳多樣性差異相對較小。包秀鳳(2014)針對親蝦源于正大集團的養殖種苗群體開展微衛星分析,發現受試群體的Na、Ne、He和PIC平均值分別為3.571、2.215、0.509和0.448,各項指標均低于本研究結果。此外,于2018年5月在正大神灣蝦苗場采集的4個批次種苗群體(Stock 2、Stock 3、Stock 4和Stock 5)的I分別為1.5908、1.6092、1.4919和1.4874,其遺傳多樣性相近,且均高于其余5個群體(Stock 1、Stock 6、Stock 7、Stock 8和Stock 9),故推測遺傳多樣性水平能反映后續對蝦養殖生產性能的差異。
Fst是衡量群體間遺傳分化程度的重要參數(黃小芳等,2020;彭敏等,2020)。Fst為0~0.05時表示群體間無遺傳分化,Fst為0.05~0.15時表示群體間存在輕度遺傳分化,Fst為0.15~0.25表示群體間存在中度遺傳分化,Fst大于0.25時表示群體間存在高度遺傳分化。謝麗等(2009)研究指出Molokai、OI、SIS和Kona Bay等4個養殖凡納濱對蝦群體間的遺傳分化占15.83%;包秀鳳(2014)對國內外7個選育凡納濱對蝦群體的遺傳分析結果表明,群體間的Fst平均值為0.2155;Zhang等(2014)研究發現7個國外引進凡納濱對蝦群體間的遺傳分化水平為11.30%。在本研究中,9個凡納濱對蝦種苗群體間的Fst平均值為0.0818,即8.18%的變異來自于群體間,表現為中低水平的遺傳分化;但兩兩群體間的遺傳分化水平存在明顯差異,Stock 2、Stock 3、Stock 4和Stock 5等4個群體間及Stock 7群體與Stock 8群體間的Fst均小于0.05(0.0079~0.0371),即群體間無遺傳分化,而其他群體間表現為輕度遺傳分化,與各群體間的Nei’s遺傳距離基本對應。一般情況下,群體間的遺傳距離越小,其親緣關系越近。基于Nei’s遺傳距離的聚類分析結果也表明,2018年5月采集自正大神灣蝦苗場的4個批次群體(Stock 2、Stock 3、Stock 4和Stock 5)間及2017年8月采集自海尚種苗培育基地的2個批次群體(Stock 7和Stock 8)間的親緣關系較接近,而較早采集自正大神灣蝦苗場的Stock 1群體與Stock 2~Stock 5群體的親緣關系相對較遠,其遺傳分化達中等水平。
本研究利用微衛星分子標記分析不同批次進口親蝦子一代種苗的遺傳變異特征,結果發現不同群體間存在遺傳多樣性差異,且表現出明顯的遺傳分化;由同一種苗培育(基地)企業提供的蝦苗在短期內各群體間的遺傳分化保持在較低水平,但隨著時間的推移可能會發生較大變化;在同一時期由不同種苗培育(基地)企業培育獲得的蝦苗即使其親蝦來源相同,也會存在明顯的遺傳分化。這可能與近年來進口親蝦子一代種苗在生長性能和養殖成活率等方面的性能變化有關聯。因此,有必要進一步系統分析凡納濱對蝦進口親蝦子一代種苗的遺傳變異狀況,并通過與之對應的養殖實踐以獲得不同群體的養殖生產性能信息,建立各群體遺傳特征與養殖生產性能的關聯數據庫,為今后我國養殖對蝦優質種苗的大數據建設及其推廣應用提供基礎數據支撐。
4 結論
不同批次凡納濱對蝦進口親蝦子一代種苗群體的遺傳特征存在明顯差異,各群體間的遺傳距離隨著采樣時間和地點的改變而發生明顯變化,且這些遺傳背景差異可能會造成不同群體在養殖生產性能方面產生差異。
參考文獻:
包秀鳳. 2014. 凡納濱對蝦選育群體遺傳多樣性分析[D]. 湛江:廣東海洋大學. [Bao X F. 2014. Analysis of genetic diversity of selected stocks of Litopenaeus vannamei[D]. Zhanjiang:Guangdong Ocean University.] doi:10.7666/d.D522247.
代平,孔杰,欒生. 2018. 我國凡納濱對蝦種質資源引進與分析[J]. 科學養魚,(1):3-5. [Dai P,Kong J,Luan S. 2018. Introduction and analysis of germplasm resources of Litopenaeus vannamei in China[J]. Scientific Fish Farming,(1):3-5.] doi:10.14184/j.cnki.issn1004-843x.2018.01. 002.
黃小芳,唐章生,劉俊丹,張宏燕,鐘一治,盧智發,侯樹鑒,王大鵬,陸專靈. 2020. 廣西不同地區克氏原螯蝦群體遺傳多樣性微衛星分析[J]. 南方農業學報,51(2):437-444. [Huang X F,Tang Z S,Liu J D,Zhang H Y,Zhong Y Z,Lu Z F,Hou S J,Wang D P,Lu Z L. 2020. Genetic diversity microsatellite analysis of Procambarus clarkii po-pulations in different regions of Guangxi[J]. Journal of Southern Agriculture,51(2):437-444.] doi:10.3969/j.issn. 2095-1191.2020.02.025.
李強勇,李旻,曾地剛,朱威霖,彭敏,楊春玲,劉青云,趙永貞,陳秀荔,陳曉漢. 2020. 凡納濱對蝦微衛星分子標記的開發及不同養殖家系遺傳多態性分析[J]. 南方農業學報,51(2):429-436. [Li Q Y,Li M,Zeng D G,Zhu W L,Peng M,Yang C L,Liu Q Y,Zhao Y Z,Chen X L,Chen X H. 2020. Development of microsatellite markers of Litopenaeus vannamei and genetic polymorphism analy-sis of different cultured families[J]. Journal of Southern Agriculture,51(2):429-436.] doi:10.3969/j.issn.2095-1191. 2020.02.024.
劉連為,陳新軍,許強華,李偉文. 2014. 北太平洋柔魚微衛星標記的篩選及遺傳多樣性[J]. 生態學報,34(23):6848-6854. [Liu L W,Chen X J,Xu Q H,Li W W. 2014. Isolation and genetic diversity of microsatellite DNA of Ommastrephes bartramii in the North Pacific Ocean[J]. Acta Ecologica Sinica,34(23):6848-6854.] doi:10.5846/stxb 201303030334.
毛守康,馬愛軍,丁福紅,閆喜武,羅海忠,李偉業,徐寶榮,王懷忠,王寶義,王廣寧. 2016. 梭魚(Liza haematocheila)4個野生地理群體遺傳多樣性的微衛星分析[J]. 漁業科學進展,37(2):68-75. [Mao S K,Ma A J,Ding F H,Yan X W,Luo H Z,Li W Y,Xu B R,Wang H Z,Wang B Y,Wang G Y. 2016. Analysis of genetic structures of four wild geographic populations of mullet Liza haematocheila by using microsatellite marker technique[J]. Progress in Fishery Sciences,37(2):68-75.] doi:10.11758/yykxjz.20150407001.
彭敏,陳慧芳,李強勇,楊春玲,曾地剛,劉青云,趙永貞,陳曉漢,林勇,陳秀荔. 2020. 凡納濱對蝦連續 3 個世代選育群體的遺傳多樣性分析[J]. 南方農業學報,51(6):1442-1450. [Peng M,Chen H F,Li Q Y,Yang C L,Zeng D G,Liu Q Y,Zhao Y Z,Chen X H,Lin Y,Chen X L. 2020. Genetic diversity of three consecutive generations of Litopenaeus vannamei[J]. Journal of Southern Agriculture,51(6):1442-1450.] doi:10.3969/j.issn.2095-1191. 2020.06.026.
孫效文,張曉鋒,趙瑩瑩,張研,賈智英,常玉梅,魯翠云,梁利群. 2008. 水產生物微衛星標記技術研究進展及其應用[J]. 中國水產科學,15(4):689-703. [Sun X W,Zhang X F,Zhao Y Y,Zhang Y,Jia Z Y,Chang Y M,Lu C Y,Liang L Q. 2008. Development and application of microsatellite markers in aquatic species[J]. Journal of Fishery Sciences of China,15(4):689-703.] doi:10.3321/j.issn:1005-8737.2008.04.022.
童馨,龔世圓,喻達輝,杜博,黃桂菊,李莉好,郭奕惠,李色東. 2007. 凡納濱對蝦不同世代生長性狀的變異[J]. 南方水產科學,3(6):30-33. [Tong X,Gong S Y,Yu D H,Du B,Huang G J,Li L H,Guo Y H,Li S D. 2007. Variation of growth traits at different generations of Pacific white shrimp(Litopenaeus vannamei)[J]. South China Fisheries Science,3(6):30-33.] doi:10.3969/j.issn.2095-0780.2007.06.005.
童馨,龔世圓,喻達輝,黃桂菊,杜博,李色東. 2009. 凡納濱對蝦(Litopenaeus vannamei)不同世代養殖群體的遺傳多樣性分析[J]. 海洋與湖沼,40(2):214-220. [Tong X,Gong S Y,Yu D H,Huang G J,Du B,Li S D. 2009. Genetic diversity of cultured Pacific white shrimp(Litopenaeus vannamei) stocks of different generations in China[J]. Oceanologia et Limnologia Sinica,40(2):214-220.] doi:10.3321/j.issn:0029-814X.2009.02.017.
王麗花,曹煜成,李卓佳. 2012. 溶藻細菌控藻作用及其在對蝦養殖池塘中的應用前景[J]. 南方水產科學,8(4):76-82. [Wang L H,Cao Y C,Li Z J. 2012. Algae-constrai-ning effect of algae-lysing bacteria and its application prospect in shrimp ponds[J]. South China Fisheries Science,8(4):76-82.] doi:10.3969/j.issn.2095-0780.2012.04.012.
王興強,馬甡,董雙林. 2004. 凡納濱對蝦生物學及養殖生態學研究進展[J]. 海洋湖沼通報,(4):94-100. [Wang X Q,Ma S,Dong S L. 2004. Studies on the biology and cultural ecology of Litopenaeus vannamei:A review[J]. Transactions of Oceanology and Limnology,(4):94-100.] doi:10.3969/j.issn.1003-6482.2004.04.016.
文國樑,曹煜成,徐煜,胡曉娟,徐武杰,李卓佳. 2015. 養殖對蝦肝胰腺壞死綜合癥研究進展[J]. 廣東農業科學,42(11):118-123. [Wen G L,Cao Y C,Xu Y,Hu X J,Xu W J,Li Z J. 2015. Review on hepatopancreas necrosis syndrome of shrimp[J]. Guangdong Agricultural Sciences,42(11):118-123.] doi:10.3969/j.issn.1004-874X.2015.11. 022.
吳怡迪,駱軒,楊章武,黃永春,游偉偉. 2016. 凡納濱對蝦選育系間雜交的生長性狀及遺傳多樣性分析[J]. 廈門大學學報(自然科學版),55(5):646-653. [Wu Y D,Luo X,Yang Z W,Huang Y C,You W W. 2016. Growth performance and genetic diversity analysis for hybrids between selective lines of Litopenaeus vannamei[J]. Journal of Xiamen University (Natural Science),55(5):646-653.] doi:10.6043/j.issn.0438-0479.201511010.
謝麗,陳國良,葉富良,栗志民. 2009. 凡納濱對蝦4個選育群體遺傳多樣性的SSR分析[J]. 廣東海洋大學學報,29(4):5-9. [Xie L,Chen G L,Ye F L,Li Z M. 2009. Genetic diversity of four selected stocks of Litopenaeus vannamei as revealed by SSR marker[J]. Journal of Guangdong Ocean University,29(4):5-9.] doi:10.3969/j.issn. 1673-9159.2009.04.002.
趙志英,梁麗運,白麗蓉. 2018. 斑節對蝦3個野生群體遺傳多樣性的微衛星標記分析[J]. 熱帶海洋學報,37(3):65-72. [Zhao Z Y,Liang L Y,Bai L R. 2018. Analysis of genetic diversity among three wild populations of Penaeus monodon using microsatellite marker[J]. Journal of Tropical Oceanography,37(3):65-72.] doi:10.11978/2017084.
周大顏,張志新,黃彩林,招志杰,莫飛龍. 2019. 3種白鯽雜交子代的轉錄組學分析[J]. 南方農業學報,50(6):1328-1338. [Zhou D Y,Zhang Z X,Huang C L,Zhao Z J,Mo F L. 2019. Transcriptomic analysis of three Carassius auratus cuvieri hybrids[J]. Journal of Southern Agriculture,50(6):1328-1338.] doi:10.3969/j.issn.2095-1191.2019. 06.24.
Alcivar-Warren A,Meehan-Meola D,Park S W,Xu Z K,Dela-ney M,Zuniga G. 2007a. ShrimpMap:A low-density,microsatellite-based linkage map of the pacific white leg shrimp,Litopenaeus vannamei:Identification of sex-linked markers in linkage group 4[J]. Journal of Shellfish Research,26(4):1259-1277. doi:10.2983/0730-8000(2007)26[1259:SALMLM]2.0.CO;2.
Alcivar-Warren A,Song L S,Meehan-Meola D,Xu Z K,Xiang J H,Warren W. 2007b. Characterization and mapping of expressed sequence tags isolated from a subtrac-ted cDNA library of Litopenaeus vannamei injected with white spot syndrome virus[J]. Journal of Shellfish Resear-ch,26(4):1247-1258. doi:10.2983/0730-8000(2007)26[1247:CAMOES]2.0.CO;2.
Cruz P,Mejia-Ruiz C H,Perez-Enriquez R,Ibarra A M. 2002. Isolation and characterization of microsatellites in Pacific white shrimp Penaeus(Litopenaeus) vannamei[J]. Molecular Ecology Notes,2(3):239-241. doi:10.1046/j.1471- 8286.2002.00211.x-i2.
Excoffier L,Laval G,Schneider S. 2005. Arlequin (version 3.0):An integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics,1:47-50. doi:10.1177/117693430500100003.
Garcia D K,Alcivar-Warren A. 2007. Characterization of 35 new microsatellite genetic markers for the pacific whiteleg shrimp,Litopenaeus vannamei:Their usefulness for studying genetic diversity of wild and cultured stocks,tracing pedigree in breeding programs,and linkage mapping[J]. Journal of Shellfish Research,26(4):1203-1216. doi:10.2983/0730-8000(2007)26[1203:CONMGM]2.0.CO;2.
Garcia D K,Dhar A K,Alcivar-Warren A. 1996. Molecular analysis of a RAPD marker(B20) reveals two microsatellites and differential mRNA expression in Penaeus vannamei[J]. Molecular Marine Biology and Biotechnology,5(1):71-83.
Jia Z Y,Sun X W,Liang L Q,Li D Y,Lei Q Q. 2006. Isolation and characterization of microsatellite markers from Pacific white shrimp(Litopenaeus vannamei)[J]. Molecular Ecology Notes,6(4):1282-1284. doi:10.1111/j.1471-8286.2006.01515.x.
Meehan D,Xu Z K,Zuniga G,Alcivar-Warren A. 2003. High frequency and large number of polymorphic microsatellites in cultured shrimp,Penaeus(Litopenaeus) vannamei [Crustacea:Decapoda][J]. Marine Biotechnology,5:311-330. doi:10.1007/s10126-002-0092-z.
Nagy S,Poczai P,Cernák I,Gorji A M,Heged?s G,Taller J. 2012. PICcalc:An online program to calculate polymorphic information content for molecular genetic studies[J]. Biochemical Genetics,50:670-672. doi:10.1007/s10528-012-9509-1.
Qiu L,Chen M M,Wan X Y,Li C,Zhang Q L,Wang R Y,Cheng D Y,Dong X,Yang B,Wang X H,Xiang J H,Huang J. 2017. Characterization of a new member of Iridoviridae,Shrimp hemocyte iridescent virus(SHIV),found in white leg shrimp(Litopenaeus vannamei)[J]. Scienti-fic Reports,7:11834. doi:10.1038/s41598-017-10738-8.
Shete S,Tiwari H,Elston R C. 2000. On estimating the he-terozygosity and polymorphism information content value[J]. Theoretical Population Biology,57(3):265-271. doi:10.1006/tpbi.2000.1452.
Tamura K,Peterson D,Peterson N,Stecher G,Nei M,Kumar S. 2011. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,28(10):2731-2739. doi:10.1093/molbev/msr121.
Wang M S,Wang W J,Xiao G X,Liu K F,Hu Y L,Tian T,Kong J,Jin X S. 2016. Genetic diversity analysis of spaw-ner and recaptured populations of Chinese shrimp(Fenneropenaeus chinensis) during stock enhancement in the Bohai Bay based on an SSR marker[J]. Acta Oceanologica Sinica,35(8):51-56. doi:10.1007/s13131-016-0830-0.
Yeh F C,Boyle T J B. 1997. Population genetic analysis of co-dominant and dominant markers and quantitative traits[J]. Belgian Journal of Botany,129(2):157.
Zhang K,Wang W J,Li W Y,Zhang Q Q,Kong J. 2014. Analysis of genetic diversity and differentiation of seven stocks of Litopenaeus vannamei using microsatellite mar-kers[J]. Journal of Ocean University of China,13(4):647-656. doi:10.1007/s11802-014-2208-2.
收稿日期:2020-05-26
基金項目:中國水產科學研究院基本科研業務費專項(2020TD54);國家現代農業產業技術體系建設專項(CARS-48);廣東省現代農業產業技術體系創新團隊建設專項(2019KJ149)
通訊作者:曹煜成(1979-),https://orcid.org/0000-0002-0995-1131,博士,研究員,主要從事水產健康養殖及養殖生態環境調控與修復研究工作,E-mail:cyc_715@163.com;楊鏗(1975-),https://orcid.org/0000-0002-6970-0668,副研究員,主要從事水產健康養殖技術及產品推廣應用研究工作,E-mail:yangkeng66@163.com
第一作者:徐煜(1987-),https://orcid.org/0000-0003-2364-6659,主要從事對蝦分子生理及健康養殖研究工作,E-mail:xuyublq@163.com
1443501186395

