基因組印記的研究進(jìn)展
蘇魯方 李振庭 鄭瑜 包純 劉小云

摘要 基因組印記(genomicimprinting),又稱遺傳印記(geneticimprinting),是基因表達(dá)的一種調(diào)控機(jī)制,屬于表觀遺傳學(xué),包括DNA甲基化修飾和組蛋白甲基化修飾。闡述了印記基因的基本特征(可逆性、成簇分布、時(shí)空性、組織特異性和保守性),其可能的形成機(jī)制,為研究動(dòng)、植物遺傳育種奠定了分子基礎(chǔ)。
關(guān)鍵詞 基因組印記;印記基因;甲基化;H3K27me3
中圖分類號(hào) Q.75文獻(xiàn)標(biāo)識(shí)碼A文章編號(hào)0517-6611(2021)07-0008-04
AbstractGenomicimprintingalsoknownasgeneticimprintingisaregulatorymechanismofgeneexpressionwhichbelongstoepigenetics,includingDNAmethylationandhistonemethylationmodification.Thispaperillustratedthebasiccharacteristics(reversibility,clustereddistribution,temporalandspatial,tissuespecificityandconservativeproperty)ofimprintedgenesanditspossibleformativemechanism,inordertostudymoleculargeneticfoundationinanimalsandplants.
KeywordsGenomicimprinting;Imprintedgene;Methylation;H3K27me3
基因組印記(genomicimprinting)指子代中來自親本的特定基因或染色體在發(fā)育過程中產(chǎn)生特異性的加工修飾導(dǎo)致父源或母源一方等位基因表達(dá),另一方等位基因沉默或表達(dá)量很少的可遺傳表觀修飾現(xiàn)象。其中,母本等位基因表達(dá),而父本等位基因不表達(dá)的基因稱為母源表達(dá)基因或父源印記(paternalimprinting);父本等位基因表達(dá)而母本等位基因不表達(dá)的基因則稱為父源表達(dá)基因或母源印記(maternalimprinting)[1-2]。因此印記基因具有親源特異性。近年來,研究調(diào)節(jié)印記基因的可能主要機(jī)制包括:DNA甲基化(DNAmethylation)修飾、組蛋白甲基化修飾(histonemethylationmodification)、染色體重塑修飾(chromatinremodelingmodification)和長(zhǎng)非編碼RNA:lncRNA(longnoncodingRNA)等,這些修飾模式易受環(huán)境因素的影響。基因組印記主要發(fā)生在動(dòng)物胎盤、胚胎和開花植物的胚乳中,任何一種調(diào)節(jié)方式發(fā)生改變都可能導(dǎo)致基因功能的變化。因此,基因組印記對(duì)動(dòng)、植物正常生長(zhǎng)發(fā)育和分化等生命活動(dòng)有重要的作用。
1印記基因概念的提出
基因印記概念首次提出于1960年,Crouse[3]在研究Sciara昆蟲試驗(yàn)中發(fā)現(xiàn)后代2條X染色體中僅來自母方的X染色體具有活性,父方的X染色體處于靜止?fàn)顟B(tài),這說明一些等位基因的表達(dá)出現(xiàn)差異性,具有親本選擇性。1970年,Kermicle[4]研究報(bào)道玉米植株存在能夠影響后代籽粒胚乳顏色產(chǎn)生顯著差異的印記基因(R基因),首次提出了印記基因可能存在于植物。1984年,Mcgrath等[5]利用核移植技術(shù)發(fā)現(xiàn)小鼠受精發(fā)育過程中,雄原核替代雌原核即兩原核同時(shí)來自父方或雌原核替代雄原核即兩原核來自母方,發(fā)育主要形成胚胎組織或胎盤組織,以致發(fā)育后期將會(huì)死亡,證明了親本等位基因在功能上具有不同等性,同樣首次提出了哺乳動(dòng)物也可能存在基因印記現(xiàn)象。1991年,Dechiara等[6]通過基因敲除技術(shù)證實(shí)了小鼠的第一個(gè)內(nèi)源性印記基因?yàn)橐葝u素生長(zhǎng)因子Ⅱ(insulinlikegrowthfactor2,IGF2),屬于母源印記基因。研究發(fā)現(xiàn)若敲除的等位基因來自父本,后代表現(xiàn)為動(dòng)物體型較小,若敲除來自母本的等位基因,后代表現(xiàn)正常。1991年,Bartolomei等[7-8]進(jìn)一步證實(shí)胰島素生長(zhǎng)因子Ⅱ受體(insulinlikegrowthfactortype-2receptor,IGF2R)和長(zhǎng)鏈非編碼RNA(LncRNA)H19基因在小鼠中為父源印記基因。隨后,Rainier等[9]首次發(fā)現(xiàn)人類存在的印記基因:IGF2和H19。目前,哺乳動(dòng)物、植物中已分別有約150、16個(gè)印記基因被鑒定[10-11]。與哺乳動(dòng)物相比,針對(duì)植物印記基因的研究不多,擬南芥中已鑒定出10個(gè)印記基因,其中具有印記現(xiàn)象的FIS(fertilisation-independentseed)基因是在研究一系列胚乳突變體中最早發(fā)現(xiàn)的,它們具有抑制中央細(xì)胞分裂和調(diào)節(jié)早期胚乳發(fā)育的功能,包括MEDEA(MEA)/FIS1、FIS2和FIS3/FIE等,其中MEA和FIS2為母源印記基因[11-13]。fis突變體有2種表型,一是在未授精情況下,中央細(xì)胞自主發(fā)育為二倍體胚乳;二是授精條件下,形成非細(xì)胞化胚乳,均導(dǎo)致胚乳發(fā)育異常[12-14]。這些基因中的部分不僅自身具有印記基因的表達(dá)特征,還可通過修飾組蛋白抑制基因表達(dá)的方式來調(diào)節(jié)其他印記基因[15]。
20世紀(jì)90年代,人類對(duì)哺乳動(dòng)物基因組印記的分子研究進(jìn)入了活躍時(shí)期。之后,隨著科技進(jìn)步和科研經(jīng)費(fèi)的投入,科學(xué)家們發(fā)現(xiàn)牛、豬、羊等家畜,有袋類動(dòng)物[16]和種子植物[17]存在印記基因,并預(yù)測(cè)有500~1000個(gè),已經(jīng)證實(shí)的有160個(gè)左右[18-20],但兩棲類、爬行類、魚類和鳥類中尚未發(fā)現(xiàn)基因印記現(xiàn)象的存在[21]。
2印記基因的特征
2.1可逆性
哺乳動(dòng)物基因組印記一般經(jīng)歷3個(gè)過程:印記的建立、維持、去除[22]。生殖細(xì)胞形成早期,來自親本雙方的印記全部去除;親本等位基因在生殖細(xì)胞形成精子或卵子時(shí)產(chǎn)生新的印記;精子和卵子受精后的個(gè)體發(fā)育過程維持印記[2]。大多數(shù)印記基因在印記消除和建立的過程中,伴隨著明顯的DNA甲基化消除和形成,認(rèn)為DNA甲基化可能是基因組印記建立的主要機(jī)制之一[23]。最近另一印記調(diào)控機(jī)制通過親本等位基因差異性組蛋白修飾,影響親本等位基因表達(dá)不同,發(fā)現(xiàn)由于母本等位基因組蛋白H3第27個(gè)賴氨酸發(fā)生三甲基化(H3K27me3)的修飾,父本等位基因未修飾,因而母本等位基因表達(dá)受抑制,父本等位基因得到表達(dá)[24]。哺乳動(dòng)物原始生殖細(xì)胞到成熟雌雄生殖細(xì)胞完成了染色體組蛋白甲基化或DNA甲基化的除去和重編程過程,類似于哺乳動(dòng)物印記基因組蛋白甲基化或DNA甲基化的除去和重編程過程。因此,可以認(rèn)為哺乳動(dòng)物原始生殖細(xì)胞在胚胎發(fā)育早期的過程中,雌雄生殖細(xì)胞首先完成基因組組蛋白或DNA的除修飾,再重新對(duì)基因組進(jìn)行組蛋白修飾或DNA修飾,直到產(chǎn)生穩(wěn)定修飾狀態(tài)的精原細(xì)胞和卵原細(xì)胞[24]。因此,基因組的印記在雌雄生殖細(xì)胞發(fā)育期通過印記的消除和建立,形成穩(wěn)定、遺傳且可具有發(fā)育潛能的細(xì)胞(圖1)。
植物與動(dòng)物基因組印記過程類似,研究發(fā)現(xiàn)擬南芥存在與哺乳動(dòng)物相似功能作用的DNA甲基化轉(zhuǎn)移酶(Methyltransferase1,AtMET1)、多梳抑制蛋白復(fù)合體2(polycombrepressivecomplex2,PRC2),分別主要負(fù)責(zé)維持CpG位點(diǎn)胞嘧啶的甲基化、H3K27三甲基化/H3K9二甲基化/CHG位點(diǎn)胞嘧啶的甲基化[25-26]。目前,植物基因組印記現(xiàn)象只存在被子植物三倍體胚乳中,擬南芥胚乳整個(gè)基因組是去甲基化的,并伴隨著廣泛的小干擾RNA在非CpG位點(diǎn)的高甲基化[27]。DNA糖基化酶(demeter,DME)是完成胚乳印記基因去甲基化的一種酶,只在雌配體中央細(xì)胞中表達(dá),雄配體中不表達(dá)[28]。可以推測(cè),雌雄配體在中央細(xì)胞形成前在AtMET1控制下為基因組甲基化狀態(tài),使這些印記基因轉(zhuǎn)錄沉默。擬南芥雌性配體的中央細(xì)胞和胚乳特異性存在另一母系表達(dá)抑制蛋白FIS-PRC2,使母本基因染色體附近發(fā)生H3K27me3修飾,同時(shí)也存在H3K9me2修飾,而且有CHG位點(diǎn)發(fā)生胞嘧啶甲基化的現(xiàn)象,從而母本基因表達(dá)沉默,父本基因表達(dá)[26]。因此,認(rèn)為植物雌雄配子基因組在發(fā)育過程中出現(xiàn)一個(gè)母源特異性機(jī)制,使胚囊中央細(xì)胞中默認(rèn)的沉默狀態(tài)基因激活或抑制某個(gè)基因的表達(dá),母本等位基因得到表達(dá)或抑制,而父源等位基因缺乏這一機(jī)制,相應(yīng)地選擇沉默或表達(dá)。這也與哺乳動(dòng)物形成雌雄配子時(shí)的基因組甲基化模式相一致,而植物在形成雌雄配子前基因組甲基化/去甲基化方式仍不清楚。
2.2成簇分布
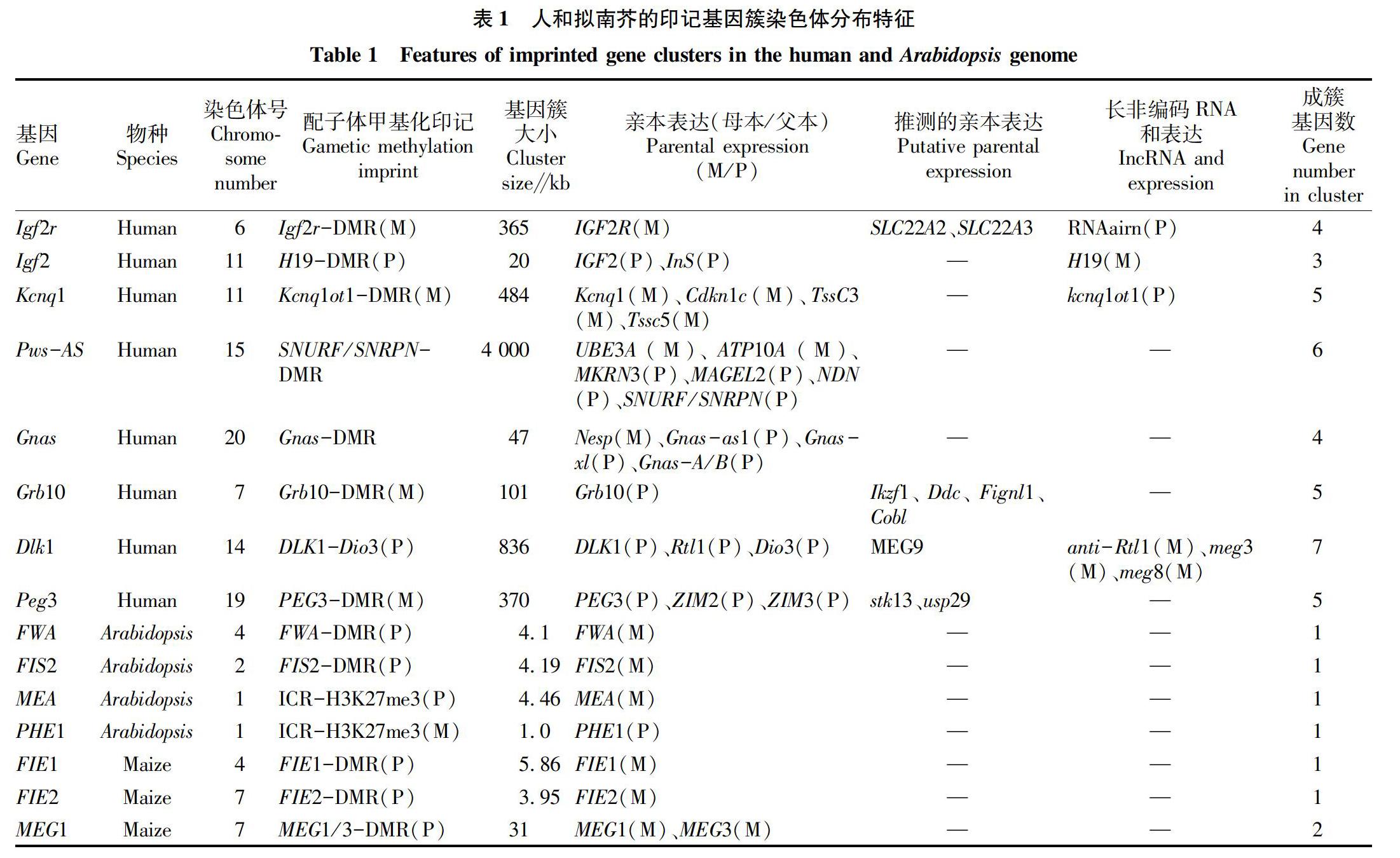
大多數(shù)印記基因的顯著特征是在基因組中成簇分布,并構(gòu)成印記域。一般情況下,父系印記基因和母系印記基因交替位于印記簇區(qū)域,它們翻譯為蛋白質(zhì)和轉(zhuǎn)錄為一些非編碼RNA。人類和小鼠中已經(jīng)發(fā)現(xiàn)100~200個(gè)印記基因,分別有70多個(gè)印記基因已證實(shí),且約80%印記基因成簇存在于人和鼠基因組[29-30]。在小鼠2號(hào)、7號(hào)、9號(hào)、11號(hào)和17號(hào)染色體中發(fā)現(xiàn)較大的印記基因簇,這些印記基因簇包括IGF2、IGF2R、PWS、PEG3,DLK1、Grb10、Gna和Kcnq1印記域[31]。在人類7號(hào)、11號(hào)、14號(hào)、15號(hào)、17號(hào)、19號(hào)和20號(hào)染色體中發(fā)現(xiàn)7個(gè)較大的印記簇,分別位于人7q21、7q32、11p15、14q32、15q11-q13、19q13和20q13,另外這些印記簇包含基因較多(表1)[31]。
植物中發(fā)現(xiàn)的且已經(jīng)證實(shí)的印記基因不多,主要發(fā)生在擬南芥和玉米中,無明顯成簇分布現(xiàn)象,大多數(shù)印記基因?yàn)槟赶颠z傳表達(dá)即父系印記基因,印記方式有DNA甲基化和組蛋白H3K27me3。擬南芥中印記基因有MEDEA(MEA)/FIS1、FIS2、FIS3/FIE和FWA等,位于1號(hào)、2號(hào)和4號(hào)染色體中,且都為母源表達(dá)基因,在營(yíng)養(yǎng)組織和胚中體現(xiàn)雙等位基因表達(dá)[11-13,32]。在玉米的4號(hào)、7號(hào)和10號(hào)染色體上發(fā)現(xiàn)FIE1、FIE2、R、MEG1等印記基因,也都為母源表達(dá)基因[4,33-34]。小鼠中發(fā)現(xiàn)的依賴組蛋白甲基化修飾的印記基因無明顯成簇現(xiàn)象,包括Grb10、Phf7、Smbt、2Slc38a4和smoc1,這些都為父源表達(dá)等位基因[24]。
2.3時(shí)空性、組織特異性和保守性
基因的印記遺傳具有時(shí)空性,即不同發(fā)育階段基因遺傳印記表現(xiàn)不同。如鼠在交配后7.5d的滋養(yǎng)層細(xì)胞中Mash2基因表現(xiàn)為雙親基因的等位表達(dá),即雙等位基因表達(dá),而8.5d之后則表現(xiàn)為只表達(dá)母方等位基因的遺傳特性[35],即單等位表達(dá);另外,鼠的Igf2r基因在妊娠期的第4.5天,胚胎未植入,所有細(xì)胞表現(xiàn)為雙親等位基因的表達(dá),但到妊娠期的第6.5天至第13.5天,此時(shí),Igf2r在已植入的胚胎細(xì)胞中僅表達(dá)母方等位基因[36]。
基因的印記遺傳具有組織特異性和物種差異性。在1月齡豬中MEST基因在心、胃、肌肉、腎臟、肺、膀胱、舌頭和脂肪中,MEST表達(dá)為父源等位基因即為母源印記,而在肝臟、小腸和脾中為雙等位表達(dá)[37];人MEST基因有2種剪接體形式存在,分別是MEST1和MEST2,在所有組織中MEST1基因剪接體只表達(dá)父本的等位基因,而剪接體MEST2在心、腦、脾、胃、腎上腺等11個(gè)組織表達(dá)雙親等位基因,在胎兒的腎和胎盤組織只表達(dá)父方等位基因[38-39]。
印記基因的遺傳具有保守性。如在小鼠中發(fā)現(xiàn)印記基因,人類這些基因也被證實(shí)具有類似的印記現(xiàn)象,發(fā)現(xiàn)有100~200個(gè)印記基因[29],說明物種間印記基因遺傳具有保守性。目前為止,人和鼠中已被鑒定的印記基因分別約為45個(gè)和74個(gè),其中在人和小鼠中共同鑒定的印記基因有34個(gè)[29]。在這34個(gè)人和小鼠同時(shí)鑒定的印記基因中,人和小鼠中印記方向一致有26個(gè)基因,2個(gè)基因表現(xiàn)為印記的組織和發(fā)育階段特異性,6個(gè)基因表現(xiàn)為印記方向不同或一方印記而另一方非印記[29]。
在植物擬南芥和玉米中證實(shí)的印記基因不多,印記現(xiàn)象只發(fā)現(xiàn)在胚乳組織中具有組織印記特異性。擬南芥中FIS和FWA基因只在胚乳中表現(xiàn)印記現(xiàn)象,且都只表現(xiàn)為母方等位基因[11-13,32];玉米中FIE1、FIE2和R基因也都在胚乳組織中表達(dá)母本等位基因,胚中無此現(xiàn)象發(fā)生[4,33-34]。
因此,哺乳動(dòng)物印記基因的表達(dá)具有時(shí)空性、組織特異性和保守性,而目前發(fā)現(xiàn)的植物印記基因不多,印記基因表現(xiàn)的特點(diǎn)為組織特異性,還未發(fā)現(xiàn)印記基因應(yīng)有的其他特點(diǎn)。
3基因組印記與生長(zhǎng)發(fā)育
基因組印記現(xiàn)象與動(dòng)植物的生長(zhǎng)發(fā)育、疾病、哺乳行為和重要經(jīng)濟(jì)性狀息息相關(guān)。PHLDA2(TssC3)是第一個(gè)與細(xì)胞凋亡相關(guān)的印記基因[40],在人和鼠中分別位于11p15和7號(hào)染色體,其母源等位基因表達(dá)方式相繼在人、鼠、牛、豬中得到證實(shí),大量表達(dá)于胎盤滋養(yǎng)細(xì)胞層細(xì)胞,對(duì)調(diào)控胎盤和胎兒生長(zhǎng)發(fā)育方面有著重要功能[41-43]。Igf2-Cdkn1印記域在人和鼠中基因序列和印記表達(dá)狀態(tài)高度保守,這些基因是最早發(fā)現(xiàn)的印記基因,調(diào)控機(jī)體的發(fā)育和分化,已證實(shí)貝金威思-威德曼綜合征(Beckwith-Wiedemannsyndrome,BWS)與該印記域內(nèi)印記基因的突變和缺失相關(guān)[44]。人的15q11-q13印記域包含SNRPN、IPW、ZNF127、NDN的父源表達(dá)基因和UBE3A的母源表達(dá)基因,該區(qū)域印記狀態(tài)的變化受SNRPN上游印記中心(imprintingcentre,IC)微缺失和突變影響,從而導(dǎo)致天使綜合征(angelmansyndrome,AS)和普拉德-威利綜合征(Prader-Willisyndrome,PWS)的發(fā)生[45-49]。鼠的父源表達(dá)基因PEG3被證實(shí)有養(yǎng)育行為的功能,與人的PEG3基因同源性高度相似,也為父源表達(dá)基因,它們都在腦組織中表達(dá)量高,推測(cè)可能同樣具有哺育后代行為的功能[50-51]。家畜中存在很多調(diào)控性狀的印記基因,包括綿羊的后群肌肉肥大母系印記基因callipyge[52]、調(diào)控蒙古羊胸椎數(shù)量的母系印記基因Homebox[53]和調(diào)控豬背膘厚度、眼肌高度和肌內(nèi)脂肪含量的印記QTL[54]。在植物擬南芥中發(fā)現(xiàn)的一系列FIS印記基因具有調(diào)控中央細(xì)胞分裂和調(diào)節(jié)早期胚乳發(fā)育的功能,相應(yīng)的突變體表現(xiàn)為胚和胚乳發(fā)育不全現(xiàn)象[15]。
4展望
基因組印記現(xiàn)象廣泛存在于哺乳動(dòng)物和開花植物體內(nèi),對(duì)動(dòng)植物的正常生長(zhǎng)和發(fā)育發(fā)揮著重要作用,包括動(dòng)物胚胎、胎盤的發(fā)育和生長(zhǎng)以及開花植物種子胚乳的發(fā)育。基因組的印記還與人的腫瘤形成相關(guān),而且基因組印記與DNA的甲基化或組蛋白的修飾因果關(guān)系尚不明確,深入研究基因組印記機(jī)制,將會(huì)為腫瘤的診斷和治療提供新的方法。目前,基因組印記現(xiàn)象存在于植物種子胚乳發(fā)育的過程中,這種方式也可能參與植物抗逆境脅迫的機(jī)理,可能包括DNA甲基化、組蛋白甲基化或乙酰化或磷酸化或泛素化和IncRNAs(longnoncodingRNAs)以及染色質(zhì)重塑等,這些修飾對(duì)基因組印記調(diào)控起著重要作用。
參考文獻(xiàn)
[1]周泉勇,唐艷強(qiáng),趙書紅.豬印記基因的研究現(xiàn)狀及其應(yīng)用前景[J].豬業(yè)科學(xué),2010,27(7):30-33.
[2]REIKW,WALTERJ.Genomicimprinting:Parentalinfluenceonthegenome[J].NatRevGenet,2001,2(1):21-32.
[3]CROUSEHV.ThecontrollingelementinsexchromosomebehaviorinSciara[J].Genetics,1960,45(10):1429-1443.
[4]KERMICLEJL.DependenceoftheRmottledaleuronephenotypeinmaizeonmodeofsexualtransmission[J].Genetics,1970,66(1):69-85.
[5]MCGRATHJ,SOLTERD.Completionofmouseembryogenesisrequiresboththematernalandpaternalgenomes[J].Cell,1984,37(1):179-183.
[6]DECHIARATM,ROBERTSONEJ,EFSTRATIADISA.ParentalimprintingofthemouseinsulinlikegrowthfactorIIgene[J].Cell,1991,64(4):849-859.
[7]BARTOLOMEIMS,ZEMELS,TILGHMANSM.ParentalimprintingofthemouseH19gene[J].Nature,1991,351(6322):153-155.
[8]BARLOWDP,STGERR,HERRMANNBG,etal.Themouseinsulinlikegrowthfactortype2receptorisimprintedandcloselylinkedtotheTmelocus[J].Nature,1991,349(6304):84-87.
[9]RAINIERS,JOHNSONLA,DOBRYCJ,etal.Relaxationofimprintedgenesinhumancancer[J].Nature,1993,362(6422):747-749.
[10]SCOTTRJ,SPIELMANM.Deeperintothemaize:Newinsightsintogenomicimprintinginplants[J].Bioessays,2006,28(12):1167-1171.
[11]BERGERF,CHAUDHURYA.Parentalmemoriesshapeseeds[J].TrendsPlantSci,2009,14(10):550-556.
[12]KINOSHITAT,YADEGARIR,HARADAJJ,etal.ImprintingoftheMEDEApolycombgeneintheArabidopsisendosperm[J].PlantCell,1999,11(10):1945-1952.
[13]BERGERF,GRINIPE,SCHNITTGERA.Endosperm:Anintegratorofseedgrowthanddevelopment[J].CurrOpinPlantBiol,2006,9(6):664-670.
[14]LUOM,BILODEAUP,DENNISES,etal.ExpressionandparentoforigineffectsforFIS2,MEA,andFIEintheendospermandembryoofdevelopingArabidopsisseeds[J].PNAS,2000,97(19):10637-10642.
[15]SCOTTRJ,SPIELMANM.Deeperintothemaize:Newinsightsintogenomicimprintinginplants[J].Bioessays,2006,28(12):1167-1171.
[16]KILLIANJK,BYRDJC,JIRTLEJV,etal.M6P/IGF2Rimprintingevolutioninmammals[J].MolCell,2000,5(4):707-716.
[17]ALLEMANM,DOCTORJ.Genomicimprintinginplants:Observationsandevolutionaryimplications[J].PlantMolBiol,2000,43(2/3):147-161.
[18]NIKAIDOI,SAITOC,MIZUNOY,etal.Discoveryofimprintedtranscriptsinthemousetranscriptomeusinglargescaleexpressionprofiling[J].GenomeRes,2003,13(6B):1402-1409.
[19]LUEDIPP,HARTEMINKAJ,JIRTLERL.Genomewidepredictionofimprintedmurinegenes[J].GenomeRes,2005,15(6):875-884.
[20]GLASERRL,RAMSAYJP,MORISONIM.Theimprintedgeneandparentoforigineffectdatabasenowincludesparentaloriginofdenovomutations[J].NucleicAcidsRes,2006,34:D29-D31.
[21]REIKW,SANTOSF,DEANW.Mammalianepigenomics:Reprogrammingthegenomefordevelopmentandtherapy[J].Theriogenology,2003,59(1):21-32.
[22]BENDERJ.DNAmethylationandepigenetics[J].AnnuRevPlantBiol,2004,55(1):41-68.
[23]BRANDEISM,KAFRIT,ARIELM,etal.Theontogenyofallelespecificmethylationassociatedwithimprintedgenesinthemouse[J].EMBOJ,1993,12(9):3669-3677.
[24]INOUEA,JIANGL,LUF,etal.MaternalH3K27me3controlsDNAmethylationindependentimprinting[J].Nature,2017,547(7664):419-424.
[25]JULLIENPE,KINOSHITAT,OHADN,etal.MaintenanceofDNAmethylationduringtheArabidopsislifecycleisessentialforparentalimprinting[J].PlantCell,2006,18(6):1360-1372.
[26]CAOR,ZHANGY.ThefunctionsofE(Z)/EZH2mediatedmethylationoflysine27inhistoneH3[J].CurrOpinGenetDev,2004,14(2):155-164.
[27]HSIEHTF,IBARRACA,SILVAP,etal.GenomewidedemethylationofArabidopsisendosperm[J].Science,2009,324(5933):1451-1454.
[28]CHOIY,GEHRINGM,JOHNSONL,etal.DEMETER,aDNAglycosylasedomainprotein,isrequiredforendospermgeneimprintingandseedviabilityinArabidopsis[J].Cell,2002,110(1):33-42.
[29]FALLSJG,PULFORDDJ,WYLIEAA,etal.Genomicimprinting:Implicationsforhumandisease[J].AmJPathol,1999,154(3):635-647.
[30]WANLB,BARTOLOMEIMS.Regulationofimprintinginclusters:NoncodingRNAsversusinsulators[J].AdvGenet,2008,61:207-223.
[31]MORISONIM,RAMSAYJP,SPENCERHG.Acensusofmammalianimprinting[J].TrendsGenet,2005,21(8):457-465.
[32]KINOSHITAT,MIURAA,CHOIY,etal.OnewaycontrolofFWAimprintinginArabidopsisendospermbyDNAmethylation[J].Science,2004,303(5657):521-523.
[33]GUTIRRZMARCOSJF,COSTALM,DALPRM,etal.Epigeneticasymmetryofimprintedgenesinplantgametes[J].NatGenet,2006,38(8):876-878.
[34]GUTIRREZMARCOSJF,COSTALM,BIDERREPETITC,etal.Maternallyexpressedgene1isanovelmaizeendospermtransfercellspecificgenewithamaternalparentoforiginpatternofexpression[J].PlantCell,2004,16(5):1288-1301.
[35]GUILLEMOTF,CASPARYT,TILGHMANSM,etal.GenomicimprintingofMash2,amousegenerequiredfortrophoblastdevelopment[J].NatGenet,1995,9(3):235-242.
[36]LERCHNERW,BARLOWDP.PaternalrepressionoftheimprintedmouseIgf2rlocusoccursduringimplantationandisstableinalltissuesofthepostimplantationmouseembryo[J].MechDev,1997,61(1/2):141-149.
[37]李順,蔣曹德,石萍,等.豬COPG2和MEST克隆、印記狀況和組織表達(dá)分析[J].中國(guó)農(nóng)業(yè)科學(xué),2010,43(23):4901-4909.
[38]KOSAKIK,KOSAKIR,CRAIGENWJ,etal.IsoformspecificimprintingofthehumanPEG1/MESTgene[J].AmJHumGenet,2000,66(1):309-312.
[39]MCMINNJ,WEIM,SADOVSKYY,etal.ImprintingofPEG1/MESTisoform2inhumanplacenta[J].Placenta,2006,27(2/3):119-126.
[40]LEEMP,F(xiàn)EINBERGAP.Genomicimprintingofahumanapoptosisgenehomologue,TSSC3[J].CancerRes,1998,58(5):1052-1056.
[41]QIANNF,F(xiàn)RANKD,O′KEEFED,etal.TheIPLgeneonchromosome11p15.5isimprintedinhumansandmiceandissimilartoTDAG51,implicatedinFasexpressionandapoptosis[J].HumMolGenet,1997,6(12):2021-2029.
[42]SIKORAKM,MAGEEDA,BERKOWICZEW,etal.PHLDA2isanimprintedgeneincattle[J].AnimGenet,2012,43(5):587-590.
[43]HOUSP,CHENYM,LIANGJ,etal.DevelopmentalstagespecificimprintingofIPLindomesticpigs(Susscrofa)[J].JBiomedBiotechnol,2010,2010:1-6.
[44]OHLSSONR,NYSTMA,PFEIFEROHLSSONS,etal.IGF2isparentallyimprintedduringhumanembryogenesisandintheBeckwithWiedemannsyndrome[J].NatGenet,1993,4(1):94-97.
[45]WEVRICKR,KERNSJA,F(xiàn)RANCKEU.IdentificationofanovelpaternallyexpressedgeneinthePraderWillisyndromeregion[J].HumMolGenet,1994,3(10):1877-1883.
[46]GLENNCC,NICHOLLSRD,ROBINSONWP,etal.Modificationof15q11q13DNAmethylationimprintsinuniqueAngelmanandPraderWillipatients[J].HumMolGenet,1993,2(9):1377-1382.
[47]REEDML,LEFFSE.MaternalimprintingofhumanSNRPN,agenedeletedinPraderWillisyndrome[J].NatGenet,1994,6(2):163-167.
[48]SUTCLIFFEJS,HANM,CHRISTIANSL,etal.Neuronallyexpressednecdingene:AnimprintedcandidategeneinPraderWillisyndrome[J].Lancet,1997,350(9090):1520-1521.
[49]ALBRECHTU,SUTCLIFFEJS,CATTANACHBM.ImprintedexpressionofthemurineAngelmansyndromegene,Ube3a,inhippocampalandPurkinjeneurons[J].NatGenet,1997,17(1):75-78.
[50]KUROIWAY,KANEKOISHINOT,KAGITANIF,etal.Peg3imprintedgeneonproximalchromosome7encodesforazincfingerprotein[J].NatGenet,1996,12(2):186-190.
[51]KIMJ,ASHWORTHL,BRANSCOMBE,etal.Thehumanhomologofamouseimprintedgene,Peg3,mapstoazincfingergenerichregionofhumanchromosome19q13.4[J].GenomeRes,1997,7(5):532-540.
[52]KOOHMARAIEM,SHACKELFORDSD,WHEELERTL,etal.Amusclehypertrophyconditioninlamb(callipyge):Characterizationofeffectsonmusclegrowthandmeatqualitytraits[J].JAnimSci,1995,73(12):3596-3607.
[53]張立嶺,菊林花,楊麗君.蒙古羊胸椎數(shù)的親本印記遺傳研究[J].內(nèi)蒙古農(nóng)業(yè)大學(xué)學(xué)報(bào)(自然科學(xué)版),2000,21(2):1-6.
[54]DEKONINGDJ,RATTINKAP,HARLIZIUSB,etal.Genomewidescanforbodycompositioninpigsrevealsimportantroleofimprinting[J].PNAS,2000,97(14):7947-7950.

