絲狀真菌代謝組學研究方法進展
閆震 陳萬權 張昊 劉太國 劉長仲 馮潔

摘要 代謝組學研究的是生物體系受到內在和外在因素刺激產生的內源性代謝變化。近年來,隨著代謝組學在病原真菌致病機理、真菌與植物互作以及新型生物活性物質開發研究領域的應用,真菌代謝組學越來越受到國內外的重視,同時也取得了較大的進展。由于絲狀真菌類型和研究目的的差異,其研究方法不盡相同。本文主要綜述近年來國內外絲狀真菌代謝組學分析方法的研究進展,從絲狀真菌樣品制備、代謝物鑒定、數據分析、生物標志物發掘、代謝通路方面進行概述,以期為該領域研究者提供參考,進一步推進代謝組學在真菌研究領域中的應用。
關鍵詞 絲狀真菌; 代謝組學; 樣品處理; 數據分析; 代謝通路
中圖分類號: Q 591.1
文獻標識碼: A
DOI: 10.16688/j.zwbh.2020357
Advances in research methods of filamentous fungal metabolomics
YAN Zhen1, 2, 3, CHEN Wanquan1, 2, 4*, ZHANG Hao2, 4*, LIU Taiguo2, 4, 5*, LIU Changzhong1, FENG Jie2
(1. College of Plant Protection, Gansu Agricultural University, Lanzhou 730070, China; 2. State Key Laboratory
for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural
Sciences, Beijing 100193, China; 3. Institute of Pomology, Chinese Academy of Agricultural Sciences,
Xingcheng 125100, China; 4. National Agricultural Experimental Station for Plant Protection at Gangu, Ministry
of Agriculture and Rural Affairs, Tianshui 741200 China; 5. Key Laboratory of Biological Hazard Factors (Plant Source)
Control of Agricultural Products Quality and Safety, Ministry of Agriculture and Rural Affairs, Beijing 100193, China)
Abstract
Metabolomics studies the endogenous metabolic changes in biological systems stimulated by internal and external factors. In recent years, with the application of metabolomics in the pathogenesis of pathogenic fungi and interaction with plants,? the development of novel biologically active substances, more and more attentions have been paid to the study of fungal metabolomics, and great progresses have been made in this area. Due to the differences in the types of filamentous fungi and the purposes of the research, the research methods were diverse. This review summarized the research progresses in the metabolomic analysis methods of filamentous fungi in recent years, including filamentous fungal sample preparation, metabolite identification, data analysis, biomarker discovery and metabolic pathways. It is aimed to provide a reference for the researchers in this field and further promote the application of metabolomics to fungal research.
Key words
filamentous fungus; metabolomics; sample preparation; data analysis; metabolic pathways
代謝組學是對某一生物或細胞在一特定生理時期內所有低分子量(小于1 000)代謝產物進行定性和定量分析的一門學科,主要包括糖類、脂質、核苷酸、氨基酸等初級代謝產物以及萜類、聚酮類、非核糖體多肽、生物堿類等次級代謝產物[1-2]。雖然經常被視為轉錄組學和蛋白質組學的補充技術,但代謝組學確實具有明顯的優勢[3-5]。 首先,作為基因轉錄和蛋白質組表達的下游產物,代謝組與生物系統表型直接相關,可以直接動態地反映出細胞的生理生化過程,能夠更準確直接地反映生命體終端和表型信息[6]。其次,相對于轉錄組或蛋白質組,代謝組的變化被放大,通過結果直接突出代謝途徑和不同調控過程。代謝組學是功能基因組學所采用的組學技術中最新的一種[7],已經成為生物學的一個新領域,有望加快對功能未知基因的功能分析。
目前,代謝組學已擴展到各個領域,例如臨床、制藥、環境、動物、植物和微生物等[8]。相比于動、植物代謝組學,真菌代謝組學起步較晚,在過去的十年中,由于植物病原真菌在微生物系統中的重要性,代謝組學技術已被廣泛用于植物病原真菌的研究中[9],主要側重于真菌分類[10-12]、致病機理[3,13]以及與植物互作過程中代謝物功能和代謝途徑[14-16]。國內外真菌代謝研究相關領域文獻發表數量見圖1,主要側重于代謝產物、生物活性、抑菌活性、結構鑒定和發酵等方面的研究。代謝組學可用于檢測正常的遺傳發育以及寄主刺激引起的代謝組特征變化,從全局角度反映出真菌的表型變化[17],也可用于獲得植物在感染病原真菌后產生的小分子代謝物[18-19]。為了研究植物的免疫力,還使用源自植物病原真菌的激發子來誘導寄主植物的免疫反應,進行代謝研究。許多植物病原真菌,例如鐮刀菌Fusarium和曲霉Aspergillus都可以在寄主細胞中產生毒素[20]。因此,代謝組學還可以很好地檢測毒素誘導的植物代謝途徑的變化。
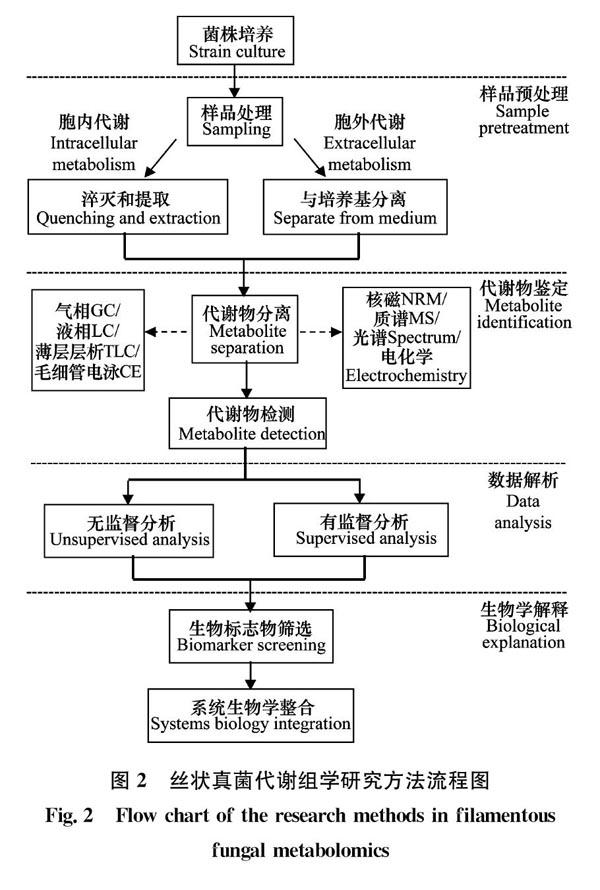
真菌代謝組分為胞內代謝物和胞外代謝物,通常將對胞內代謝物的定量定性分析稱為代謝指紋分析(metabolic fingerprinting analysis),將胞外代謝物的定量定性分析稱為代謝足跡分析(metabolic footprinting analysis)[21]。代謝指紋分析相比于代謝足跡分析,樣品取樣和準備過程較為復雜,但代謝指紋分析所涵蓋的代謝物種類顯著多于代謝足跡分析,所含信息更加豐富,所以當前以微生物為研究對象的代謝組學研究中,多數集中于代謝指紋分析。然而,由于真菌培養的樣品中生物質濃度較低,限制了對細胞內數量少的代謝物的檢測。以曲霉和青霉Penicillium為例,真菌細胞的次級代謝產物據估計達到10 000多種,但目前已知的代謝產物不足10%[22];另外,將細胞外和細胞內代謝物的分離與細胞內代謝的淬滅結合起來非常困難, 致使真菌代謝組學的研究面臨很多挑戰[23]。真菌代謝組學研究方法主要包括樣品制備、代謝物鑒定、數據解析、生物標志物發掘、生物學解釋等一系列關鍵步驟(圖2),本文主要圍繞這幾方面展開討論闡述。
1 樣品制備
樣品制備是代謝組學分析的關鍵步驟,對隨后的數據采集和生物學解釋具有重要的意義[24]。因此,應根據研究目的針對性地選擇樣品處理方法,真實客觀地反映樣品中代謝物的存在信息。理想的樣品制備方法應簡單易于重復、步驟少可變性最低、成本低適用于大樣本量,且能夠保持樣品的完整性[25]。由于細胞對外界環境變化極其敏感,故代謝產物的含量及水平會隨環境改變迅速發生變化[26]。因此,應盡快完成采樣并終止細胞代謝(淬滅),以確保分析時樣品的代謝組成分與取樣時一致。真菌樣品制備主要包括菌株培養、快速取樣、代謝淬滅和代謝萃取[27]。通常取樣和淬滅步驟結合在一起以縮短處理時間。
1.1 菌株培養
菌株需要在一定的可控生物反應器中培養。生物反應器中的培養基組分、溫度、pH、光照以及溶解于其中的O2、CO2 等必須是確定和易于控制與檢測的,這樣才能獲得標準和重現的培養條件。真菌多采用分批培養和連續培養方式。Chen等[28]和Palyzov等[29]使用PDA培養基培養真菌,從中提取代謝產物,但由于PDA培養基中含有具有生物活性的馬鈴薯,其不同批次之間營養成分難以達到統一標準,僅適用于少量樣品統一處理,對于大量樣本檢測的重復性操作不可控制,因此,培養基成分適合選擇化學合成的物質,便于控制培養條件的一致性[30-31]。
1.2 快速取樣
快速取樣不僅能夠防止底物濃度發生巨大變化,而且有助于維持代謝物的穩定性。對于真菌最關鍵的問題是菌絲與生長培養基的有效分離,目前主要分離方式是冷離心或快速過濾[32]。細胞外代謝產物的取樣和樣品制備相對簡單,一般離心或過濾后,取上清液或濾液直接淬滅或冷凍儲存[24]。然而對于胞內代謝物, Francisca等[33]研究黑曲霉Aspergillus niger時發現,通過離心分離菌絲體是不切實際的,因為沒有形成合適的菌絲團沉淀,盡管Seifar等[34]曾嘗試離心后清洗菌絲沉淀,但試管中仍會存在大量的培養基或清洗液,導致細胞外代謝產物殘留。與離心相比,過濾可以更有效地去除菌絲體中的細胞外代謝物[31,33]。Juliana等[35]研究尖孢鐮刀菌F.oxysporum聚酮類代謝物時,采用先離心后真空過濾的方法,但暴露時間過長。Choi等[36]分析釀酒酵母Saccharomyces cerevisiae胞內代謝物時,比較了淬滅后離心和快速過濾對代謝物泄漏的影響,同樣表明快速過濾能夠很大程度上減少代謝物的損失。盡管過濾過程中可能會導致具有快速周轉率代謝物(例如,磷酸化的中間體)的數量在重復樣品中會有所不同[37],但可以考慮通過中心代謝產物的變化補償不一致性[26]。比如,磷酸烯醇丙酮酸、NAD+/NADP+和谷氨酸等。所以迄今為止,快速過濾仍然是絲狀真菌的最佳取樣方法[30]。
1.3 代謝淬滅
理想的淬滅技術應實現瞬間淬滅,快速淬滅酶活力并且保持細胞或生物的完整性,避免造成胞內代謝物外流[27]。由于真菌細胞壁的結構不同,對滲透壓的耐受程度以及膜通透性也存在差異,通常很難通過單一的提取方法提取全部胞內代謝物[38]。針對不同樣品發展了各類淬滅試劑,液氮、高氯酸、冷甲醇(-40℃)、熱乙醇、冷緩沖鹽(PBS、Tricine、HEPES或碳酸銨)等[39]。也有報道用等滲鹽水(09%m/V NaCl,0.5℃)淬滅能有效防止細胞膜損傷,從而避免不必要的代謝物損失。盡管也有研究者提出采用快速熱休克來使新陳代謝失活,但這可能導致熱不穩定代謝產物降解并增加細胞通透性[23,40]。使用60%甲醇(-40℃)的淬滅方法在各種真菌中應用最廣泛[7,22,31]。但許多研究證明冷甲醇淬滅會破壞細胞完整性,影響細胞內代謝物的回收,從而導致代謝物水平的低估[41-43]。Zheng等[44]研究黑曲霉代謝物時,從綜合淬滅效率、細胞損傷和代謝產物泄漏3個方面評估了3種冷甲醇淬滅方法(-20℃ 40%甲醇,-40℃ 60%甲醇和-40℃純甲醇)與液氮的差異,發現與冷甲醇淬滅方法相比,液氮對細胞完整性破壞最小,產生的代謝物更穩定。當測定胞內代謝物時,為避免受到胞外代謝物的干擾,有時需要洗滌菌絲以去除細胞外基質。洗滌緩沖液應符合生理水平,采用比較溫和的溶液,最大程度減少代謝物泄漏,一般是預冷的PBS、生理鹽水或蒸餾水[30,45-46]。包括洗滌在內的整個過濾過程要求在30 s或45 s內完成[30,47]。
1.4 代謝萃取
代謝物的提取是樣品前處理過程中又一重要步驟,對于非靶標代謝組學分析,其無偏向性和全覆蓋率的內在要求,對代謝物的提取提出了較高的外在要求。理想的方法應盡可能提高代謝物的覆蓋率和回收率,保持代謝物的物理化學性質穩定,減少代謝物損失[48-49]。首要步驟是細胞破碎,使細胞膜可滲透提取劑(例如有機溶劑),從而釋放細胞內代謝產物[18]。隨后進行代謝物的提取和樣品濃縮。細胞破碎方法分機械破碎和非機械破碎。機械破碎方法主要有超聲破碎細胞,微波輔助萃取,加壓液體萃取,超臨界流體萃取、手動研磨,球磨機等。非機械破碎包括酶促裂解、物理溶解和化學溶解。物理溶解常用的是凍融和加熱法,由于樣品中存在熱不穩定化合物,加熱法不太適用于全組分。化學溶解法主要是各類有機溶劑對細胞膜的破壞而使細胞內代謝物釋放[39]。
目前,常用的提取劑有冷甲醇、熱乙醇、乙腈、乙酸乙酯、二氯甲烷、高氯酸或堿以及氯仿-甲醇混合液等[27,33,50-51]。Karpe等[52]提取尖孢鐮刀菌胞內代謝物時采用乙腈/水/乙酸混合液,雖然乙腈除蛋白的效果比甲醇和乙醇好,但乙腈對代謝物的提取效率最低,可重復的代謝特征較少[21]。美國Scripps研究所通過聚類分析發現,100%的甲醇或包含甲醇的混合提取溶劑可以實現高重復性檢出代謝物。Smart等[26]使用冷甲醇水溶液提取與凍融循環結合,提高了細胞的滲透性,增強了代謝產物的化學提取。Tomas等[48]的研究表明使用甲醇和水混合液對鐮刀菌代謝產物進行提取,能夠兼顧高極性和低極性的化合物。由于真菌微生物樣品內代謝物種類多,物理化學性質多元(極性和溶解性等),到目前為止,還未有一種提取體系可實現全覆蓋提取代謝物,因此根據研究目的有針對性地選取提取劑至關重要[9]。
2 代謝物鑒定
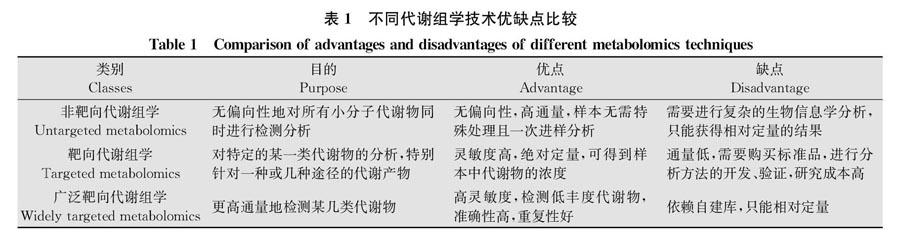
由于特定的代謝物在每一個含有它的有機體中都是相同的,所以代謝物鑒定技術是通用的。根據研究目的和研究技術不同,代謝組學主要分為非靶向代謝組學和靶向代謝組學兩種典型的方法,前者可利用高分辨率質譜(high resolution mass spectrometry,HRMS)獲得樣品中盡可能多的代謝物信息,具有無偏性,但只能相對定量,通常主要用于區分代謝表型或發現差異代謝物的研究[21,53];后者則主要通過使用色譜(LC/GC)或串聯低分辨率質譜(MS),針對特定代謝物進行分析,能夠絕對定量,但覆蓋率低。Chen等[54]基于Q-Trap質譜儀發展了“廣泛靶向”代謝組學技術,將非靶向代謝組學高通量的優點與靶向代謝組學高準確度、高靈敏度的優點相結合,對關注的某類特定物質進行檢測,生物標志物篩選更加準確,顯著提高了信息覆蓋率和定量準確性。三者的優缺點見表1。
代謝物分析鑒定的兩大難點是生物體內小分子種類繁多、濃度相差懸殊,從真菌中獲得代謝物化學成分的精確信息是一項具有挑戰性的任務[55]。目前還沒有一種代謝組學分析平臺可以無偏向性地涵蓋所有代謝物。色譜、質譜、核磁共振、紅外光譜、紫外吸收、熒光散射、光散射等分離分析手段及其組合都出現在代謝組學的研究中[23],當前核磁共振(NMR)和質譜(MS)是代謝組學研究領域的主流分析平臺,在代謝組學中發揮重要作用[56]。
2.1 核磁共振分析技術
NMR是一種無損、高通量的檢測技術,利用生物流體的NMR光譜提供生物中所有小分子代謝物的豐富信息,并通過多變量統計分析和模式識別處理來確定相關生物的完整代謝圖[9]。當前使用的NMR技術包括氫譜(1H-NMR),碳譜(13C-NMR)和磷譜(31P-NMR),其中最廣泛使用的是1H-NMR[57]。NMR相對于MS的優勢在于制備簡單,物質的結構易于識別。此外,頻譜上的信號強度與代謝物的濃度直接相關,可以準確定量代謝產物[58]。但是,NMR的靈敏度很低,很難同時檢測生物體內濃度差異高的代謝物系統,并且無法區分具有非常相似結構和化學位移的代謝物,例如肌酸與磷酸肌酸,或尿苷二磷酸鹽與尿苷三磷酸[3,17],限制了其在真菌代謝組學中的發展。
2.2 質譜分析技術
由于靈敏度高,質量準確度高和動態范圍廣,MS在代謝組學研究中起著越來越多的作用[59-61],因為它可以連接不同的色譜分離模式[62-64],例如氣相色譜-質譜(GC-MS)、氣相色譜串聯質譜(GC-MS/MS)、液相色譜-質譜(LC-MS)、液相色譜串聯質譜(LC-MS/MS)。氣質聯用主要針對具有揮發性且在高溫下必須穩定的化合物。一些非揮發性代謝物(例如糖,氨基酸)可以通過衍生作用轉化成揮發性化合物。盡管化學衍生化改善了代謝物的覆蓋范圍,但這可能為代謝組學研究引入了另一種變異性來源。該方法的最大優點是可以使用標準庫進行結構鑒定,并且可以在大量數據庫中檢索[65]。與氣質聯用不同,液質聯用主要利用“軟電離”技術將分析物電離,通過選擇離子監測模式(SIM)或多反應監測(MRM)模式進行,保留了完整的分子信息,這對于識別至關重要。由于它有更寬的線性范圍,更好的可重復性和更高的靈敏度,被更加廣泛地應用到靶向代謝組學研究中。
高分辨質譜(HRMS)憑借其普適性、高靈敏度和特異性的特點,逐漸成為非靶向和廣靶代謝組學研究的主流技術[66]。目前主流的HRMS有飛行時間質譜(TOF-MS)、靜電場軌道阱質譜(Orbitrap-MS)、傅立葉變換離子回旋共振質譜(FTICR-MS)[67]。相比TOF-MS,Orbitrap-MS具有更低的測量質量誤差, 同時在儀器穩定性和重現性上顯著優于TOF-MS, FTICR-MS結合了超高的質量分辨率和卓越的質量精度,是目前測量精度最高的質譜,但其掃描速度較前兩者更慢, 在代謝組學研究中應用較少[21]。平行反應監測(PRM)是在HRMS平臺上開發的新方法,對準確量化目標代謝物的貢獻越來越大[61]。Zhou等[68]在Q-Exactive LC-MS系統上開發了基于PRM的目標代謝物檢測策略,并進行了PRM和MRM測量的比較,結果表明,與QTRAP 6500上的MRM測量相比,PRM具有更高的質量精度和更全面的二級質譜信息。
不同分析工具各具優缺點,沒有適合的單一的分析工具可以精確地鑒定和定量數千個感興趣的小分子,需要組合使用不同的分析技術才能更好地覆蓋具有廣泛極性和分子量范圍的代謝物[69]。
3 數據分析
色譜分離代謝物后,會生成大量光譜數據和多變量數據,頻譜的每個信號峰都包含有關代謝物中各種物質的定性和定量信息[70]。因此,有必要使用統計學和化學計量學進行分析,具體分析流程見圖3。首先需要對原始數據進行預處理,包括基線校正,特征檢測,噪聲過濾,峰提取,峰對齊,解卷積和歸一化以消除干擾因素。可以使用軟件來實現這些過程。例如MetAlign,MZmine,XCMS,METIDEA,AMDIS和MSFACTS等。許多儀器制造商還開發了自己的專有軟件,例如MarkerLynx 和QI(美國Waters公司),AnalyzerPro(英國SpectralWorks公司),Mass Profiler(美國安捷倫公司),ChromsTof(美國Leco公司),MarkerView和SIEVE(美國Thermo公司)等[71]。
預處理后的數據需要進行多元統計分析和生物信息學分析,包括無監督分析和有監督分析兩種[3]。無監督分析能夠在未知樣品信息的情況下將樣品進行聚類分組,包括多變量數據分析(MADA)、主成分分析(PCA)、層次聚類分析(HCA)和自組織映射(SOA)[9,72]等。無監督分析方法不能忽略組內誤差,如果樣本組之間的差異太小或組內差異太大,則很難確定組之間的差異[73-74]。有監督分析包括偏最小二乘判別分析(PLS-DA)、正交偏最小二乘判別分析(OPLS-DA)、多元單變量數據分析(MUDA),線性判別分析(LDA)、神經網絡(NN)和支持向量機分析(SVM)等[75]。有監督分析方法能夠減少組內隨機誤差,突出組間系統誤差[76]。通過這些多元變量統計分析,可以獲得潛在的有效信息,并通過生物標志物和代謝途徑找到差異代謝物[77],其中PLS-DA是使用最多的有監督分析方法,用于篩選有標記的代謝物[78]。
4 代謝通路分析
生物標志物確定以后,需結合所研究的問題考察其生物學功能。數據的可視化可以展示數據集的特性,通常繪制代謝網絡的某一部分代謝途徑圖使數據可視化, 有助于理解代謝物之間的相互關系, 也能夠與基因表達數據相聯系進行功能基因組學研究,闡述病理生理作用相關的作用機制[79]。關于絲狀真菌代謝通路研究相對較少,目前主要關注在中心碳代謝和能量轉換方面。Liu等[51]探究了海藻糖生物合成基因TPS敲除導致禾谷鐮刀菌F.graminearum基礎代謝的變化,繪制了代謝途徑圖(圖4),證明了TPS基因在糖酵解、TCA循環、磷酸戊糖途徑中均具有調節作用。Vessela等[6]詳細闡述了禾谷鐮刀菌單端孢霉烯毒素的產生與其他中心碳代謝和次級代謝過程的關系,將收集的代謝物整合到代謝框架圖中(圖5),能夠將可能發生在代謝途徑之間的聯系可視化,并可以指導與代謝輪廓修飾相關的生物學解釋。Gu等[80]剖析了乙酰輔酶A合酶基因在炭疽病菌Colletotrichum higginsianum脂質代謝和毒力方面的作用,首次提出了參與病原體毒力的假設機制和模型(圖6),揭示了脂肪酸β-氧化、肉堿穿梭、乙醛酸的生化途徑,TCA循環、糖酵解、糖異生和脂肪酸生物合成之間的關聯。Cao等[81]證明了轉錄因子CRF1在稻瘟病菌Pyricularia oryzae發育和致病性中的生物學功能,該因子參與脂肪分解,過氧化物酶體β-氧化、乙醛酸循環、糖異生和甘油代謝,影響附著胞的滲透性和毒力。Brisson等[82]研究了擬青霉Paecilomyces真菌檸檬酸與中心碳代謝之間的關系。Aguilar-Pontes等[83]描述了黑曲霉初級碳代謝網絡途徑,通過對高質量黑曲霉基因組的功能預測,深入挖掘了該途徑中相關酶基因、反應及通路。然而,由于每種代謝物都可以參與多種生物合成途徑[75],要正確解釋代謝組學數據,繪制的代謝途徑應該完整,即包含代謝物相關的所有反應。傳統的代謝途徑圖,如常用的數據庫KEGG,可能不包括所有的平行反應, 這也增加了代謝組學數據解釋的難度。
Fig.6 Hypothetical model of the function of acetyl-CoA synthase in Colletotrichum higginsianum
5 展望
代謝組學是進行生物表型研究的主要手段之一,是系統生物學研究不可或缺的部分。真菌代謝組學是生物科學發展迅速的領域,隨著樣品制備方法的不斷完善以及分析技術的快速發展,在真菌領域中將會發揮更大的作用。然而,與環境和人類代謝組學相反,其潛力尚未得到充分利用,從總體來看,它仍然處于發展階段,目前代謝組學的最大挑戰是如何克服從大量的代謝產物中找出特異性的生物標志物,獲得最佳的生物學解釋。另一個主要挑戰是亞細胞區室化,因為代謝組學數據反映了各種細胞器中代謝物的總和。盡管從特定細胞器中分析代謝物是更可取的,但目前在保持這些結構代謝狀態的同時分離細胞器是非常困難的。由于生物系統的復雜性,其他組學的配合和交叉至關重要。通過組合來自全基因組測序,基因表達譜,蛋白質組學和保守的生化途徑信息,整合高通量、高分辨率的分析技術與生物信息學,實現自動分析和可視化,生成標準化代謝組學數據,進而構建細胞代謝模型。理想的模型可以預測生物體中存在的未知代謝途徑,進而推進該領域的快速發展。
參考文獻
[1] MATICH E K, NITA G, CHAVEZ S, et al. Applications of metabolomics in assessing ecological effects of emerging contaminants and pollutants on plants [J]. Journal of Hazardous Materials, 2019, 373: 527-535.
[2] 徐天潤, 劉心昱, 許國旺. 基于液相色譜-質譜聯用技術的代謝組學分析方法研究進展[J]. 分析測試學報, 2020, 39(1): 10-18.
[3] 劉宏有, 陳柳龍, 高江濤. 代謝組及其在真菌研究中的應用[J]. 菌物學報, 2019, 38(12): 2078-2086.
[4] KELL D B, BROWN M, DAVEY H M, et al. Metabolic footprinting and systems biology: The medium is the message [J]. Nature Reviews Microbiology, 2005, 3(7): 557-565.
[5] HOLLYWOOD K, BRISON D R, GOODACRE R. Metabolomics: Current technologies and future trends [J]. Proteomics, 2006, 6(17): 4716-4723.
[6] VESSELA P, LAURIE L, STPHANE B, et al. Mycotoxin biosynthesis and central metabolism are two interlinked pathways in Fusarium graminearum, as demonstrated by the extensive metabolic changes induced by caffeic acid exposure [J/OL]. Applied and Environmental Microbiology, 2018, 84(8): e01705-17. DOI: 10.1128/AEM.01705-17.
[7] JOEL P A, GUMMER, CHRISTIAN K, et al. Metabolomics protocols for filamentous fungi [J]. Methods in Molecular Biology, 2012, 835: 237-254.
[8] GONG Zhigang, HU Jing, WU Xi, et al. The recent developments in sample preparation for mass spectrometry-based metabolomics [J]. Critical Reviews in Analytical Chemistry, 2017, 47(4): 325-331.
[9] CHEN Fangfang, MA Ruijing, CHEN Xiaolin. Advances of metabolomics in fungal pathogen-plant interactions [J/OL]. Metabolites, 2019, 9(8): 169. DOI: 10.3390/metabo9080169.
[10]KANG D, KIM J, CHOI J N, et al. Chemotaxonomy of Trichoderma spp. using mass spectrometry-based metabolite profiling [J]. Journal of Microbiology and Biotechnology, 2011, 21(1): 5-13.
[11]FRISVAD J C. Fungal chemotaxonomy, biosynthesis and molecular genetics of fungal secondary metabolites [M]. New York: Springer-Verlag, 2015, 2: 103-121.
[12]ALIFERIS K A, CUBETA M A, JABAJI S. Chemotaxonomy of fungi in the Rhizoctonia solani species complex performing GC/MS metabolite profiling [J]. Metabolomics, 2013, 9(1): 159-169.
[13]KONSTANTINOS A A, DENIS F, SUHA J. A metabolic profiling strategy for the dissection of plant defense against fungal pathogens [J/OL]. PLoS ONE, 2014, 9(11): e111930. DOI: 10.1371/journal.pone.0111930.
[14]TENENBOIM H, BROTMAN Y. Omic relief for the biotically stressed: metabolomics of plant biotic interactions [J]. Trends in Plant Science, 2016, 21(9): 781-791.
[15]ROJAS C M, SENTHIL-KUMAR M, TZIN V, et al. Regulation of primary plant metabolism during plant-pathogen interactions and its contribution to plant defense [J/OL]. Frontiers in Plant Science, 2014, 5: 17. DOI: 10.3389/fpls.2014.00017.
[16]TUGIZIMANA F, MHLONGO M I, PIATER L A, et al. Metabolomics in plant priming research: the way forward ? [J/OL]. International Journal of Molecular Sciences, 2018, 19(6): 1759. DOI: 10.3390/ijms19061759.
[17]NAKABAYASHI R, SAITO K. Integrated metabolomics for abiotic stress responses in plants [J]. Current Opinion in Plant Biology, 2015, 24: 10-16.
[18]FEUSSNER I, POLLE A. What the transcriptome does not tell-proteomics and metabolomics are closer to the plants patho-phenotype [J]. Current Opinion in Plant Biology, 2015, 26: 26-31.
[19]TAN K C, IPCHO S V, TRENGOVE R D, et al. Assessing the impact of transcriptomics, proteomics and metabolomics on fungal phytopathology [J]. Molecular Plant Pathology, 2009, 10(5): 703-715.
[20]NESIC K, IVANOVIC S, NESIC V. Fusarial toxins: secondary metabolites of Fusarium fungi [M]. Switzerland: Springer International Publishing, 2014, 228: 101-119.
[21]謝華里. 基于液質聯用的黃曲霉代謝組學方法研究[D]. 北京: 中國農業科學院, 2017.
[22]靳夢琦, 李軍, 朱鳳妹, 等. 黑曲霉代謝組學研究進展[J]. 食品工程, 2017(4): 1-4.
[23]王智文, 馬向輝, 陳洵, 等. 微生物代謝組學的研究方法與進展[J]. 化學進展, 2010, 22(1):163-172.
[24]MAGORZATA P, JULIA J, MICHA J M. Sample preparation procedures utilized in microbial metabolomics: an overview [J]. Journal of Chromatography B, 2017, 1043: 150-157.
[25]JULIJANA I, ELIZABETH J W. From samples to insights into metabolism: uncovering biologically relevant information in LC-HRMS metabolomics data [J/OL]. Metabolites, 2019, 9(12): 308. DOI: 10.3390/metabo9120308.
[26]SMART K F, AGGIO R B, VAN H J R, et al. Analytical platform for metabolome analysis of microbial cells using methyl chloroformate derivatization followed by gas chromatography-mass spectrometry [J]. Nature Protocols, 2010, 5(10): 1709-1729.
[27]席曉敏, 張和平. 微生物代謝組學研究及應用進展[J]. 食品科學, 2016, 37(11): 283-289.
[28]CHEN Fangfang, ZHANG Jingtao, SONG Xiushi, et al. Combined metabonomic and quantitative real-time PCR analyses reveal systems metabolic changes of Fusarium graminearum induced by Tri5 gene deletion [J]. Journal of Proteome Research, 2011, 10(5): 2273-2285.
[29]PALYZOV A, SVOBODOV K, SOKOLOV L, et al. Metabolic profiling of Fusarium oxysporum f.sp. conglutinans race 2 in dual cultures with biocontrol agents Bacillus amyloliquefaciens, Pseudomonas aeruginosa, and Ttrichoderma harzianum [J]. Folia Microbiologica, 2019, 64(6): 779-787.
[30]LU Hengqian, CHEN Haiqin, TANG Xin, et al. Evaluation of metabolome sample preparation and extraction methodologies for oleaginous filamentous fungi Mortierella alpine [J/OL]. Metabolomics, 2019, 15(4): 50. DOI: 10.1007/s11306-019-1506-5.
[31]RUTGER D D, LODEWIJK P J, CASPAR T H, et al. Intracellular metabolite determination in the presence of extracellular abundance: application to the penicillin biosynthesis pathway in Penicillium chrysogenum [J]. Biotechnology and Bioengineering, 2010, 107(1): 105-115.
[32]TUULIA H, SUSANNE W. Chromatographic methods in metabolomics [M]. Finland: Royal Society of Chemistry, 2013: 19-34.
[33]FRANCISCA L, JOSEPH J H, WALTER M G. Development of tools for quantitative intracellular metabolomics of Aspergillus niger chemost at cultures [J]. Metabolomics, 2015, 11(5): 1253-1264.
[34]SEIFAR R M, ZHAO Zheng, VAN DAM J, et al. Quantitative analysis of metabolites in complex biological samples using ion-pair reversed-phase liquid chromatography-isotope dilution tandem mass spectrometry [J]. Journal of Chromatography A, 2008, 1187(1/2): 103-110.
[35]JULIANA L, THOMAS P, LAURENT D, et al. Putative metabolic pathway for the bioproduction of bikaverin and intermediates thereof in the wild Fusarium oxysporum LCP531 strain [J/OL]. AMB Express, 2019, 9(1): 186. DOI: 10.1186/s13568-019-0912-4.
[36]CHOI J N, KIM J, JUNG W H, et al. Influence of iron regulation on the metabolome of Cryptococcus neoformans [J/OL]. PLoS ONE, 2012, 7(7): e41654. DOI: 10.1371/journal.pone.0041654.
[37]VILLAS B, SILSA G. BRUHEIM P. Cold glycerol-saline: the promising quenching solution for accurate intracellular metabolite analysis of microbial cells [J]. Analytical Biochemistry, 2007, 370(1): 87-97.
[38]李娟, 任路靜, 孫冠男, 等. 氣相色譜-質譜聯用技術及其在代謝組學中的應用[J]. 生物工程學報, 2013, 29(4): 434-446.
[39]謝華里, 李培武, 王秀嬪, 等. 非靶標代謝組研究溫度對黃曲霉菌代謝的影響[J]. 分析測試學報, 2017, 36(11): 1304-1311.
[40]MATTHEW A L, CHARLES F B, ROBERT T K. Reducing time and increasing sensitivity in sample preparation for adherent mammalian cell metabolomics [J]. Analytical Chemistry, 2011, 83(9): 3406-3414.
[41]SOOAH K, DO Y L, GERT W, et al. Evaluation and optimization of metabolome sample preparation methods for Saccharomyces cerevisiae [J]. Analytical Chemistry, 2013, 85(4): 2169-2176.
[42]UDAYKUMAR K, SHIVAPPA H, AJJAMADA C K. Liquid chromatography and high-resolution mass spectrometry-based metabolomics to identify quantitative resistance-related metabolites and genes in wheat QTL-2DL against Fusarium head blight [J]. European Journal of Plant Pathology, 2018, 151(1): 125-139.
[43]ROHAN L, MLANIE J, GAIL C, et al. The induction of mycotoxins by trichothecene producing Fusarium species [J]. Plant Fungal Pathogens: Methods and Protocols, 2012, 835: 439-455.
[44]ZHENG Xiaomei, YU Jiandong, CAIRNS T C, et al. Comprehensive improvement of sample preparation methodologies facilitates dynamic metabolomics of Aspergillus niger [J/OL]. Biotechnology Journal, 2018, 14(3): 1800315. DOI: 10.1002/biot.201800315.
[45]ZABEK A, JUNKA A, SZYMCZYK P, et al. Metabolomics analysis of fungal biofilm development and of arachidonic acid-based quorum sensing mechanism [J]. Journal of Basic Microbiology, 2017, 57(5): 428-439.
[46]LOWE R G T, ALLWOOD J W, GALSTER A M, et al. A combined 1h nuclear magnetic resonance and electrospray ionization-mass spectrometry analysis to understand the basal metabolism of plant-pathogenic Fusarium spp. [J]. Molecular Plant Microbe Interact, 2010, 23(12): 1605-1618.
[47]CHRISTOPH J B, PATRICK K, FABIEN L, et al. Sampling for metabolome analysis of microorganisms [J]. Analytical Chemistry, 2007, 79(10): 3843-3849.
[48]TOMAS C, MARTA V, ZBYNEK D, et al. Rapid LC-MS-based metabolomics method to study the Fusarium infection of barley [J]. Journal of Separation Science, 2014, 37(8): 912-919.
[49]SHEN Yang, FATEMEH T, TANG Leihan, et al. Quantitative metabolic network profiling of Escherichia coli: an overview of analytical methods for measurement of intracellular metabolites [J]. Trends in Analytical Chemistry, 2016, 75: 141-150.
[50]PHUONG A N, CAROLINE S, MARIE L, et al. Study of in vitro interaction between Fusarium verticillioides and Streptomyces sp. using metabolomics [J]. Folia Microbiologica, 2020, 65(2): 303-314.
[51]LIU Caixiang, CHEN Fangfang, ZHANG Jingtao, et al. Metabolic changes of Fusarium graminearum induced by TPS gene deletion [J]. Journal of Proteome Research, 2019, 18(9): 3317-3327.
[52]KARPE A V, DUNN M S, TAYLOR M C T. et al. Nitrogen deprivation in Fusarium oxysporum promotes mycotoxin production via intermediates in the Krebs cycle and unreported methylmalonyl-CoA mutase activity [J/OL]. Metabolomics, 2018, 14(12): 160. DOI: 10.1007/s11306-018-1459-0.
[53]趙春霞, 許國旺. 基于液相色譜-質譜技術的代謝組學分析方法新進展[J]. 分析科學學報, 2014, 30(5): 761-766.
[54]CHEN Wei, GONG Liang, GUO Zilong, et al. A novel integrated method for large-scale detection, identification, and quantification of widely targeted metabolites: application in the study of rice metabolomics [J]. Molecular Plant, 2013, 6(6): 1769-1780.
[55]WOLFENDER J L, MARTI G, THOMAS A, et al. Current approaches and challenges for the metabolite profiling of complex natural extracts [J]. Journal of Chromatography A, 2015, 1382: 136-164.
[56]HAUTBERGUE T, JAMIN E L, DEBRAUWER L, et al. From genomics to metabolomics, moving toward an integrated strategy for the discovery of fungal secondary metabolites [J]. Natural Product Reports, 2018, 35(2): 147-173.
[57]SEVASTOSA A, KALAMPOKISA I F, PANAGIOTOPOULOUB A, et al. Implication of Fusarium graminearum primary metabolism in its resistance to benzimidazole fungicides as revealed by 1H NMR metabolomics [J]. Pesticide Biochemistry and Physiology, 2018, 148: 50-61.
[58]ISSAQ H J, VAN Q N, WAYBRIGHT T J, et al. Analytical and statistical approaches to metabolomics research [J]. Journal of Separation Science, 2015, 32(13): 2183-2199.
[59]RODRIGUES A M, MIGUE C, CHAVES I, et al. Mass spectrometry‐based forest tree metabolomics [J]. Mass Spectrometry Reviews, 2019, 47(2): 126-157.
[60]ZHAO Liang, THOMAS H. Metabonomics and toxicology [J].? Methods in Molecular Biology, 2015, 1277: 209-231.
[61]LIU Xinyu, ZHOU Lina, SHI Xianzhe, et al. New advances in analytical methods for mass spectrometry-based large-scale metabolomics study [J/OL]. Trends in Analytical Chemistry, 2019, 121: 115665. DOI: 10.1016/j.trac.2019.115665.
[62]CAJKA T, FIEHN O. Increasing lipidomic coverage by selecting optimal mobile-phase modifiers in LC-MS of blood plasma [J/OL]. Metabolomics, 2016, 12(2): 34. DOI: DOI 10.1007/s11306-015-0929-x.
[63]LI Yong, RUAN Qiang, LI Yanli, et al. A novel approach to transforming a non-targeted metabolic profiling method to a pseudo-targeted method using the retention time locking gas chromatography/mass spectrometry-selected ions monitoring [J]. Journal of Chromatography A, 2012, 1255: 228-236.
[64]TSUGAWA H, TSUJIMOTO Y, SUGITATE K, et al. Highly sensitive and selective analysis of widely targeted metabolomics using gas chromatography/triple-quadrupole mass spectrometry [J]. Journal of Bioscience and Bioengineering, 2014, 117(1): 122-128.
[65]HALKET J M, WATERMAN D, PRZYBOROWSKA A M, et al. Chemical derivatization and mass spectral libraries in metabolic profiling by GC/MS and LC/MS/MS [J/OL]. Journal of Experimental Botany, 2005, 56(410): 219. DOI: 10.1093/jxb/eri069.
[66]WEN Jing, YANG Lina, QIN Feng, et al. An integrative UHPLC-MS/MS untargeted metabonomics combined with quantitative analysis of the therapeutic mechanism of Si-Ni-San [J]. Analytical Biochemistry, 2019, 567: 128-135.
[67]MARINA G, BASEM K, FRANK S, et al. Comprehensive analysis of the Alternaria mycobolome using mass spectrometry-based metabolomics [J/OL]. Molecular Nutrition Food Research, 2020, 64(3): 1900558. DOI: 10.1002/mnfr.201900558.
[68]ZHOU Juntuo, LIU Huiying, LIU Yang, et al. Development and evaluation of a parallel reaction monitoring strategy for large-scale targeted metabolomics quantification [J]. Analytical Chemistry, 2016, 88(8): 4478-4486.
[69]FERNAND M G, ROULLIER C, GUITTON Y, et al. Fungi isolated from Madagascar shrimps-investigation of the Aspergillus niger metabolism by combined LC-MS and NMR metabolomics studies [J]. Aquaculture, 2017, 479: 750-758.
[70]GOODACRE R. Metabolomics of a superorganism [J]. Journal of Nutrition, 2007, 137: 259S-266S.
[71]LIU Xiaojing, LOCASALE J W. Metabolomics: A primer [J]. Trends in Biochemical Sciences, 2017, 42(4): 274-284.
[72]KOUSKOUMVEKAKI I, YANG Zhiyong, JNSDTTIR S O, et al. Identification of biomarkers for genotyping Aspergilli using non-linear methods for clustering and classification [J/OL]. BMC Bioinformatics, 2008, 9: 59. DOI: 10.1186/1471-2105-9-59.
[73]MASTRANGELO A, FERRARINI A, REY S F, et al. From sample treatment to biomarker discovery: A tutorial for untargeted metabolomics based on GC-(EI)-Q-MS [J]. Analytica Chimica Acta, 2015, 900: 21-35.
[74]TAYLOR J, KINGR D, ALTMANN T, et al. Application of metabolomics to plant genotype discrimination using statistics and machine learning [J]. Bioinformatics, 2002, 18(S2): S241-S248.
[75]SEVASTO A, KALAMPOKIS I F, PANAGIOTOPOULOU A, et al. Fusarium graminearum 1H NMR metabolomics [J]. Data in Brief, 2018, 19: 1162-1165.
[76]HUANG Baoming, ZHA Qinglin, CHEN Tingbo, et al. Discovery of markers for discriminating the age of cultivated ginseng by using UHPLC-QTOF/MS coupled with OPLS-DA [J]. Phytomedicine, 2018, 45: 8-17.
[77]俞邱豪, 張九凱, 葉興乾, 等. 基于代謝組學的食品真實屬性鑒別研究進展[J]. 色譜, 2016, 34(7): 657-664.
[78]KALIVODOV A, HRON K, FILZMOSER P, et al. PLS-DA for compositional data with application to metabolomics [J]. Journal of Chemometrics, 2015, 29(1): 21-28.
[79]王清利. 基于UHPLC-MS/MS技術的細胞代謝組學方法的建立及應用[D]. 鄭州:鄭州大學, 2018.
[80]GU Qiongnan, YUAN Qinfeng, ZHAO Dian, et al. Acetyl-coenzyme A synthetase gene ChAcs1 is essential in lipid metabolism, carbon utilization and virulence of the hemibiotrophic fungus Colletotrichum higginsianum [J]. Molecular Plant Pathology, 2019, 20(1): 107-123.
[81]CAO Huijuan, HUANG Pengyun, YAN Yuxin, et al. The basic helix-loop-helix transcription factor Crf1 is required for development and pathogenicity of the rice blast fungus by regulating carbohydrate and lipid metabolism [J]. Environmental Microbiology, 2018, 20(9): 3427-3441.
[82]BRISSON V L, ZHUANG Weiqin, ALVAREZ-COHEN L. Metabolomic analysis reveals contributions of citric and citramalic acids to rare earth bioleaching by a Paecilomyces fungus [J/OL]. Frontiers in Microbiology, 2020, 10: 3008. DOI: 10.3389/fmicb.2019.03008.
[83]AGUILAR-PONTES M V, BRAND J, MCDONNELL E, et al. The gold-standard genome of Aspergillus niger NRRL 3 enables a detailed view of the diversity of sugar catabolism in fungi [J]. Studies in Mycology, 2018, 91: 61-78.
(責任編輯:楊明麗)

