基于高通量轉錄組測序技術的龍頭魚微衛星信息分析
黃新芯,蔣艷琳,蔣小姿,楊天燕
(浙江海洋大學水產學院,浙江舟山 316022)
微衛星DNA(Microsatellite DNA)又稱為簡單重復序列(simplesequence repeat,SSR),是生物基因組,尤其是高等生物基因組中廣泛分布的以1~6 個核苷酸為基元,首尾串聯而成的重復序列[1],具有遺傳變異水平高、重復序列多、數量豐富、呈共顯性遺傳、引物具有通用性等特點[2-4]。微衛星最早發現于人類基因組中[5],有關魚類微衛星的研究是從虹鱒Oncorhynchus mykiss 開始的,而后大西洋鮭Salmo salar、大西洋鱈魚Gadus morhua、羅非魚Oreochromis niloticus 等多種魚類微衛星位點也得到了開發[6]。發展至今,高度多態性的微衛星分子標記技術已十分成熟[7],在多種水產動物群體遺傳結構和遺傳多樣性、遺傳連鎖圖譜構建,系統發育和親緣關系鑒定等研究領域得到了廣泛應用[8]。
龍頭魚Harpadon nehereus 俗稱豆腐魚、水潺或狗母魚,隸屬于是燈籠魚目Myctophiformes,龍頭魚科Harpadontidae,龍頭魚屬Harpadon[9],是廣泛分布于印度洋和西太平洋沿岸水域的中下層魚類,在我國主要集中于黃海南部、東海以及南海河口[10]。20 世紀80 年代以來,以龍頭魚為代表的中小型魚類生物量呈迅猛增加趨勢,已成為我國近海漁業優勢種,經濟地位和生態價值也得到提升[11]。然而近年來,由于海洋生態環境破壞和捕撈壓力的增大,包括孟加拉灣以及我國東海在內的部分海區龍頭魚群體逐漸呈現出低齡化、小型化趨勢[12-14]。如何保護與合理開發利用龍頭魚生物資源已成為亟待解決的現實問題。
已有的研究工作主要立足于通過種群的動態監測和生物學特征的測定來反映龍頭魚資源狀況,從群體遺傳學角度揭示其遺傳多樣性水平的研究報道則多以線粒體基因組序列分析為主。李海燕等[15]和XU T J,et al[16]曾采用磁珠富集法開發龍頭魚微衛星標記,但由于操作步驟繁瑣、技術難度大,開發微衛星位點數量有限,難以滿足進一步研究需求。近年來,隨著以Roche 公司的454 技術、Illumina 公司的Solexa、Hiseq 技術和LifeTechnologies 公司的Solid 技術為代表的二代高通量測序技術的發展[17],多種海洋生物微衛星分子標記的大規模開發得以實現,然而至今未見到采用高通量測序數據開發龍頭魚微衛星分子標記的相關報道。本研究采用新一代Illumina 高通量測序技術對龍頭魚肌肉組織轉錄組進行測定分析,挖掘微衛星序列信息,探討其分布、組成和長度等特征,以期為龍頭魚及其近緣物種多態性分子標記的有效開發及遺傳多樣性研究提供基礎信息,為其資源的合理開發與可持續利用政策的制定提供遺傳學依據。
1 材料與方法
1.1 實驗材料
用于轉錄組測序的龍頭魚樣品于2020 年5 月采自浙江舟山近海。剪取適量樣品背部肌肉裝入含有RNAhold 保存液的凍存管中保存,送至上海生工生物工程股份有限公司進行cDNA 文庫構建和基于Illumina HiseqTM2500 平臺的轉錄組測序。
1.2 RNA 測序數據的拼接及組裝
測序獲得的龍頭魚樣本轉錄組Raw data 經FastQC 和Trimmomatic[18]軟件進行質量評估和剪切,去除接頭、樣品標識序列、低質量reads 以及帶N 堿基較多的reads,獲取Clean data。使用Trinity 軟件[19]對純凈讀數進行De nove 組裝成Transcript,去冗余取每個轉錄本聚類中最長的轉錄本作為Unigene,用于后續分析研究。
1.3 SSR 位點篩選
使用Micro-Satellite(MISA)[20]軟件對龍頭魚轉錄組Unigenes 中潛在的微衛星序列(SSR)進行搜索,設置篩選條件為:基元長度1~6 bp,單堿基重復次數≥10,二堿基重復次數≥6,三、四、五、六堿基重復次數≥5。若2 個SSR 之間的距離小于100 bp,記為1 個復合微衛星[21]。
1.4 數據分析
使用Excel 軟件統計SSR的數量、發生頻率、出現頻率、分布距離與密度、重復類型、重復區段長度變化、重復基元類別。在微衛星初始統計時,保持其原始重復類別,不進行歸類,如將G 和C 單獨統計,而在討論部分為與他人結果進行比較,考慮堿基互補配對的原則。通過以上數據的統計整理,分析龍頭魚轉錄組微衛星序列的分布特征。
SSR 發生頻率=含SSR的Unigenes 總數/ Unigenes 總數
SSR 相對豐度=篩選獲得的SSR 總數/轉錄組Unigenes 總長度
SSR 平均距離=Unigenes 總長度/篩選獲得的SSR 總數
SSR 出現頻率=檢測所得SSR 總數/ Unigenes 總數
2 結果與分析
2.1 轉錄組測序、拼接和組裝
基于Illumina HiseqTM2500 平臺測序,總計產出41 886 302 條Raw reads,經質控得到39 233 566 條Clean reads,共計5 584 743 187 個核苷酸,GC 含量為51.39%、N 堿基比例0.02%,Q20(質量值≥20的堿基數目所占百分比)為97.94%,Q30(堿基質量值≥30的堿基數目所占百分比)為91.58%。經Trinity 從頭組裝獲得35 859 條Transcripts,去冗余后得到29 756 條Unigenes,平均長度為570.45 bp,N50 為797 bp,N90為249 bp(表1)。長度大于1 Kb的Unigens 有4 120 條,占總Unigenes的13.84%,大于500 bp的有9 634條,占比32.38%。表明,本研究中轉錄組文庫的測序和組裝結果較好,可以進行后續生物學研究。

表1 轉錄組數據組裝結果統計表Tab.1 Statistical table of transcriptome data assembly of H.nehereus
2.2 龍頭魚轉錄組SSR 數量及分布
使用MISA 軟件對總長16 974 320 bp的29 756 條Unigenes 序列進行篩選,發現共有6 023 個SSR 位點分布在5 085 條Unigens 中,發生頻率為17.1%,其中4 215 條Unigenes 含有單一的SSR 位點,約占序列總數的83%。去除以復合形式存在的551 個SSR 位點,獲得6 類完美型SSR 共5 652 個,總長度86 517 bp,相對豐度為332.97 個/Mb,即平均每3 Kb 出現1 個SSR 位點。在完美型SSR 位點中,67.59%為單堿基重復類型(3 820 個),二堿基重復類型(1 171 個)占比20.72%,三、四、五、六堿基重復類型相對較少,僅11.69%,發生頻率、相對豐度、總長度也呈現出同樣的趨勢(表2)。
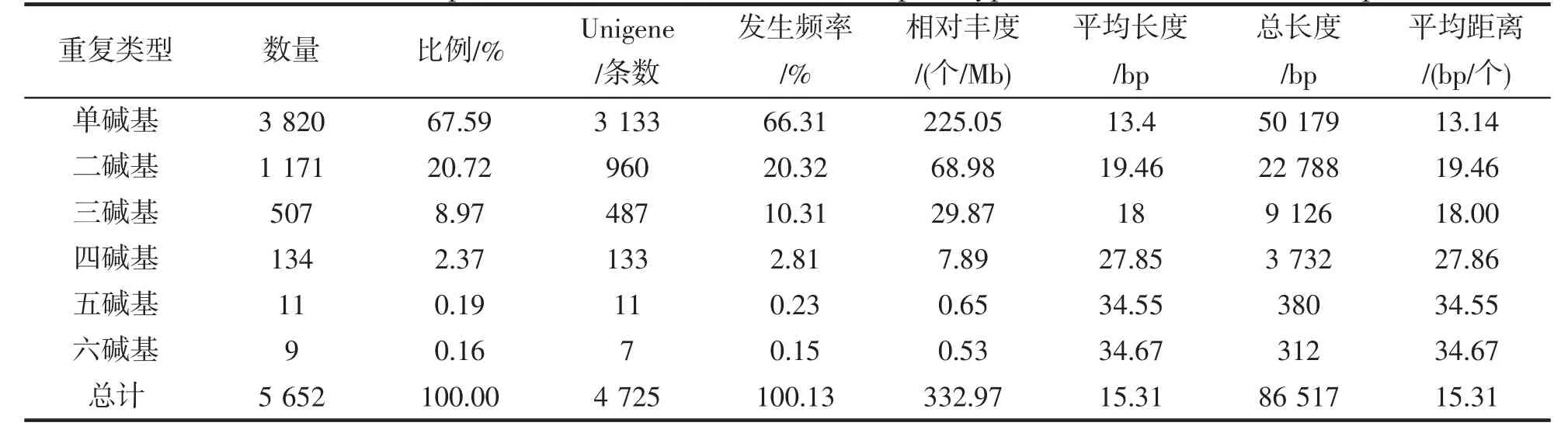
表2 龍頭魚轉錄組不同重復類型完美型微衛星位點分布特征Tab.2 Distribution of perfect SSR loci with different repeat types in H.nehereus transcriptome
2.3 龍頭魚轉錄組SSR 重復基元類型
龍頭魚轉錄組微衛星中共檢測出重復基元148 種,其中三、四堿基重復基元種類最為豐富,分別有51、63 種,單堿基受堿基數量的限制,重復基元種類最少,僅有4 種,但在數量上占據了絕對優勢,其中A、T 重復基元占到了總SSR 數量的65.20%,相對而言五、六堿基重復基元種類與數量分布均較少(表3)。從基元在各自重復類型中的含量來看,A 在單堿基重復中最多,達到了1 848 個,C 最少為54 個;二堿基重復以GT(16.74%),TG(16.57%),AC(16.23%),CA(12.98%)為主;三堿基重復中,占比較高的有GAG(5.72%),GAT(4.93%),TTG(4.54%),AAT(4.34%),CCT(4.34%),TTA(3.94%);四堿基重復出現頻率較多的是TTTG(8.21%),AAAC(7.46%),CTGT(5.22%);五、六堿各重復類型數量分布較為均勻,無明顯優勢重復基元(圖1)。
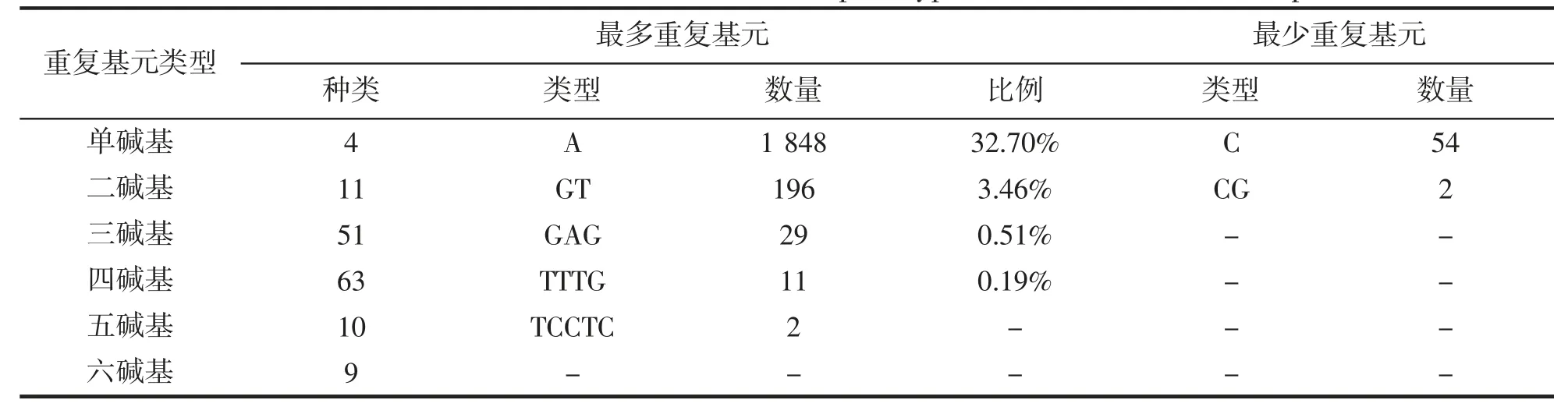
表3 龍頭魚轉錄組SSR 中優勢重復堿基類別Tab.3 Dominant base classes in each base repeat type in H.nehereus transcriptome
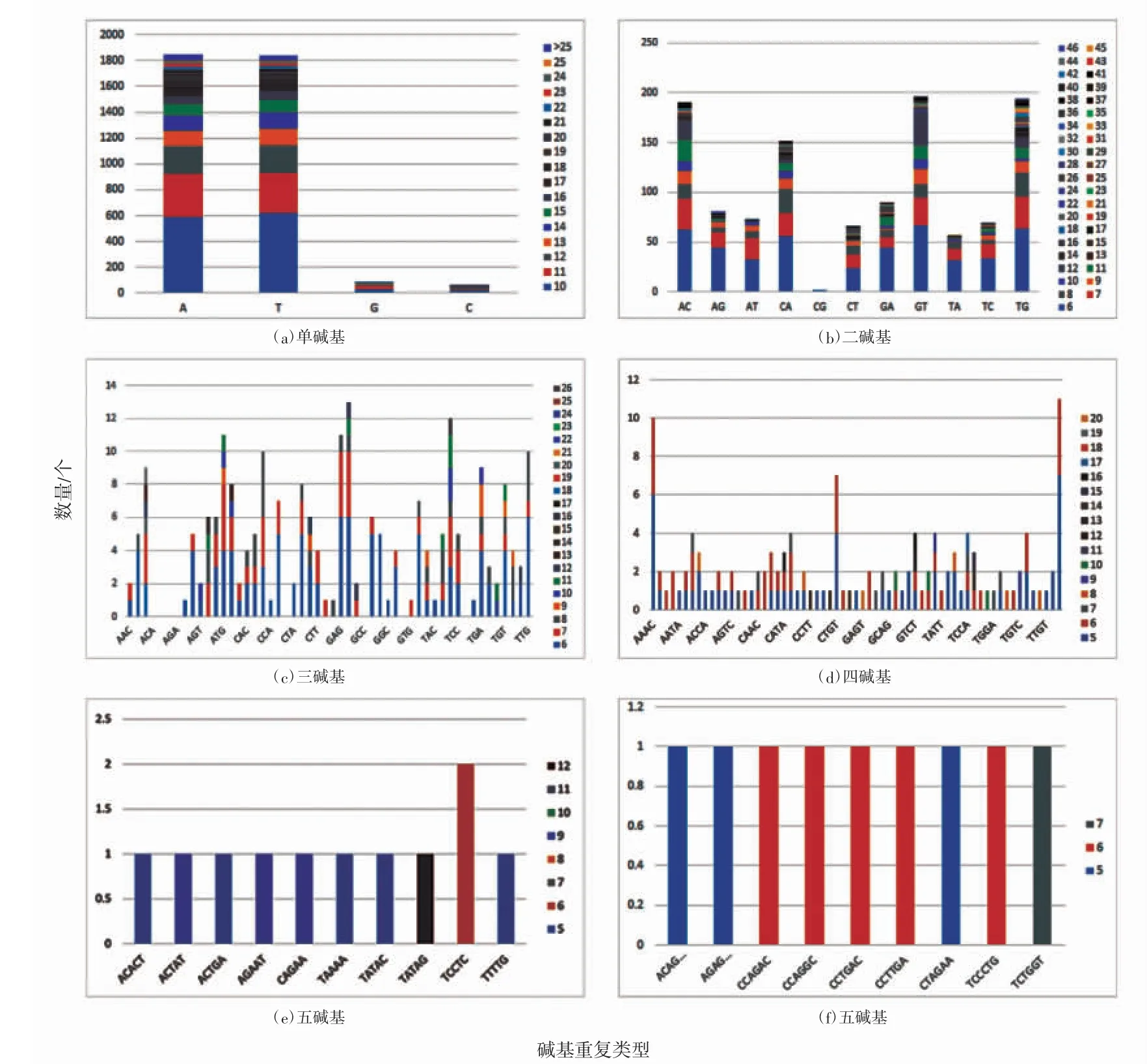
圖1 龍頭魚轉錄組中各類型微衛星重復數分布Fig.1 Distribution of different copy numbers of various types of microsatellites in H.nehereus transcriptome
2.4 龍頭魚轉錄組SSR 序列重復次數分布
龍頭魚轉錄組微衛星位點中,10 次重復的SSR 數量最多,達1 313 個,占SSR 總數的23.23%;其次是6,11,12 次重復,位點數量均在500~800 個之間,約占總SSR 數目的33.78%(圖2)。統計表明,單堿基重復次數分布在6~58 次,其中11~15 次最多,占單堿基總數的47.70%;二堿基重復拷貝數在6~46 次之間,以6~10 次重復居多,共894 個,達到了該類型核苷酸總數的76.34%;三、四堿基重復次數分別分布在5~26次和5~20 次之間,尤其是5~10 次重復,分別占其重復類型總數的96.45%、88.81%;五、六堿基基元重復次數均小于16 次,以5~10 次居多(表4)。
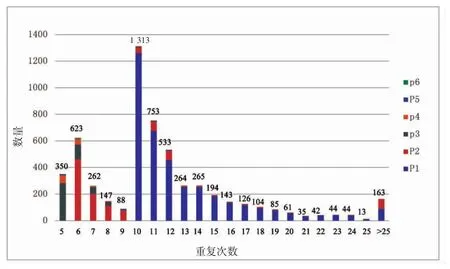
圖2 龍頭魚轉錄組中SSR 重復次數分布Fig.2 Distribution of the repeats number of SSR repeats in H.nehereus transcriptome
總的來說,該研究中SSR基元重復次數主要分布在6~10 次,對應的SSR 位點共2 431 個,占總SSR 數目的43.01%;其次是11~15 次,共2 009 個,占比33.54%;重復次數大于25 次的位點相對較少,主要由單堿基和二堿基組成,共163 個,占SSR總數的2.89%(表4)。此外,當SSR 重復次數達到了10 次,隨著重復次數的增加其位點總數呈顯出遞減的趨勢,且單堿基重復所占的比例逐漸增大。
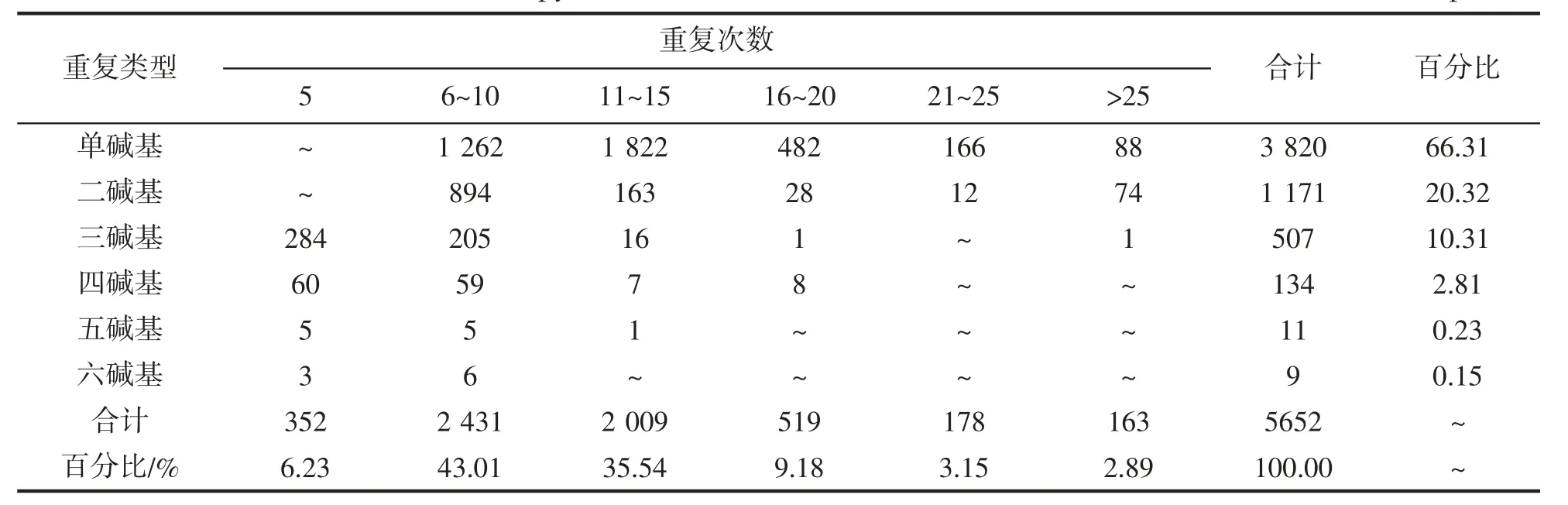
表4 龍頭魚轉錄組不同類型微衛星重復次數區間分布特征Tab.4 Distribution interval of the copy number in different microsatellite motifs for H.nehereus transcriptome
2.5 龍頭魚轉錄組SSR 長度分布及多態性評價
龍頭魚微衛星序列長度區間跨度較大,范圍在10~92 bp,其中六堿基重復序列長度變化最小在30~42 bp 之間;二堿基重復序列長度范圍最大,為12~92 bp;單、三、四、五、核苷酸重復序列長度范圍分別為10~58、15~78、20~80 和25~60 bp。微衛星的多態性源于等位基因間重復單位數的不同導致的序列長度差異,根據長度的不同可將微衛星分為2 類:具有高度多態性的Ⅰ型(重復序列長度大于等于20 bp)和中度多態性Ⅱ型(重復序列長度在12~20 bp 之間),當重復序列長度小于12 bp 時多態性較低[22]。Ⅰ與Ⅱ型微衛星作為分子標記的潛在可用性較高[23]。在本研究中,Ⅰ型SSR 共896 條,占SSR 總數的15.85%,Ⅱ型SSR 有2 819條,占SSR 總數的49.88%(圖3)。
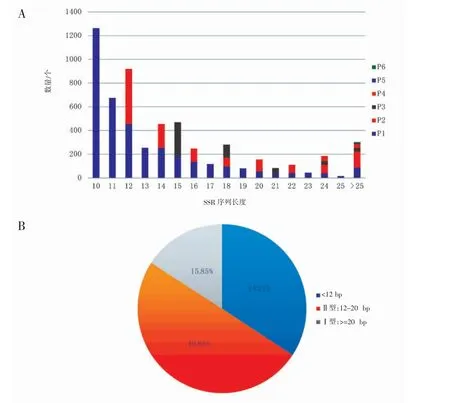
圖3 龍頭魚轉錄SSR 長度分布特征Fig.3 Length distribution of transcriptomic SSR in H.nehereus
3 討論
近年來,隨著轉錄組測序技術在非模式生物遺傳學研究中的廣泛應用,基于轉錄組數據挖掘微衛星位點逐漸成為微衛星標記開發的重要手段之一[24]。ZHENG Jian,et al[25]通過轉錄組測序開發得到了7 490 個小黃魚Larimichthys polyactis SSR 標記;SONG Chenyu,et al[26]從斑尾刺蝦虎魚Acanthogobius ommaturus 測序數據中篩選出4 756 個SSR 位點。而目前有關龍頭魚的相關研究報道較少。本研究對龍頭魚肌肉組織進行轉錄組測序,從組裝所得序列中鑒定出大量SSR 位點。相較于XU T J,et al[16]采用鏈霉親和素磁珠吸附這種傳統方式開發龍頭魚微衛星,轉錄組測序經濟、快捷且高效。同時,由于轉錄本具有較高的保守性,通過轉錄組開發得到的微衛星在近緣物種中具有更高的通用性[27]。此外,來源于轉錄組的微衛星標記具有特異性并與某些功能基因緊密連鎖,可為后續遺傳圖譜構建、QTL 定位等奠定基礎。
本研究通過對龍頭魚轉錄組進行SSR 位點分析,在29 756 條Unigenes 中共識別位點5 652 個,出現頻率為18.99%,與江鱈Lota lota(16.61%)[28]和缺須盆唇魚Placocheilus cryptonemus(12.08%)[29]轉錄組微衛星數據相比,龍頭魚轉錄組中SSR 出現頻率較高,但低于銀鯧Pampus argenteus(28.49%)[23],總體來說,龍頭魚轉錄組中SSR 數量保持在較為豐富的水平。從相對豐度來看,龍頭魚(0.333 SSR/Kb)遠大于大口黑鱸Micropterus salmoides(0.153 SSR/Kb)[30]、雙須骨舌魚Osteoglossum bicirrhosum(0.101 SSR/Kb)[31],低于黃唇魚Bahaba flavolabiata(0.429 SSR/Kb)[32]。造成這種差異的原因可能與物種間差異、轉錄組結構、檢測標準、搜索軟件選擇以及SSR 位點所在基因的表達豐度有關[33]。此外,盡管本研究未采用多組織混合樣本,無法進行基因差異化表達研究,但從微衛星標記開發角度來看,轉錄組數據質量及微衛星含量均處在中上水平,能夠為分子標記開發及遺傳學研究提供充足的序列資源。
所獲得的龍頭魚轉錄組中不同類型的SSR 豐度差異較大,其中單堿基重復為最主要的重復類型。有研究推測,多數脊椎動物(包括魚類)基因組微衛星中單堿基重復豐度最高,而在轉錄組中似乎也有同樣的趨勢,這可能與微衛星序列越長,突變率就越高,因此穩定性就越差有關[34]。由于單堿基重復出現極低多態性位點的可能性相對較大[35],為提高后續分子標記開發的可用性,在對重復堿基類型進行統計時可將其扣除,則余下的微衛星以二堿基重復居多,其次是三堿基重復。一般認為,重復基元的含量與物種進化水平的高低有緊密的聯系,低級重復基元出現頻率高的物種進化水平較高,而高級重復基元的大量存在表明了進化時間較短或變異頻率較低,即高級基元微衛星多態性普遍比低級基元低[36]。龍頭魚中1~3 堿基重復類型約占總SSR的97.28%,表明其可能具有較長的進化歷史較長或較高的變異頻率,開發潛力較高;4~6 堿基重類型相對較少,與團頭魴Megalobrama amblycephala[37]等研究結果相似。
龍頭魚二堿基重復以GT/AC 基元為主,與牙鲆P.olivaceus[38]一致。相對來說,GC 含量十分稀少,這一特點在翹嘴鱖Siniperca chuatsi[39]、草魚Ctenopharyngodon idella[40]、紅鰭東方鲀Takifugu rubripes[41]等水生生物轉錄組微衛星序列中均有體現。已有研究表明在脊椎動物基因組中GC 重復類型較為罕見,這可能是基因組DNA 由于CpG 甲基化,胞苷酸C 易通過脫氨基作用轉變為胸腺嘧啶T[42]。值得注意的是,三堿基重復類型中AGG/CCT 含量較為豐富,脊椎動物和木本植物均有類似報道,但在魚類研究中并不常見[43]。AGG與生物早期生長和發育密切相關,加之三堿基重復在某些遺傳疾病控制上的特殊作用,推測AGG的高頻率分布可能有助于龍頭魚早期生長調控以及控制致病基因的表達[44]。在單、四堿基重復中A/T、TTTG/CAAA 和AAAC/GTTT 基元含量豐富,體現出一定的A/T 優勢,也在一定程度上對GC 含量的稀少起到了支撐作用。總的來說,SSR 各堿基類型的優勢基元分布在不同物種間存在差異,但仍然保留了一定程度的相似性。
分子標記的性能優劣及可用性主要通過其多態性來評價,微衛星憑借其豐富的長度多態性,成為了一種理想的分子標記[45]。目前認可度較高的有關SSR 多態性形成機制主要有2 種:一種是DNA 復制過程中聚合酶的滑移造成重復次數的差異[46];另一種是在減速分裂過程中,同源染色體及非姐妹染色單體的不對等交換和重組[47]。微衛星多態性在基元特征上主要表現為重復次數的不同以及堿基數不同形成的片段長度差異。一般情況下,隨著重復次數的增加,微衛星序列的變異性也會增加,其多態性潛力也就越大[33]。從重復次數來看,龍頭魚微衛星基元重復次數在5~58 之間,主要集中在5~25 次,若不考慮容易發生錯配的單堿基重復類型,重復數最高達到46 次。從片段長度來看,超過60%的SSR 位點具有中度及以上水平的多樣性,表明龍頭魚轉錄組來源的SSR 具有較高的多態性潛能。
生物種群資源量的衰退常伴隨著遺傳多樣性水平的下降及遺傳結構的改變。龍頭魚作為我國重要的海水經濟魚類,其資源的保護與開發利用理應受到充分的關注。本研究通過高通量測序揭示龍頭魚轉錄組中SSR 分布特征和規律,提供了高效開發多態性微衛星標記的數據基礎。這些分子標記將應用于龍頭魚群體遺傳多樣性與遺傳結構研究,評估龍頭魚種質資源,探討不同地理群體的遺傳差異,闡明其系統地理格局,進而為龍頭魚漁業管理單元的劃分提供科學依據,促進龍頭魚資源的合理開發、利用與保護。同時由于微衛星側翼序列的保守性,經過篩選的SSR 標記有望用于其它親緣關系較近物種的研究。

