茶樹CLH基因家族的鑒定與轉錄調控研究及其在白化茶樹中的表達分析
王濤,王藝清,漆思雨,周喆,陳志丹,孫威江*
茶樹CLH基因家族的鑒定與轉錄調控研究及其在白化茶樹中的表達分析
王濤1,3,4,5,王藝清1,3,4,5,漆思雨1,3,4,5,周喆1,3,4,5,陳志丹2,3,4,5*,孫威江1,3,4,5*
1. 福建農林大學園藝學院,福建 福州 350002;2. 福建農林大學安溪茶學院,福建 泉州 362400;3. 福建省茶產業工程技術研究中心,福建 福州 350002;4. 海峽兩岸特色作物安全生產省部共建協同創新中心,福建 福州 350002;5. 福建省茶產業技術開發基地,福建 福州 350002
葉綠素酶(Chlorophyllase,CLH)是葉綠素降解過程中的關鍵酶,將葉綠素a脫去植醇,形成脫植基葉綠素a。以白化茶樹白雞冠新梢葉片為材料,克隆獲得3條基因cDNA全長序列,并進行生物信息學分析。結果表明,3條基因分布于2個亞家族,其蛋白質編碼區(Coding sequence,CDs)長度為894~975?bp,編碼氨基酸個數為297~324,蛋白質分子量為31.99~34.91?kDa,等電點為4.89~7.61,不穩定系數為38.94~48.24,其中CsCLH1.1和CsCLH1.2為不穩定蛋白,CsCLH2為穩定蛋白。Cell-PLoc亞細胞定位預測結果表明,3個CsCLHs蛋白均定位于葉綠體;而WolfPsort亞細胞定位預測結果顯示,CsCLH1.1和CsCLH1.2定位于細胞質,CsCLH2定位于葉綠體。遮陰和恢復光照處理下的qRT-PCR結果顯示,遮陰抑制白雞冠葉片的表達,光照誘導白雞冠葉片的表達。不同品種中表達模式分析表明,在白化葉中高表達。另外,酵母單雜交結果表明,可以與和啟動子結合。綜上所述,CsCLHs在白化茶樹葉片中可能參與葉綠素降解,在葉片白化過程中發揮重要作用,結果可為進一步探究茶樹CLH基因家族的功能及茶樹葉片白化機理提供參考。
茶樹;葉綠素酶;基因家族;表達差異
葉片白化是綠色植物葉綠素缺陷出現的表型[1-3],植物白化通常會造成減產和品質下降[4-5],但在白化茶樹的葉片中有氨基酸含量高,兒茶素、咖啡堿含量低的特點[6-7]。茶樹白化類型可劃分為光照敏感型和溫度敏感型,光照敏感型白化茶樹在強光下芽葉呈現黃白色,在遮陰處理后芽葉轉綠,同時葉綠素含量顯著上升[8-10],但恢復光照后,葉片葉綠素含量顯著下降[11],且顯著低于綠葉茶樹品種[12-13]。研究表明,茶樹葉綠素合成途徑關鍵基因的低表達與葉綠素降解途徑關鍵基因的高表達是造成茶樹芽葉白化的主要原因[11,14-15]。
葉綠素酶(Chlorophyllase,CLH)作為葉綠素降解途徑中的限速酶,參與葉綠素降解途徑的第一步,將葉綠素a脫去植醇(Phytol)[16]。研究表明,在花色葉片中,非綠色部分的葉綠素酶活性高于綠色部分[17],在葉片花色由淺至深的轉化過程中,葉綠素酶的活性也逐漸減小,表明葉綠素的含量與葉綠素酶活性緊密關聯[18]。CLH基因在擬南芥()[19]、草地早熟禾()[20]等植物中被相繼克隆,但前人對植物CLH的研究多集中于植物衰老和貯藏方面,目前關于CLH在白化茶樹中的作用機制尚無深入研究。
白化茶樹是優異、特異種質資源,研究其葉綠素降解過程中的分子機制,對于揭示白化茶樹體內的葉色調控及葉片發育調控機理等有重要的理論價值,同時在茶樹良種選育方面有重要的應用價值。因此,本研究以光照敏感型白化茶樹白雞冠為材料,克隆了3條的cDNA全長序列,并對其進行生物信息學分析和表達分析,初步研究遮陰與恢復光照處理下的表達差異,以及在不同白化茶樹品種間的表達模式,為深入研究茶樹白化的分子機制提供試驗基礎。
1 材料與方法
1.1 材料處理
以武夷星種質資源圃內的白雞冠為試驗材料,選取白雞冠茶樹白化表型最顯著的第二葉,用錫箔紙進行遮陰處理,處理于2018年9月進行。遮陰方法如下:取處理前未遮陰樣品為對照(NS),分別于遮陰處理1?d和3?d時取樣,依次標記為S1和S3;遮陰3?d取樣后去除錫箔紙開始恢復光照,分別在恢復光照1?d和3?d時取樣,標記為RL1和RL3。取樣標準為葉片生長發育狀況和形狀大小相似,所取樣品用液氮速凍后保存于–80℃冰箱,用于提取RNA。所有樣品均設置3個重復,由北京百邁客生物科技有限公司進行轉錄組測序。
以白雞冠、黃化品系、黃金芽、金光、千年雪、御金香、中黃1號和金牡丹為試驗材料,分別取其一芽二葉新梢,于液氮中速凍,用于不同白化品種中的表達量分析。并以鐵觀音為試驗材料,分別取其嫩葉、成熟葉、老葉、嫩莖、老莖、花苞、果實和根等組織器官,于液氮中速凍,用于的組織表達模式分析。
1.2 茶樹CsCLHs基因家族成員鑒定
在擬南芥數據庫TAIR(www.arabidopsis.org/index.jsp)下載擬南芥AtCLH1、AtCLH2的氨基酸序列,從TPIA數據庫下載茶樹基因組數據。用已鑒定的擬南芥AtCLH1、AtCLH2氨基酸序列作為種子序列,在TPIA上進行BLASTP對茶樹基因組進行序列比對搜索(E-value≤10-5),得到茶樹CsCLHs成員氨基酸序列,同時通過在線數據庫Pfam(pfam.xfam.org)下載Chlorophyllase結構域文件,通過SPDE軟件[21]HMMER Search功能進行比對,將比對出的序列通過NCBI CDD(www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)驗證候選蛋白結構域,以確定其含有Chlorophyllase(PF07224)完整保守結構域。
1.3 茶樹CsCLHs基因的克隆及測序
采用RNAprep Pure多糖多酚植物總RNA提取試劑盒[天根生化科技(北京)有限公司]提取白雞冠葉片總RNA,采用Nano Drop 2000c超微量分光光度計(美國Thermo Fisher公司)測定RNA濃度,2%瓊脂糖凝膠電泳檢測所得RNA的完整性。根據Promega goscriptTMreverse transcription system試劑盒說明書步驟進行cDNA反轉錄。利用Primer Premier 5軟件設計各個基因CDs區全長特異性引物(表1)。使用GeneStar公司的2×SuperStar Plus PCR Mix對各基因的CDs區全長進行擴增,對目的片段進行純化并連接至pEASY-Blunt Cloning載體(北京全式金生物技術有限公司),經菌落PCR驗證后送至生工生物工程(上海)股份有限公司測序。
1.4 茶樹CsCLHs蛋白質結構預測
通過ProtParam網站(web.expasy.org/protparam)對CsCLHs蛋白分子量大小、理論等電點等理化特征進行分析預測;通過Cell-PLoc 2.0(www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2)和Wolfpsort(wolfpsort.hgc.jp)對CsCLHs編碼的蛋白進行亞細胞定位預測;使用在線工具SignalP 5.0(www.cbs.dtu.dk/services/SignalP)對CsCLHs各蛋白的信號肽進行預測。通過TMHMM Server v.2.0(services.healthtech.dtu.dk/service.php?TMHMM-2.0)工具對CsCLHs蛋白的跨膜結構域進行預測;通過SOPMA(https://npsaprabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在線預測CsCLHs亞家族蛋白的二級結構,使用Phyre2(www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index)工具預測各蛋白的三級結構模型,并通過Ramachandran plot(http://mordred.bioc.cam.ac.uk/~rapper/rampage.php)對所得蛋白的三級結構模型進行評估。
1.5 茶樹CsCLHs基因家族進化樹構建
利用NCBI BLASTP(www.ncbi.nlm.nih.gov)下載已經報道的各植物CLHs蛋白序列,并通過MEGA 7.0軟件與茶樹CsCLHs氨基酸序列進行Clustal W多序列比對,多序列比對參數為默認值。隨后使用近鄰相接法(Neighbor-Joining)構建系統發生樹,其參數均為默認值。
1.6 茶樹CsCLHs基因家族保守基序與結構預測
利用Pfam在線數據庫(pfam.xfam.org)和NCBI CDD對茶樹CsCLHs家族蛋白結構進行預測。使用在線工具MEME(meme-suite.org/tools/meme)分別對茶樹CsCLHs氨基酸序列保守基序進行預測分析,基序數量設置為10個,其余參數均為默認值。從TPIA數據庫下載茶樹基因結構注釋文件gff3,通過TBtools軟件[22]對CsCLHs系統進化樹、基序分布以及內含子、外顯子分布進行可視化。
1.7 茶樹CsCLHs基因家族順式作用元件預測
在TPIA數據庫中下載茶樹基因組,通過TBtools軟件提取家族成員起始密碼子ATG上游2?000?bp序列,利用啟動子在線預測軟件PlantCARE(bioinformatics.psb.ugent.be/webtools/plantcare/html)進行順式作用元件預測,利用TBtools進行順式作用元件可視化處理。
1.8 茶樹CsCLHs上游調控轉錄因子預測及表達量測定
利用PlantTFDB數據庫(http://plantregmap.gao-lab.org/index.php)中Regulation prediction功能,輸入序列為ATG上游2?000?bp啟動子區域,以擬南芥為參考,預測上游調控轉錄因子,并通過TPIA數據庫BLASTP獲得擬南芥同源基因的茶樹基因序列號,基于轉錄組數據中FPKM值為參考,分析可調控轉錄因子在遮陰及恢復光照條件下的表達模式。
基于基因組和轉錄組數據,選取CSS0007067(BPC6)和CSS002729(CDF5)進行酵母單雜交驗證,根據和啟動子序列與BPC6和CDF5編碼區序列設計引物進行PCR擴增(表2)。對pAbAi載體和pGADT7載體分別進行雙酶切,采用ClonExpress Ⅱ One Step Cloning Kit(南京諾唯贊生物科技股份有限公司)構建pAbAi-Pro、pAbAi-Pro、pGADT7-BPC6和pGADT7-CDF5重組載體。將重組載體通過PGE/LiAc法轉化至酵母Y1H感受態中。為篩選出具有抑制Y1H(Pro)和Y1H(Pro)生長的AbA(金擔子素A)濃度,以pAbAi-P53為陽性對照,空載Y1H為陰性對照,分別在含有AbA(25、50、150、300、500?ng·mL-1)的SD/-URA培養基上培養3~5?d,觀察是否有菌落出現。確定AbA抑制濃度后,將pGADT7-BPC6和pGADT7-CDF5分別轉入Y1H(Pro)和Y1H(Pro)感受態中,得到酵母重組菌株Y1H(Pro+BPC6)、Y1H(Pro+CDF5)、Y1H(Pro+BPC6)和Y1H(Pro+CDF5),在SD/-Leu和SD/-Leu/AbA150培養基上培養3~5?d,以Y1H(Pro)和Y1H(Pro)菌株為陰性對照,觀察是否有菌落出現。
1.9 茶樹CsCLHs基因家族表達分析

表2 酵母單雜交試驗所用引物
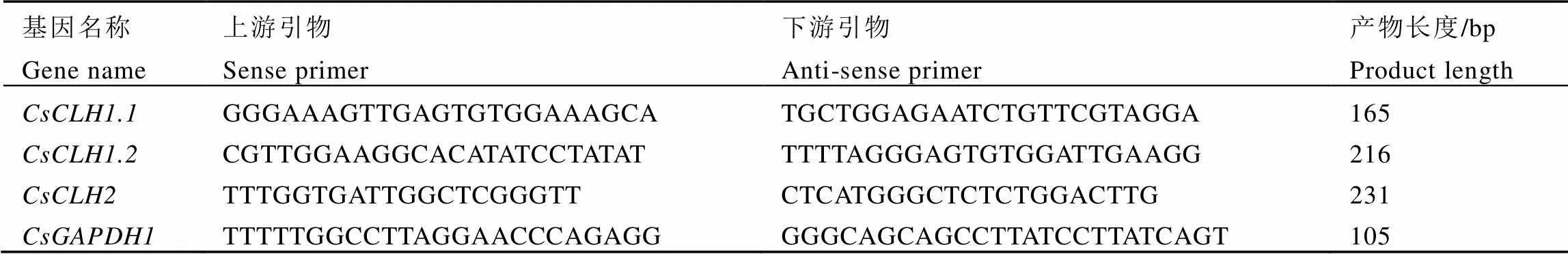
表3 實時熒光定量PCR引物序列信息
2 結果與分析
2.1 茶樹CsCLHs基因家族成員的鑒定與克隆
根據擬南芥AtCLH1和AtCLH2的蛋白序列,在茶樹基因組數據庫中得到5條茶樹基因,通過NCBI CDD蛋白質結構域在線預測網站對所獲得的蛋白質序列進行分析,只有3條蛋白序列包含完整的葉綠素酶結構域。將茶樹CLHs與葡萄、擬南芥等植物CLHs的蛋白質序列使用Clustal W進行多重比對,構建系統進化樹。結果顯示(圖1),茶樹與葡萄、擬南芥等植物CLHs聚類后可分為3組,其中CSS004684和CSS0033432與葡萄等CLH1聚為一類,CSS0028283與葡萄、擬南芥等CLH2聚為一類。根據進化樹聚類分析與在茶樹染色體的定位(圖2),將CSS004684、CSS0033432和CSS0028283分別命名為、和。以上述基因的基因組序列為參考,以白雞冠新梢葉片的cDNA為模板,通過RT-PCR的方式克隆得到CsCLHs家族的3條基因(圖3)。測序結果表明,茶樹中3條的CDs區長度為894~975?bp。
2.2 茶樹CsCLHs家族蛋白質特征分析
CsCLHs家族的氨基酸長度差異較小,在297~324個氨基酸(表4),蛋白質分子量為31.99~34.91?kDa,等電點為4.89~7.61,不穩定系數為38.94~48.24,其中CsCLH1.1和CsCLH1.2為不穩定蛋白,CsCLH2為穩定蛋白,CsCLHs蛋白脂肪族氨基酸含量較高,GRAVY值在–0.001~0.200,其中CsCLH1.1和CsCLH1.2為疏水性蛋白,CsCLH2為親水性蛋白。信號肽預測表明,CsCLHs均不含有信號肽。TMHMM跨膜結構分析表明,CsCLHs均不含有跨膜結構(表5)。
2.3 茶樹CsCLHs基因結構、染色體定位與保守基序分析
通過MEME在線工具對CsCLHs蛋白的10個保守基序進行預測分析,在CsCLHs各蛋白中基序3、基序2、基序4、基序1和基序5的排列順序較一致,CsCLH2缺少基序8(圖4-A),因此推測CsCLH1s和CsCLH2在葉綠體中所發揮的作用存在一定差異。通過NCBI CDD在線數據庫分析發現,茶樹CsCLHs蛋白都含有Chlorophyllase結構域(PLN00021),該結構域屬于cl21492/水解酶超家族(圖4-B)。CsCLH1.1、CsCLH1.2和CsCLH2蛋白成員的Chlorophyllase結構域分別分布在第1~300個氨基酸、第1~297個氨基酸和第21~324個氨基酸,各蛋白的保守結構域相對保守。外顯子-內含子結構分析表明,家族成員的外顯子和內含子數量存在差異,外顯子數量2~6個,無內含子(圖4-C)。
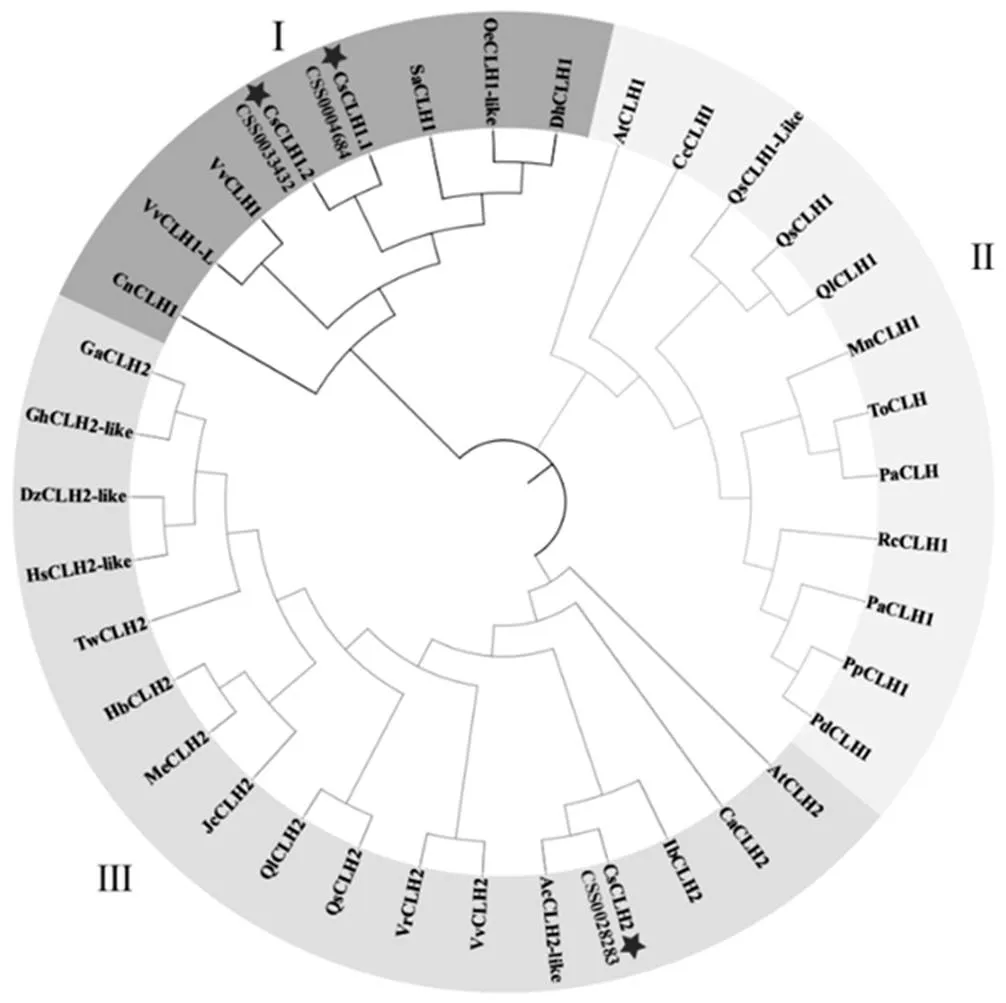
注:茶樹,CsCLH1.1,CsCLH1.2,CsCLH2;擬南芥,AtCLH1(NP_564094.1),AtCLH2(NP_199199.1);山谷櫟,QlCLH1(XP_030946150.1),QlCLH2(XP_030924067.1);東方苔草,ToCLH(PON83887.1);歐洲栓皮櫟,QsCLH1(XP_023917005.1),QsCLH1-like(XP_023873288.1),QsCLH2(XP_023900495.1);桃,PpCLH1(XP_007200272.1);扁桃,PdCLH1(XP_034228050.1);歐洲甜櫻桃,PaCLH1(XP_021821960.1);山黃麻,PaCLH(PON76085.1);月季,RcCLH1(XP_024162290.1);大麻,CsCLH(XP_030509823.1);桑,MnCLH(XP_010096444.1);海棗,PdCLH1(XP_008787534.2);旋蒴苣苔,DhCLH1(KZV50452.1);獨角金,SaCLH1(GER41106.1);木豆,CcCLH1(XP_020203352.1);椰子,CnCLH1(KAG1361310.1);葡萄,VvCLH1(RVX20904.1),VvCLH2(RVX22292.1);河岸葡萄,VrCLH1-like(XP_034690185.1),VrCLH2(XP_034684712.1);木樨欖,OeCLH1-like(XP_022879035.1);獼猴桃AcCLH2-like(PSS01417.1);橡膠,HbCLH2(XP_021691950.1);木薯,MeCLH2(XP_021608126.1);麻風樹,JcCLH2(XP_012069695.1);雷公藤,TwCLH2(XP_038705126.1);陸地棉,GhCLH2-like(XP_016699405.2);澳洲棉,GaCLH2(XP_016699405.2);甘薯,IbCLH2(AWK91225.1);小果咖啡,CaCLH2(XP_027116339.1)

圖2 CsCLHs家族基因在茶樹染色體分布圖

注:M:2?000?bp DNA marker

表4 CsCLHs鑒定及亞細胞定位
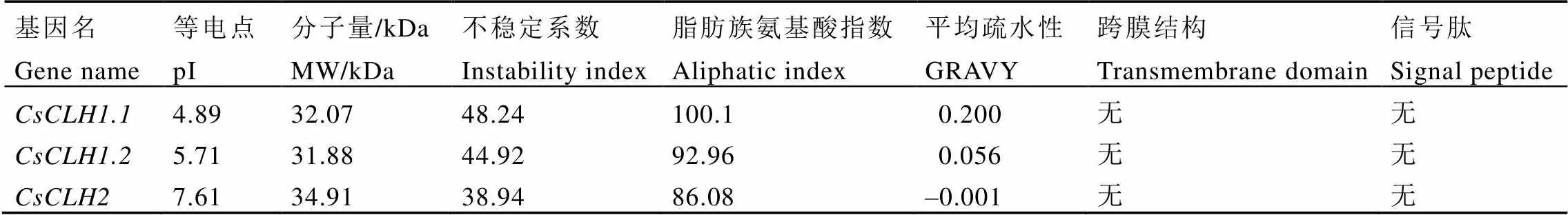
表5 CsCLHs理化性質
2.4 茶樹CsCLHs蛋白二級、三級結構分析
CsCLHs蛋白的二級結構預測結果表明(表6),3個蛋白均含有螺旋、轉角、無規則卷曲和延伸鏈。其中無規則卷曲是CsCLHs蛋白中的主要結構,占比在48.51%~50.93%;其次為螺旋和延伸鏈,分別占比24.38%~29.63%和16.50%~19.33%;轉角在CsCLHs蛋白的二級結構組成中占比最低。使用Phyre2在線工具的Intensive模式對CsCLHs蛋白的三級結構進行預測(圖5),預測所得到的CsCLHs蛋白結構與模板的覆蓋度均達到96%以上。通過Ramachandran在線評估蛋白質結構,結果顯示位于允許區域范圍內的氨基酸位點為93.2%~97.6%,表明所構建的CsCLHs蛋白的三級結構模型準確可信。
2.5 茶樹CsCLHs啟動子與順勢作用元件分析
從TPIA上下載上游2?000?bp序列,利用PlantCARE在線預測網站分析其順式作用元件。結果表明(圖6),除了具有典型核心啟動子CAAT-box、TATA-box外,還包含大量的光響應元件、激素響應元件、逆境響應元件及較少的組織特異性元件和MYB識別位點。激素類響應元件主要包括脫落酸、水楊酸、赤霉素、乙烯、茉莉酸甲酯和生長素,其中均含有脫落酸響應元件,生長素響應元件、茉莉酸甲酯響應元件(as-1、TGACG-motif和CGTCA-motif)和赤霉素響應元件(TATC-box)只分布于和,茉莉酸響應元件(TCA元件)和乙烯響應元件(ERE元件)只分布于和;非生物脅迫響應元件中,干旱誘導響應元件(DRE core)、缺氧誘導響應元件(GC-motif)、壓力響應元件(TC-rich repeats和STRE元件)只分布于,損傷誘導響應元件(WUN-motif)只分布于,低溫誘導響應元件(LTR元件)只分布于。在家族中,中響應激素和脅迫的順式作用元件數量最多,推測其可能在激素和脅迫響應中的作用更為顯著。
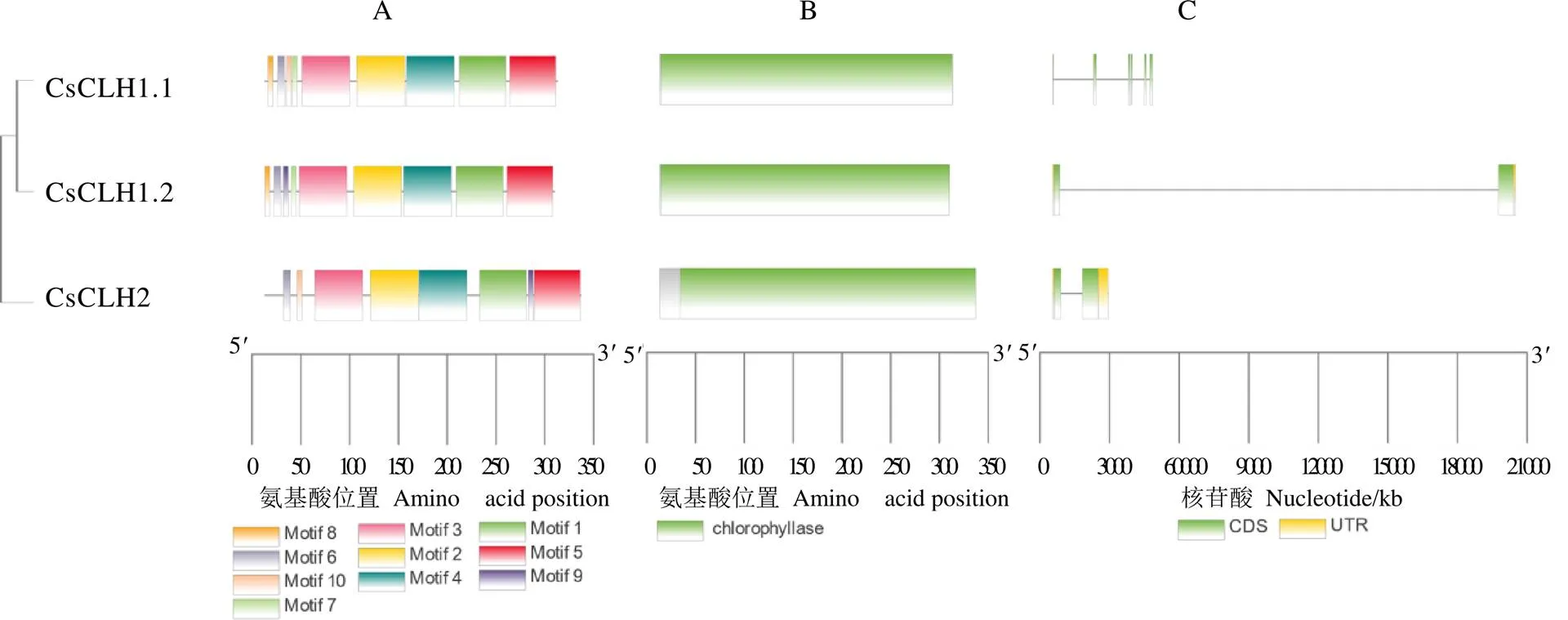
注:A:CsCLHs保守基序分布;B:蛋白質保守結構域;C:CsCLHs內含子-外顯子結構分布圖
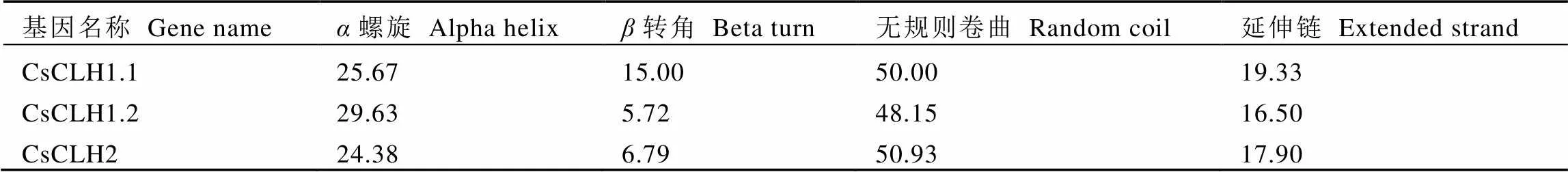
表6 茶樹CsCLHs家族蛋白二級結構預測

圖5 茶樹CsCLHs蛋白三級結構預測

注:Ⅰ:激素響應;Ⅱ:非生物脅迫響應;Ⅲ:光響應;Ⅳ:轉錄因子識別、結合位點;Ⅴ:組織特異性元件;Ⅵ:生長發育相關元件;Ⅶ:核心元件
2.6 茶樹CsCLHs上游調控轉錄因子預測及表達量分析
以模式植物擬南芥為參考,通過PlantTFDB在線數據庫預測與上游啟動子結合的轉錄因子并進行同源序列比對,共鑒定出22個轉錄因子,其中有12個轉錄因子同時與3個啟動子結合(表7)。通過分析遮陰和恢復光照處理下白雞冠第二葉轉錄組數據發現(圖7),22個轉錄因子中有6個差異表達基因,其中有1個(CSS0047505)表達量呈現出遮陰后下調,恢復光照后上調的表達趨勢,有2個(CSS0007067和CSS0027296)表達量呈現出遮陰后上調,恢復光照后下調的表達變化。
由酵母單雜交結果可知,pAbAi+Pro和pAbAi+Pro單轉Y1H后培養在SD/-URA培養基中,通過AbA篩選抑制酵母生長的濃度,發現在150?ng·mL-1AbA中酵母不生長,因此Pro和Pro啟動子的自激活質量濃度為150?ng·mL-1。以Y1H-pAbAi(Pro)和Y1H-pAbAi(Pro)為陰性對照,將pGADT7-BPC6和pGADT7-CDF5分別轉入Y1H-pAbAi(Pro)和Y1H-pAbAi(Pro)后在SD/-Leu和SD/-Leu/AbA 150?ng·mL-1平板培養。由圖8可知,Y1H(Pro+BPC6)、Y1H(Pro+CDF5)、Y1H(Pro+BPC6)和Y1H(Pro+CDF5)在SD/-Leu平板上正常生長,未轉入pGADT7-BPC6和pGADT7-CDF5的Y1H(Pro)和Y1H(Pro)無法生長,表明pGADT7-BPC6和pGADT7-CDF5成功轉入,而在SD/-Leu/AbA 150?ng·mL-1的平板上,Y1H(Pro+BPC6)和Y1H(Pro+BPC6)無法生長,Y1H(Pro+CDF5)和Y1H(Pro+CDF5)正常生長,表明CDF5能結合和的啟動子。
表7 茶樹CsCLHs上游調控轉錄因子基因組信息
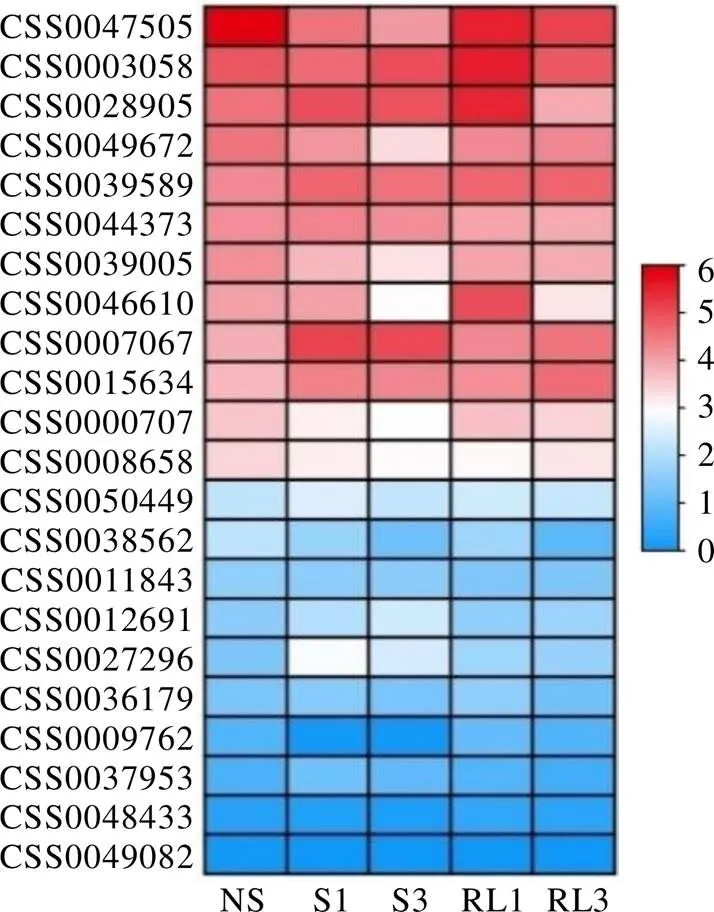
圖7 預測調控CsCLHs的轉錄因子表達量

注:A:SD/-Ura/AbA培養基;B:SD/-Leu/AbA培養基
2.7 茶樹CsCLHs基因在白化葉片中的表達分析
通過實時熒光定量PCR分析茶樹在遮陰和恢復光照處理下的表達情況,結果表明(圖9),在遮陰處理1?d和3?d,均呈現下調表達;恢復光照1?d,的表達量下調,相對表達穩定,而表達量極顯著上調;恢復光照2?d,的表達量均呈現極顯著上調趨勢。
為了進一步探究在調控茶樹葉色白化過程中的作用,以正常綠葉品種金牡丹為對照,對不同茶樹品種中的表達量進行檢測。結果表明(圖10),在白化茶樹中的表達量均顯著高于金牡丹;的表達模式顯示,除黃金芽和中黃1號外,其余品種的表達量均顯著高于金牡丹;的表達圖譜顯示,僅白雞冠和黃金芽的表達量高于金牡丹。
2.8 茶樹CsCLHs在不同組織中的表達分析
為了解家族基因在茶樹不同組織的表達模式,利用熒光定量PCR對的表達模式進行分析。結果表明(圖11),在嫩葉、成熟葉和老莖中的表達量較高,在根中表達量最低;在嫩葉和果實中的表達量較高,在其余各部位的表達量均處于較低水平;而在各個組織部位中均有較高的表達量,其中在成熟葉中表達量最高。

注:以CsCLH1.1在不遮陰葉片中的表達量為對照;顏色刻度表示差異倍數log2轉換均一化的值,紅色為高水平表達,藍色為低水平表達。NS,不遮陰;S1,遮陰1?d;S3,遮陰3?d;RL1,恢復光照1?d;RL2,恢復光照2?d

注:MD:金牡丹;BJG:白雞冠;HH:黃化品系;HJY:黃金芽;JG:金光;QNX:千年雪;YJX:御金香;ZH1:中黃1號;不同小寫字母表示品種間具有顯著差異,P<0.05
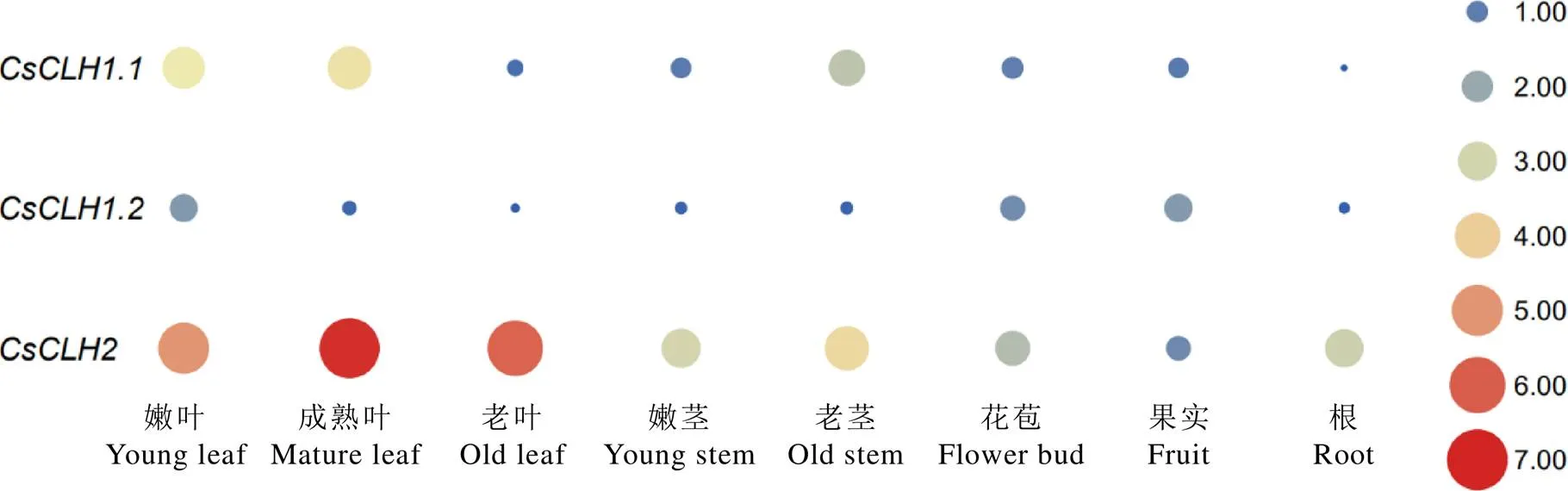
注:以CsCLH1.1在花苞中的表達量為對照;顏色刻度表示差異倍數log2轉換均一化的值,紅色為高水平表達,藍色為低水平表達
3 討論
植物葉綠素降解存在兩條途徑,分別是以葉綠素為底物的葉綠素酶降解途徑和以脫鎂葉綠素為底物的脫鎂葉綠素酶降解途徑[23],其中葉綠素酶被認為是葉綠素代謝途徑中的第一個酶[24],在葉綠素分解代謝中作為限速酶進行翻譯后調節[25]。在葉綠素酶理化研究方面,已有對波羅蜜[26]、雞爪槭[27]、小麥[28]等植物葉片和采后貯藏過程中的葉綠素酶活性變化的研究報道,而在茶樹中主要針對葉綠素酶的分離和純化,加工過程葉綠素酶活性變化,以及不同茶樹品種和不同葉位葉綠素酶活性的差異等進行研究。研究表明,在不同季節中春茶新梢葉綠素酶活性最高,其次為夏季、秋季,在茶樹不同葉位中,葉綠素酶活性隨著葉片衰老而降低[29]。與葉綠素酶理化性質研究相比,關于葉綠素酶分子水平上的研究尤其是基因克隆和轉錄表達水平的研究甚少。本研究在茶樹中共克隆出3條,根據前人的報道,CLH基因家族在不同植物中包含的基因數量不同,如擬南芥[16]、青花菜[30]、發財樹[31]等只有兩條CLH基因。系統進化樹分析結果顯示,茶樹CsCLH1s與葡萄等CLH1s聚在同一類亞群,茶樹CsCLH2和葡萄、擬南芥等植物的CLH2聚在一個亞群。亞細胞定位預測發現,Cell-PLoc和WolfPsort預測結果存在差異,Cell-PLoc預測CsCLH1s定位于葉綠體,而WolfPsort預測CsCLH1s定位于細胞質中,此結果和前人報道一致,在銀杏葉[32]、柑橘[33]等植物中,定位于嫩葉的葉綠體中,而在成熟葉或衰老葉中定位于細胞質、液泡膜和內質網中[34-36],表明葉綠素酶在植物不同組織間的功能存在差異。
CDF(Cycling dof factors)轉錄因子是植物中特有的轉錄因子家族,參與植物生長發育、光照和激素等非生物脅迫響應、碳氮代謝平衡等功能[37]。通過預測啟動子的上游調控因子,結合基因組和白雞冠茶樹第二葉遮陰和恢復光照處理的轉錄組數據,鑒定出兩個可能參與調控的轉錄因子(CSS0007067)和(CSS0027296),酵母單雜交結果表明可以與和的啟動子結合。Henriques等[38]研究發現,CDF5的表達受到光調控,在黑暗條件下高表達,在光照條件下低表達。本研究轉錄組數據中在遮陰后表達量上調,恢復光照后表達量下調,與呈現相反的表達模式,表明在白雞冠茶樹白化葉片中可能負調控和的表達。
葉綠素酶是葉綠素降解過程中的重要組成部分,順式作用元件分析結果表明,茶樹基因啟動子區包含了大量光響應有關的順式作用元件,因此本研究分析了白雞冠第二葉在遮陰和恢復光照處理下的表達模式。家族基因在遮陰及恢復光照處理后表達水平發生變化,即在遮陰下低表達,在恢復光照后高表達,這與周喆等[11]研究白雞冠茶樹第二葉在遮陰及恢復光照后的葉綠素含量和表達模式變化規律一致。在光照敏感型黃化品種黃金芽中也有類似的結果[39],表明在遮陰條件下,葉綠素降解途徑中葉綠素和脫鎂葉綠素的降解過程受阻,葉綠素和脫鎂葉綠素在葉片中積累,葉色復綠,而在光照條件下葉綠素酶和脫鎂葉綠素酶高表達,影響白雞冠茶樹葉片葉色變化。
研究表明,葉綠素酶在芽和嫩葉中有較高的活性[29],本研究中在嫩葉中表達量最高,在各部位中均有表達,其中在成熟葉、老葉和嫩葉中表達量較高,與前人研究結果一致。為了探究基因家族成員在調控茶樹葉色白化過程中的作用,本研究對金牡丹和白雞冠、黃金芽等8個品種表達量進行檢測,其中在白化品種中具有較高的表達量,李遠華等[29]研究表明茶樹葉綠素酶活性與葉綠素含量呈負相關,推測基因參與白化茶樹葉片白化過程。
本研究在茶樹全基因組范圍內鑒定獲得3條家族基因,并分析了其結構和功能,研究結果可為CsCLHs參與茶樹葉綠素代謝途徑并調控芽葉白化的機制研究提供參考。
[1] Zhong X M, Sun S F, Li F H, et al. Photosynthesis of a yellow-green mutant line in maize [J]. Photosynthetica, 2015, 53(4): 499-505.
[2] Li W X, Yang S B, Lu Z G, et al. Cytological, physiological, and transcriptomic analyses of golden leaf coloration inL [J]. Horticulture Research, 2018, 5: 12. doi: 10.1038/s41438-018-0015-4.
[3] Gang H X, Liu G F, Chen S, et al. Physiological and transcriptome analysis of a yellow-green leaf mutant in birch (×) [J]. Forests, 2019, 10(2): 120. doi: 10.3390/f10020120.
[4] Slattery R A, VanLoocke A, Bernacchi C J, et al. Photosynthesis, light use efficiency, and yield of reduced-chlorophyll soybean mutants in field conditions [J]. Frontiers in Plant Science, 2017, 8: 549. doi: 10.3389/fpls.2017.00549.
[5] 元世昌, 黃亞偉, 王若蘭, 等. 優質稻黃變期間營養組分的變化規律研究[J]. 食品科技, 2019, 44(6): 156-161.
Yuan S C, Huang Y W, Wang R L, et al. Changes of nutrient components during high quality rice yellowing [J]. Food Science and Technology, 2019, 44(6): 156-161.
[6] Ma L L, Liu Y L, Cao D, et al. Quality constituents of high amino acid content tea cultivars with various leaf colors [J]. Turkish Journal of Agriculture and Forestry, 2018, 42(6): 383-392.
[7] Shin Y H, Yang R, Shi Y L, et al. Light-sensitive albino tea plants and their characterization [J]. Hortscience, 2018, 53(2): 144-147.
[8] 范延艮, 趙秀秀, 王翰悅, 等. 黃金芽不同色澤葉片生理特性研究[J]. 茶葉科學, 2019, 39(5): 530-536.
Fan Y G, Zhao X X, Wang H Y, et al. Study on physiological charateristics of leaves with different colors of ‘Huangjinya’ [J]. Jounal of Tea Science, 2019, 39(5): 530-536.
[9] 吳全金. ‘白雞冠’茶樹響應光調控的基因差異及理化特征分析[D]. 福州: 福建農林大學, 2015.
Wu Q J. Gene differential analysis and physicochemical characteristics ofcv.in response to light [D]. Fuzhou: Fujian Agriculture and Forestry University, 2015.
[10] Li N, Yang Y P, Ye J H, et al. Effects of sunlight on gene expression and chemical composition of light-sensitive albino tea plant [J]. Plant Growth Regulation, 2016, 78(2): 253-262.
[11] 周喆, 陳志丹, 吳全金, 等. 白雞冠茶樹基因全長cDNA克隆與表達分析[J]. 茶葉科學, 2020, 40(1): 43-54.
Zhou Z, Chen Z D, Wu Q J, et al. Cloning and expression analysis ofgene in tea plant () [J]. Jounal of Tea Science, 2020, 40(1): 43-54.
[12] Dong F, Zeng L T, Yu Z M, et al. Differential accumulation of aroma compounds in normal green and albino-induced yellow tea () leaves [J]. Molecules, 2018, 23(10): 2677. doi: 10.3390/molecules23102677.
[13] Wang L, Yue C, Cao H L, et al. Biochemical and transcriptome analyses of a novel chlorophyll-deficient chlorina tea plant cultivar [J]. BMC Plant Biology, 2014(14): 352. doi: 10.1186/s12870-014-0352-x.
[14] Wu Q J, Chen Z, Sun W J, et al.sequencing of the leaf transcriptome reveals complex light-responsive regulatory networks incv.[J]. Frontiers in Plant Science, 2016, 7: 332. doi:10.3389/fpls.2016.00332.
[15] 李旭敏. 光敏型白化茶轉錄組分析及葉綠素代謝途徑相關基因研究[D]. 杭州: 浙江大學, 2019.
Li X M. Study on transcriptome and gene expression with regard to chlorophylls metabolism pathway in photosensitive albino tea plant [D]. Hangzhou: Zhejiang University, 2019.
[16] Tsuchiya T, Ohta H, Okawa K, et al. Cloning of chlorophyllase, the key enzyme in chlorophyll degradation: finding of a lipase motif and the induction by methyl jasmonate [J]. PNAs, 1999, 96(26): 15362-15367.
[17] Chen C M M, Chao P Y, Huang M Y, et al. Chlorophyllase activity in green and non-green tissues of variegated plants [J]. South African Journal of Botany, 2012, 81(4): 44-49.
[18] 梁俊林, 顧國軍, 唐實玉, 等. 銀杏葉變色期的生理特征[J]. 四川農業大學學報, 2020, 38(1): 65-70.
Liang J L, Gu G J, Tang S Y, et al. Physiological characteristics of leaf color change period ofL. [J]. Journal of Sichuan Agricultural University, 2020, 38(1): 65-70.
[19] Tian Y N, Zhong R H, Wei J B, et al.CHLOROPHYLLASE 1 protects young leaves from long-term photodamage by facilitating FtsH-mediated D1 degradation in photosystem Ⅱ repair [J]. Molecular Plant, 2021, 14(7): 1149-1167.
[20] 張蘭, 滕珂, 尹淑霞. 草地早熟禾葉綠素酶1基因的克隆和表達分析[J]. 中國草地學報, 2016, 38(4): 1-7.
Zhang L, Teng K, Yin S X. Cloning and expression analysis of chlorophyllase 1 genefromL. [J]. Chinese Journal of Grassland, 2016, 38(4): 1-7.
[21] Xu D, Lu Z C, Jin K M, et al. SPDE: a multi-functional software for sequence processing and data extraction [J]. Bioinformatics, 2021, 37(20): 3686-3687.
[22] Chen C J, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data [J]. Molecular Plant, 2020, 13(8): 1194-1202.
[23] Schelbert S, Aubry S, Burla B, et al. Pheophytin pheophorbide hydrolase (pheophytinase) is involved in chlorophyll breakdown during feaf senescence in[J]. Plant Cell, 2009, 21(3): 767-785.
[24] Takamiya K I, Tsuchiya T, Ohta H. Degradation pathway(s) of chlorophyll: what has gene cloning revealed? [J]. Trends in Plant Science2000, 5(10): 426-431.
[25] Harpaz-Saad S, Azoulay T, Arazi T, et al. Chlorohyllase is a rate-limiting enzyme in chlorophyll catabolism and is posttranslationally regulated [J]. The Plant Cell, 2007, 19(3): 1007-1022.
[26] 周丹, 羅燦, 于旭東, 等. 波羅蜜葉片突變體葉綠素含量測定和超微結構觀察[J]. 熱帶作物學報, 2021, 42(10): 2935-2941.
Zhou D, Luo C, Yu X D, et al. Determination of chlorophyll content and observation of ultrastructure in leaves of mutants of[J]. Chinese Journal of Tropical Crops, 2021, 42(10): 2935-2941.
[27] 梁俊林, 李儉, 代鑫, 等. 施加Fe2+和Cu2+對雞爪槭葉色變化的生理影響[J]. 應用與環境生物學報, 2021, 27(3): 549-554.
Liang J L, Li J, Dai X, et al. Physiological effects of Fe2+and Cu2+application on leaf color changes in[J]. Chinese Journal of Applied and Environmental, 2021, 27(3): 549-554.
[28] 王繪艷. 小麥葉綠素和脫鎂葉綠素酶基因的作用及表達分析[D]. 太原: 山西農業大學, 2015.
Wang H Y. The function and gene expression analysis of chlorophyllase and pheophttinase in[D]. Taiyuan: Shanxi Agricultural University, 2015.
[29] 李遠華, 顧瑋, 倪德江, 等. 茶樹葉綠素酶活性的變化研究[J]. 茶葉科學, 2011, 31(1): 27-32.
Li Y H, Gu W, Ni D J, et al. Study on variation of chlorophyllase activity in tea plant () [J]. Journal of Tea Science, 2011, 31(1): 27-32.
[30] 樊艷燕, 劉玉梅, 李占省, 等. 青花菜衰老過程中葉綠素降解相關基因的表達分析[J]. 園藝學報, 2015, 42(7): 1338-1346.
Fan Y Y, Liu Y M, Li Z X, et al. Analysis of the expression of chlorophyll degrading genes during senescence of[J]. Acta Horticulturae Sinica, 2015, 42(7): 1338-1346.
[31] Chen M C M, Yang J H, Liu C H, et al. Molecular, structural, and phylogenetic characterization of two chlorophyllase isoforms in[J]. Plant Systematics and Evolution. 2014, 300: 633-643.
[32] Okazawa A, Tang L, Itoh Y, et al. Characterization and subcellular localization of chlorophyllase from[J]. Zeitschrift für Naturforschung C, 2006, 61(1/2): 111-117.
[33] Shemer T A, Harpaz-Saad S, Belausov E, et al.chlorophyllase dynamics at ethylene-induced fruit color-break: a study of chlorophyllase expression, posttranslational processing kinetics, and in situ intracellular localization [J]. Plant Physiology, 2008, 148(1): 108-118.
[34] Hrtensteiner S, Krutler B. Chlorophyll breakdown in higher plants [J]. Biochimica et Biophysica Acta, 2011, 1807(8): 977-988.
[35] Nicole S A, Silivia S B, Marion K C, et al. The chlorophyllases AtCLH1 and AtCLH2 are not essential for senescence-related chlorophyll breakdown in[J]. FEBS Letters, 2007, 581(28): 5517-5525.
[36] Hu X, Makita S, Schelbert S, et al. Reexamination of chlorophyllase function implies its involvement in defense against chewing herbivores [J]. FEBS Letters, 2015, 167(3): 660-670.
[37] Begoa R M, Laura C, Jose D F, et al. CDF transcription factors: plant regulators to deal with extreme environmental conditions [J]. Journal of Experimental Botany, 2020, 71(13): 3803-3815.
[38] Henriques R, Wang H, Liu J, et al. The antiphasic regulatory module comprising CDF5 and its antisense RNA FLORE links the circadian clock to photoperiodic flowering [J]. The New phytologist, 2017, 216(3): 854-867.
[39] Fan Y G, Zhao X X, Wang H Y, et al. Effects of light intensity on metabolism of light-harvesting pigment and photosynthetic system inL. cultivar 'Huangjinya' [J]. Environmental and Experimental Botany, 2019, 166: 103796. doi: 10.1016/j.envexpbot.2019.06.009.
Identification and Transcriptional Regulation of CLH Gene Family and Expression Analysis in Albino Tea Plants ()
WANG Tao1,3,4,5, WANG Yiqing1,3,4,5, QI Siyu1,3,4,5, ZHOU Zhe1,3,4,5, CHEN Zhidan2,3,4,5*, SUN Weijiang1,3,4,5*
1. College of Horticulture, Fujian Agriculture and Forestry University, Fuzhou 350002, China; 2. Anxi College of Tea Science, Fujian Agriculture and Forestry University, Quanzhou 362400, China; 3. Engineering Technology and Research Center of Fujian Tea Industry, Fuzhou 350002, China; 4. Ministerial and Provincial Joint Innovation Centre for Safety Production of Cross-Strait Crops, Fujian Agriculture and Forestry University, Fuzhou 350002, China; 5. Tea Industry Technology Development Base of Fujian Province, Fuzhou 350002, China
Chlorophyllase (CLH) is the key enzyme in the degradation of chlorophyll, stripping its phytol to form dephytolithochlorophyll a. The full-length cDNA sequences of threegenes were obtained from the second leaves of albino tea cultivar ‘Baijiguan’, and bioinformatics analysis was performed. The results show that the threegenes could be divided into two subfamilies. The full length ofwas 894-975?bp, encoding 297-324 amino acids. The protein molecular weights were 31.99-34.91?kDa. The isoelectric points were 4.89-7.61, and the instability coefficients were 38.94-48.24. CsCLH1.1 and CsCLH1.2 were unstable proteins, while CsCLH2 was a stable protein. The subcellular localization prediction results of Cell Ploc show that three CsCLH proteins were located in chloroplast, while the results of Wolf Psort show that CsCLH1.1 and CsCLH1.2 were located in cytoplasm and CsCLH2 was located in chloroplast. The qRT-PCR results on the ‘Baijiguan’ leaves indicated that expressions ofwere inhibited by shading treatment and light induced’ expressions. Expression pattern analysis ofshows thatwere highly expressed in the albino cultivars. In addition, it was identified thatcould bind to theandpromoters according to the yeast one hybrid system. In conclusion, CsCLHs in albino tea leaves might be involved in chlorophyll degradation and play an important role in the process of albino leaf, which provided a reference for further investigation in the function of the CLH gene family and the albinism of leaves in tea plants.
, chlorophyllase, gene family, sequence analysis, differential expression
S571.1
A
1000-369X(2022)03-331-16
2021-12-14
2022-01-24
福建省高校產學合作項目(2019N5007)、國家自然科學基金(31770732)、國家重點研發計劃(2019YFD1001601)、福建農林大學“雙一流”建設科技創新能力提升培育計劃(KSYLC005)、福建張天福茶葉發展基金會科技創新基金(FJZTF01)
王濤,男,碩士研究生,主要從事茶樹栽培育種與生物技術方面研究。*通信作者:asbulletdan@163.com;swj8103@126.com
(責任編輯:黃晨)

