基于網絡藥理學的莫匹羅星抗結直腸癌潛在靶點及作用機制研究
黃吉 李子烜 孫震曉

摘要:目的 基于網絡藥理學探討莫匹羅星抗結直腸癌(CRC)的潛在靶點及作用機制。方法 通過SwissTargetPrediction數據庫預測莫匹羅星作用靶點,通過GeneCard數據庫獲取CRC相關靶點,利用Venny平臺篩選出二者交集靶點作為莫匹羅星抗CRC潛在靶點,并通過String數據庫構建潛在靶點的蛋白—蛋白相互作用(Protein-protein interaction,PPI)網絡,利用Cytoscape軟件對PPI進行拓撲分析及可視化。將潛在靶點導入Metascape及Bioinformatics平臺,進行GO和KEGG富集分析,構建熱圖,氣泡圖以及通路圖。結果 獲得了莫匹羅星抗CRC潛在作用靶點89個,其中關鍵靶點為JUN、PTGS2、SRC、STAT3、IL1B及CASP3。富集度較高的生物學過程、細胞組成及分子功能分別主要為細菌源性分子的反應、內溶酶體和蛋白激酶c活性等。莫匹羅星抗CRC的信號通路有TRP通道的炎癥介質調節、HIF-1信號通路等。結論 莫匹羅星抗CRC可能機制包括抑制癌細胞增殖和誘導癌細胞凋亡、抗炎以及破壞癌細胞微環境等。
關鍵詞:網絡藥理學;莫匹羅星;結直腸癌;靶點;作用機制;信號通路
中圖分類號:R978.1? ? ? ? ?文獻標志碼:A? ? ? ? ?文章編號:1001-8751(2023)01-0007-06
Possible Targets and Mechanisms of Mupirocin Against Colorectal Cancer Based on Network Pharmacology
Huang Ji,? ? Li Zi-xuan,? ? Sun Zhen-xiao
(School of Life Sciences, Beijing University of Chinese Medicine,? ? Beijing? ?102488)
Abstract: Objective To investigate the possible targets and mechanisms of mupirocin (MPC) against colorectal cancer (CRC) by network pharmacology. Methods The targets of mupirocin were predicted by the SwissTargetPrediction database, and the CRC-related targets were obtained from the GeneCard database. Using Venny platform to screen the intersection target of the two as the potential anti-CRC target of mupirocin,and the protein-protein interaction network (PPI) of the potential targets was constructed through the String database. Cytoscape was used to analyze and visualize the PPI topology. Potential targets of MPC against CRC were imported into Metascape and Bioinformatics platforms, GO and KEGG enrichment analysis was conducted, and heat maps, bubble maps and pathway maps were built. Results 89 potential targets of mupirocin anti-CRC were obtained, among which the key targets were JUN, PTGS2, SRC, STAT3, IL1B and CASP3. The biological process, cellular composition and molecular function with high enrichment were mainly the reaction of bacterial-derived molecules, endolysosome and protein kinase c activity, etc. The signaling pathways of mupirocin against CRC include the regulation of inflammatory mediators of TRP channels and the HIF-1 signaling pathway, etc. Conclusion The anti-colorectal mechanisms of MPC include inhibiting cancer cell proliferation and inducing cancer cell apoptosis, anti-inflammatory, and destroying cancer cell microenvironment, etc.
Key words: network pharmacology; mupirocin;colorectal cancer;targets; mechanisms;signaling pathways
近年來,我國結直腸癌(Colorectal cancer, CRC)的發病率和死亡率均呈上升趨勢。2018中國癌癥統計報告顯示,我國CRC發病率和死亡率在全部惡性腫瘤中分別位居第3位和第5位,新發病例37.6萬例,死亡病例19.1萬例,CRC發病率上升顯著,多數患者在確診時已屬于中晚期[1]。全球結直腸癌2020—2040年癌癥負擔分析指出,2020年,CRC占全球癌癥發病率的10%,占癌癥死亡人數的9.4%,根據對老齡化、人口增長和人類發展的預測,預計到2040年,全球CRC新發病例數將達到320萬例[2]。
研究發現[3],腸道微生物組在CRC的發展中起重要作用,而抗生素可以改變腸道微生物組,因此抗生素可能具有抗CRC活性。莫匹羅星是假單胞菌酸A,是臨床廣泛使用的抗生素,可通過與細菌異亮氨酰-tRNA合成酶特異性結合抑制蛋白質合成,具有抗菌作用[4]。本課題組[5]前期在有關抗CRC小分子藥物篩選中,發現莫匹羅星可能有抗CRC作用,體外研究發現,不同濃度的莫匹羅星作用于人結直腸癌HCT116細胞24、48和72 h,可以劑量和時間依賴性的方式抑制HCT116細胞活力。
已知在CRC發生發展過程中涉及大量癌細胞細胞增殖、分化、凋亡及炎癥等微環境相關的蛋白質及靶點,單一基因突變或信號通路改變無法解釋CRC的形成,同樣藥物抗結直腸癌也往往不是通過作用單一靶點或信號通路。網絡藥理學是融合系統生物學、生物信息學、網絡科學等的學科,從系統層次和生物網絡的整體角度出發,解析藥物與治療對象之間的分子關聯,揭示藥物的系統性藥理機制,從而指導新藥研發和臨床診療,是人工智能和大數據時代藥物系統性研究的新興原創學科[6]。本文將采用網絡藥理學來分析莫匹羅星抗CRC可能的靶點和作用機制,為后續莫匹羅星抗CRC臨床前及臨床研究提供理論依據。
1 材料與方法
1.1 莫匹羅星作用靶點預測
利用PubChem(https://pubchem.ncbi.nlm.nih.gov/)數據庫檢索莫匹羅星(Mupirocin),獲取化合物簡化分子線性輸入系統(Simplified Molecular Input Line Entry System,SMILES)結構式。將檢索得到的莫匹羅星的SMILES導入 SwissTargetPrediction 數據庫(http://www.swisstargetpre-diction.ch)進行反向分子對接,獲得莫匹羅星作用靶點。
1.2 獲取CRC相關疾病靶點
以“Colorectal Cancer”為關鍵詞,通過GeneCards(http://www. genecards. org/)數據庫檢索CRC相關的疾病靶點。
1.3 獲取莫匹羅星抗CRC的潛在靶點
將1.1,1.2獲取到的靶點通過Venny(https://bioinfogp.cnb.csic.es/tools/venny/)繪制韋恩圖,取交集作為莫匹羅星抗CRC的潛在靶點進行機制探討。
1.4 蛋白互作網絡構建及關鍵靶點篩選
將莫匹羅星抗CRC潛在靶點導入String數據庫(https://string-db. org),構建蛋白—蛋白相互作用(Protein-protein interaction,PPI)網絡,物種設定為“Homo sapiens”,利用Cytoscape 3.8.0 軟件對PPI進行可視化,并進行拓撲分析,根據介度中心性(Betweenness centrality),緊密中心性(Closeness centrality),節度中心性(Degree)篩選莫匹羅星抗CRC的關鍵靶點。
1.5 莫匹羅星抗CRC潛在靶點生物功能分析及通路分析
將潛在靶點導入Metascape及Bioinformatics平臺,對莫匹羅星抗CRC的潛在靶點進行GO(Gene ontology)功能富集分析和 KEGG(Kyoto encyclopedia of genes and genomes)信號通路富集分析,GO功能富集內容包括生物學過程(Biological process,BP)、細胞組成(Cellular component,CC)和分子功能(Molecular function,MF);KEGG整合了基因組、化學分子和生化系統等方面的數據,富集內容主要涉及代謝通路(Pathway)。篩選出莫匹羅星抗CRC可能參與的生物學過程與涉及的信號通路,并對結果進行可視化。
2 結果
2.1 莫匹羅星抗CRC疾病靶點篩選
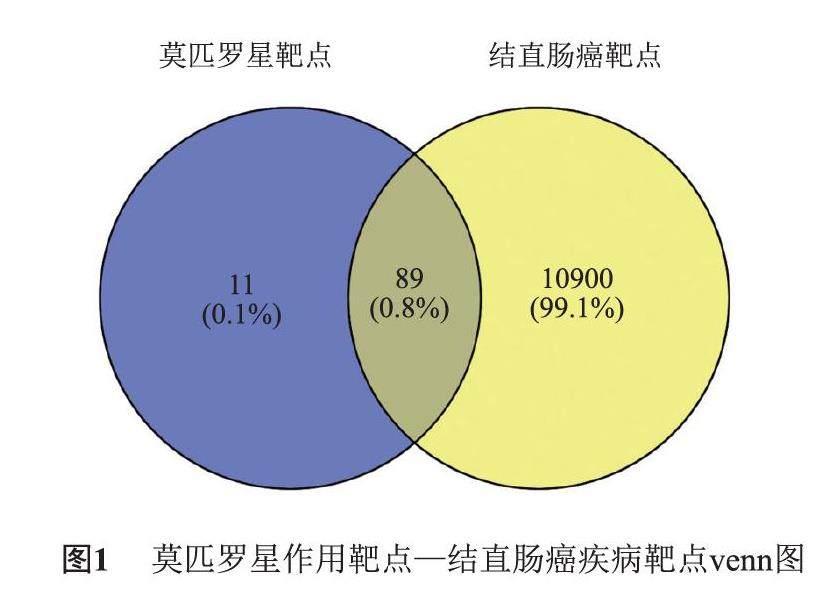
共獲得莫匹羅星作用靶點100個,CRC相關疾病靶點10 989個,二者共同作用靶點89個為潛在的莫匹羅星抗CRC靶點,繪制venn圖(圖1)。
2.2 莫匹羅星抗CRC關鍵靶點篩選
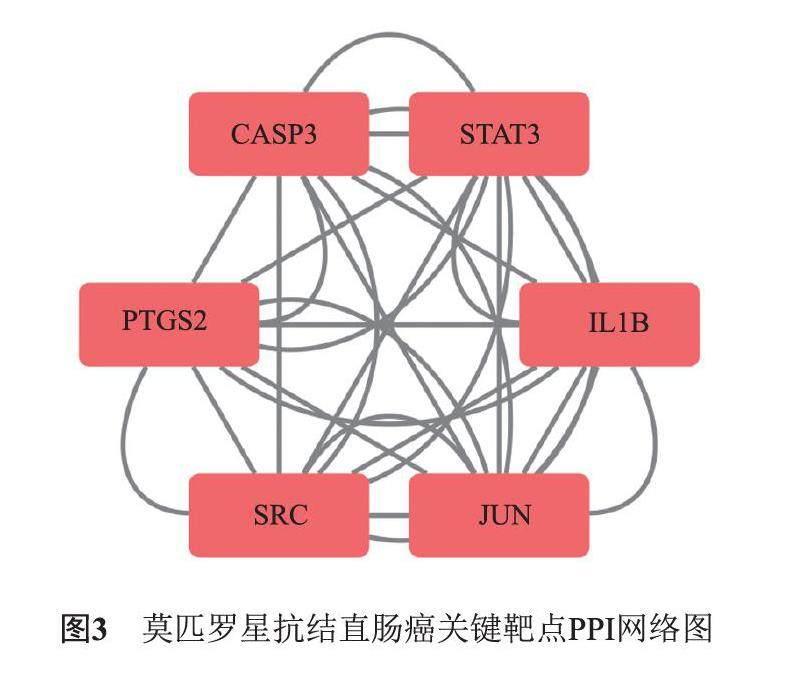
將莫匹羅星抗CRC潛在靶點導入String數據庫,構建PPI網絡圖(圖2),再利用Cytoscape軟件對潛在靶點進行拓撲分析,以其介度中心性(Betweenness centrality),緊密中心性(Closeness centrality),節度中心性(Degree)作為指標進行篩選,各組數值中大于本組中位數的靶點予以保留,按上述規則重復篩選3次,獲得莫匹羅星抗CRC關鍵靶點,即JUN,PTGS2,SRC,STAT3,IL1B及CASP3,并利用Cytoscape軟件將其可視化(圖3)。
2.3 莫匹羅星抗CRC潛在靶點GO功能分析
利用Metascape及Bioinformatics平臺對莫匹羅星抗CRC潛在靶點進行GO生物過程富集分析。選取P值最小的前10個項目進行可視化(圖4),生物學過程主要涉及細菌源性分子的反應、脂多糖的反應、藥物的反應、循環系統血管功能、肽分泌的調節、氧氣水平的反應、蛋白質分泌的調節、缺氧的反應、炎癥反應的調節以及血循環調節的反應。細胞組成主要包括內溶酶體、花萼突觸、內腔、膜筏、膜微區、膜區、細胞頂端、頂端質膜、空泡腔以及突觸后致密蛋白等。同時莫匹羅星抗CRC可能與蛋白激酶c活性、鈣依賴性蛋白激酶c活性、鈣依賴性蛋白絲氨酸/蘇氨酸激酶活性、鈣依賴性蛋白激酶活性、天冬氨酸型內肽酶活性、天冬氨酸型肽酶活性、核受體活性、配體、活化轉錄因子活性、內肽酶活性、腎上腺素受體結合等分子功能密切相關。
2.4 莫匹羅星抗CRC潛在靶點KEGG通路富集分析
利用Metascape及Bioinformatics平臺對莫匹羅星抗CRC潛在靶點進行KEGG通路富集分析(圖5),莫匹羅星抗CRC可能涉及的主要信號通路包括TRP通道的炎癥介質調節(Inflammatory mediator regulation of TRP channels),HIF-1信號通路(HIF-1 signaling pathway),人巨細胞病毒感染? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?(Human cytomegalovirus infection),糖尿病并發癥中的AGE-RAGE信號通路(AGE-RAGE signaling pathway in diabetic complications),化學致癌-受體激活(Chemical carcinogenesis-receptor activation),cAMP信號通路(cAMP signaling pathway),鈣信號通路(Calcium signaling pathway),甲狀旁腺激素的合成、分泌和作用(Parathyroid hormone synthesis, secretion and action),EGFR酪氨酸激酶抑制劑耐藥性(EGFR tyrosine kinase inhibitor resistance)以及非洲錐蟲病代謝(African trypanosomiasis)等。
3 討論
本研究通過網絡藥理學對莫匹羅星可能抗CRC的靶點和機制進行了解析。篩選到莫匹羅星抗CRC可能的關鍵靶點為JUN、PTGS2、SRC、STAT3、IL1B及CASP3;主要生物學過程、細胞組成及分子功能分別主要為細菌源性分子的反應、內溶酶體和蛋白激酶c活性等。莫匹羅星抗CRC的主要信號通路為TRP通道的炎癥介質調節等。
莫匹羅星抗CRC的6個關鍵靶點涉及細胞增殖、細胞凋亡、炎癥調控及腫瘤血管生成等。如非受體酪氨酸激酶(SRC),首先在CRC患者中被發現,其在超過80%的CRC患者中異常過度表達和激活[7],與健康人群正常黏膜相比,CRC患者SRC平均表達水平高2.4倍,CRC肝轉移瘤中增加2.2倍,肝外結直腸轉移瘤上調12.7倍[8]。SRC是有絲分裂關鍵調節因子,可活化調節黏附蛋白在細胞基質附著位點上的積累和局部密度,促進CRC細胞進入M期,促進細胞增殖[9]。白細胞介素-1β(IL-1β),參與多種細胞活動,如細胞增殖、分化和凋亡,另外研究發現,由IL-1β介導的髓系細胞產生的CTLA4信號直接促進腫瘤發生,并間接抑制免疫作用,促進腫瘤進展和轉移,阻斷IL-1β可顯著抑制抗PD1治療小鼠模型中的腫瘤進展和轉移[10],研究還發現,巨噬細胞產生的IL-1β誘導CRC細胞,可促進細胞遷移和侵襲的血清淀粉樣蛋白A1(SAA1)表達[11]。胱天蛋白酶3(CASP3)是細胞凋亡通路中最為關鍵的酶之一,與癌癥的發生等有重要聯系。miR-524-5p(CASP3為其靶基因)在GC(胃癌)組織樣品和細胞系中的表達顯著降低,GC細胞中miR-524-5p的增加顯著降低了細胞增殖能力,促進了細胞凋亡并誘導了G0/G1期的細胞周期停滯,miR-524-5p水平降低則促進了腫瘤的發生發展 [12]。JUN蛋白包含了v-Jun、c-Jun、Jun-B和Jun-D,是激活子蛋白-1(Activator Protein-1,AP-1)轉錄因子的家族成員之一,其中c-Jun作為AP-1家族的關鍵組分,在腫瘤的形成、轉移和侵襲中發揮重要作用[13]。c-Jun可以抑制miR-22的轉錄,miR-22直接與HuR的3'-UTR結合,導致HuR蛋白的抑制,從而抑制CRC在體外增殖和遷移,并減緩CRC異種移植腫瘤在體內的生長[14]。環氧合酶2(PTGS2),是前列腺素(PG)生物合成中的關鍵酶,PG參與細胞增殖分化凋亡等,影響炎癥、癌癥等病理過程。分析表明,基質PTGS2的表達促進癌癥相關成纖維細胞在腸腔附近發揮屏障功能[15]。但在放射治療時,PTGS2可激活NF-κB信號通路,防止DNA損傷,促進了腫瘤細胞增殖并提高其耐受性[16]。信號傳感器和轉錄激活器3(STAT3)與腫瘤細胞增殖、轉移和細胞因子促炎等有關。在原發性CRC中,集落刺激因子1受體(CSF1R)增加,STAT3通過介導miR-34a下調誘導CSF1R活化使CRC細胞遷移,侵襲和轉移[17]。血管生成對腫瘤發展至關重要,STAT3可激活具有促血管生成活性的YAP(Yes 相關蛋白)和TAZ(具有 PDZ 結合基序的轉錄共激活因子)。在小鼠CRC模型中,使用YAP/TAZ抑制劑Verteporfin治療,降低了血管密度和腫瘤進展[18]。
對莫匹羅星抗CRC潛在靶點進行KEGG通路富集分析,排名第一的為TRP通道的炎癥介質調節通路(圖6),影響背景框代表在該通路中莫匹羅星抗CRC的潛在靶點。由圖可見,莫匹羅星可以通過作用于MAPK信號通路(MAPK signaling pathway)、花生四烯酸代謝(Arachidonic acid metabolism)、鈣信號通路(Calcium signaling pathway)等途徑上的蛋白靶點調控CRC發生發展。
c-Jun N-末端激酶(JNK)是有絲分裂原活化蛋白激酶(MAPK)三個主要亞家族成員之一[19]。MAPK的3個主要亞家族均參與細胞代謝調控,通過上調基質金屬蛋白酶的表達,使癌細胞逃避凋亡[19-20]。莫匹羅星亦可作用IL-1B,在CRC細胞中, IL-1B可通過激活p38/JNK途徑誘導miR-31的轉錄,而miR-31與癌細胞的惡性程度、浸潤水平和轉移能力呈正相關[21-22]。炎癥是公認的癌癥標志,腫瘤微環境(TME)中炎癥細胞和介質的釋放可以增加癌細胞的存活率,促進腫瘤的進展,侵襲和轉移[23]。多不飽和脂肪酸(PUFAs)的氧化代謝對結腸腫瘤發生有強烈影響,花生四烯酸的水平可有助于炎癥微環境的發展[24],莫匹羅星可促進花生四烯酸的代謝,從而抑制炎癥的進展。TME往往由癌細胞、巨噬細胞等免疫細胞、內皮細胞、血細胞和腫瘤相關成纖維細胞(CAFs)組成,CAFs是生長因子的主要來源。鈣信號轉導是細胞內和細胞間信號轉導通路的關鍵第二信使,在腫瘤進展中重要的細胞機制通常是受鈣離子變化的驅動或影響[25]。CAFs參與腫瘤細胞的復雜雙向通信,有研究發現,白藜蘆醇激活CAFs中的TRPA1鈣通道,并導致大量的Ca2+流入和分泌VEGF和HGF[26]。這些發現表明CAFs產生并分泌基質細胞衍生因子-1(SDF-1)可促進癌細胞的遷移和侵襲。而莫匹羅星可通過作用鈣信號通路,從而影響CAFs的合成。
目前針對莫匹羅星抗腫瘤的實驗研究較少,除了前述我們課題組研究發現莫匹羅星體外有抗CRC活性[5],也有其他課題組報道了莫匹羅星對黑色素瘤細胞和乳腺癌細胞有細胞毒作用,還發現莫匹羅星可誘導腫瘤細胞凋亡和抑制(PI3K)-Akt信號通路[27]。由于莫匹羅星血清半衰期僅有30 min,通常作為外用抗菌藥物,但已有文獻報道,可將莫匹羅星配制成納米結構脂質載體,其釋放藥物的時長可長達24? h[28],也有研究證明莫匹羅星脂質體可以血管內給藥[29],因此通過網絡藥理學對莫匹羅星抗CRC的可能靶點和機制的解析,可以為將來莫匹羅星藥物重定位用于抗CRC研究提供支持。
參 考 文 獻
中華人民共和國國家衛生健康委員會醫政醫管局, 中華醫學會腫瘤學分會. 中國結直腸癌診療規范(2020年版)[J]. 中國實用外科雜志, 2020, 40(6): 601-625.
Xi Y, Xu P. Global colorectal cancer burden in 2020 and projections to 2040[J]. Transl Oncol, 2021, 14(10): 101174.
Aneke-Nash C, Yoon G, Du M, et al. Antibiotic use and colorectal neoplasia: a systematic review and meta-analysis[J]. BMJ Open Gastroenterol, 2021, 8(1): e000601.
Khoshnood S, Heidary M, Asadi A, et al. A review on mechanism of action, resistance, synergism, and clinical implications of mupirocin against Staphylococcus aureus[J]. Biomed Pharmacother, 2019, 109: 1809-1818.
Li Y, Li Y, Li D, et al. Repositioning of hypoglycemic drug linagliptin for cancer treatment[J]. Front Pharmacol, 2020, 11:187.
牛明, 張斯琴, 張博, 等.《網絡藥理學評價方法指南》解讀[J]. 中草藥, 2021, 52(14): 4119-4129.
Zhang S, Yang Z, Bao W, et al. SNX10 (sorting nexin 10) inhibits colorectal cancer initiation and progression by controlling autophagic degradation of SRC[J]. Autophagy, 2020, 16(4): 735-749.
Jin W. Regulation of Src family kinases during colorectal cancer development and its clinical implications[J]. Cancers, 2020, 12(5): E1339.
Sun X, Qi H, Zhang X, et al. Src activation decouples cell division orientation from cell geometry in mammalian cells[J]. Biomaterials, 2018, 170: 82-94.
Imazeki H, Ogiwara Y, Kawamura M, et al. CD11b+CTLA4+ myeloid cells are a key driver of tumor evasion in colorectal cancer[J]. J Immunother Cancer, 2021, 9(7): e002841.
Sudo G, Aoki H, Yamamoto E, et al. Activated macrophages promote invasion by early colorectal cancer via an interleukin 1β-serum amyloid A1 axis[J]. Cancer Sci, 2021, 112(10): 4151-4165.
Zhu C Y, Meng F Q, Liu J. MicroRNA-524-5p suppresses cell proliferation and promotes cell apoptosis in gastric cancer by regulating CASP3[J]. Eur Rev Med Pharmacol Sci, 2019, 23(18): 7968-7977.
Zhu G, Cheng Z, Huang Y, et al. TRAF6 promotes the progression and growth of colorectal cancer through nuclear shuttle regulation NF-kB/c-jun signaling pathway[J]. Life Sciences, 2019, 235: 116831
Liu Y, Chen X, Cheng R, et al. The Jun/miR-22/HuR regulatory axis contributes to tumourigenesis in colorectal cancer[J]. Mol Cancer, 2018, 17(1): 11.
Venè R, Costa D, Augugliaro R, et al. Evaluation of glycosylated PTGS2 in colorectal cancer for NSAIDS-based adjuvant therapy[J]. Cells, 2020, 9(3): 683.
Tan C, Liu L, Liu X, et al. Activation of PTGS2/NF-κB signaling pathway enhances radiation resistance of glioma[J]. Cancer Med, 2019, 8(3): 1175-1185.
Shi X, Kaller M, Rokavec M, et al. Characterization of a p53/miR-34a/CSF1R/STAT3 feedback loop in colorectal cancer[J]. Cell Mol Gastroenterol Hepatol, 2020, 10(2): 391-418.
Shen Y, Wang X, Liu Y, et al. STAT3-YAP/TAZ signaling in endothelial cells promotes tumor angiogenesis[J]. Sci Signal, 2021, 14(712):eabj8393.
Kim E K, Choi E J. Pathological roles of MAPK signaling pathways in human diseases[J]. Biochimica et Biophysica Acta, 2010, 1802(4): 396-405.
Huang Y, Zou Y, Lin L, et al. miR101 regulates the cell proliferation and apoptosis in diffuse large Bcell lymphoma by targeting MEK1 via regulation of the ERK/MAPK signaling pathway[J]. Oncology Reports, 2019, 41(1): 377-386.
Stefani C, Miricescu D, Stanescu-Spinu Ⅱ, et al. Growth factors, PI3K/AKT/mTOR and MAPK signaling pathways in colorectal cancer pathogenesis: where are we now?[J]. International J Molecular Sciences, 2021, 22(19): 10260.
Zhong L, Simoneau B, Huot J, et al. p38 and JNK pathways control E-selectin-dependent extravasation of colon cancer cells by modulating miR-31 transcription[J]. Oncotarget, 2017, 8(1): 1678-1687.
Mantovani A, Allavena P, Sica A, et al. Cancer-related inflammation[J]. Nature, 2008, 454(7203): 436-444.
Tutino V, De Nunzio V, Caruso M G, et al. Elevated AA/EPA ratio represents an inflammatory biomarker in tumor tissue of metastatic colorectal cancer patients[J]. International J Molecular Sciences, 2019, 20(8): E2050.
Monteith G R, Prevarskaya N, Roberts-Thomson S J. The calcium-cancer signalling nexus[J]. Nature Reviews Cancer, 2017, 17(6): 367-380.
Sadras F, Monteith G R, Roberts-Thomson S J. An emerging role for calcium signaling in cancer-associated fibroblasts[J]. Int J Mol Sci, 2021, 22(21): 11366.
Reva O N, Rademan S, Visagie M H, et al. Comparison of structures and cytotoxicity of mupirocin and batumin against melanoma and several other cancer cell lines[J]. Future Med Chem, 2019, 11(7): 677-691.
Singh A P, Sharma S K, Gaur P K, et al. Fabrication of mupirocin-loaded nanostructured lipid carrier and its In vitro characterization[J]. Assay Drug Dev Technol, 2021, 19(4): 216-225.
Alcantara K P, Zulfakar M H, Castillo A L. Development, characterization and pharmacokinetics of mupirocin-loaded nanostructured lipid carriers (NLCs) for intravascular administration[J]. Int J Pharm, 2019, 571: 118705.

