深度學習在抗菌肽藥物研究中的應用進展
朱尤卓?劉紅玉?游宇豪?鄭珩

摘要:抗菌肽(AMP)作為未來最有希望解決病原微生物耐藥性的新型抗菌藥物之一,其研發備受關注。抗菌肽一般較短,組成多樣,迄今人們已發現數千條天然抗菌肽,并建立了多個公開的抗菌肽數據庫,為新型抗菌肽的研發和設計奠定了基礎。另一方面,深度學習和人工智能作為信息處理的有力工具,已被大量應用于醫學影像信息處理、疾病診斷、藥物設計等領域,在抗菌肽的設計和研發上也受到廣泛關注。在抗菌肽的信息描述方面,人們使用了偽氨基酸殘基組成、位置特異性評分矩陣、獨熱碼等多種特征向量;在深度學習方法上,研究人員應用了循環神經網絡、卷積神經網絡、對抗生成網絡等多種算法,開發了ACEP、CLaSS等抗菌肽活性預測和序列生成模型。這些模型有望加速新型抗菌肽的發現,為應對耐藥菌感染,尤其是臨床上難以治療的耐藥性革蘭陰性菌感染,提供新的手段。
關鍵詞:深度學習;抗菌肽;數據庫;特征向量
中圖分類號:R978.1文獻標志碼:A
Application progress of deep learning in antimicrobial peptide drug research
Zhu You-zhuo, Liu Hong-yu, You Yu-hao, and Zheng Heng
(School of Life Science and Technology, China Pharmaceutical University, Nanjing 211198)
Abstract As one of the most promising new antimicrobial therapy to solve the drug resistance of pathogenic microorganisms in the future, the research and development of antimicrobial peptides (AMP) has attracted much attention. Antimicrobial peptides generally have short sequences and diverse composition. By now, thousands of natural antimicrobial peptides have been discovered, and many public antimicrobial peptide databases have been established, which lays a foundation for the research and develop of new antimicrobial peptides. On the other hand, as powerful tools of information processing, deep learning and artificial intelligence have been widely used in medical image processing, disease diagnosis, drug design, and so on. They have also attracted extensive attention in the design and research of antimicrobial peptides. For the descriptors of antimicrobial peptides, people use a variety of feature vectors such as pseudo amino acid composition, position specific scoring matrix, and one-hot coding. In the deep learning method, researchers apply a variety of algorithms such as Recurrent Neural Network, convolutional neural network, and Generative Adversarial Networks, and develop the models of antimicrobial peptide activity prediction and sequence generation such as ACEP and CLaSS. These models are expected to accelerate the discovery of new antimicrobial peptides, and provide new means to deal with drug-resistant bacterial infection, especially drug-resistant Gram-negative bacterial infections that are difficult to treat clinically.
Key words Deep learning; Antimicrobial peptides; Database; Eigenvector
一份抗生素耐藥性的評估報告指出,2050年可能有1000萬人死于耐藥細菌感染[1]。雖然目前上市的抗生素對治療絕大多數感染仍有效[2],但由于人類長期廣泛使用抗生素,越來越多的耐藥菌出現,尤其令人擔憂的多重耐藥菌,包括屎腸球菌、金黃色葡萄球菌、肺炎克雷伯菌、鮑曼不動桿菌、銅綠假單胞菌、腸桿菌屬等,它們造成了醫院中的許多嚴重感染[3]。目前作為治療耐藥菌的最后手段的碳青霉烯類抗生素和黏菌素也開始面臨耐藥性的問題[4],因此需要新的抗菌藥物來應對這一問題。
抗菌肽(antimicrobial peptide, AMP)是最有希望解決耐藥菌問題的新型抗菌藥物之一,序列一般較短,組成變化多樣,但多為陽離子兩親性多肽分子,其抗菌作用具有多種可能機制,其中最常見的是通過與帶負電荷的脂多糖(革蘭陰性)或脂磷壁酸(革蘭陽性)的磷酸基團的靜電相互作用到達細胞膜,以庫侖力吸附于細胞膜或進入細胞,隨后膜破裂、細胞質滲漏,導致細菌死亡[5]。抗菌肽通過靶向整個細胞成分,而不是特定的分子,具有廣譜的抗菌活性,同時避開了碳青霉烯類和替加環素等單一靶點藥物的耐藥性機制,該生化特性和藥效學性質使其比傳統抗生素更難耐藥[6]。可惜的是,雖然迄今人們已發現成千上萬條天然抗菌肽,且已有多個抗菌肽數據庫被建立并公開,但是一方面由于抗生素新藥研發耗時、昂貴、失敗率高且盈利空間小,新抗菌藥物的研發進入了冷門期,大型制藥公司已基本放棄該市場[2],另一方面因抗菌肽結構不穩定性、多肽易降解和非特異性膜裂解的體內毒性等因素,限制了抗菌肽臨床使用[7]。目前只有極少數公開的AMP獲得美國食品和藥物管理局(Food and Drug Administration, FDA)的批準[8]。
深度學習作為大數據處理的有力工具,已被大量應用于醫學影像信息處理、疾病診斷、藥物設計等領域,其有著高效且準確的判別能力,Stokes等[9]就利用深度神經網絡從1.07億個分子發現了在小鼠體內有廣譜抗菌活性的新抗生素halicin,使用這一方法可在4 d內完成十多億化合物分子的虛擬篩選,其效率遠超傳統的篩選手段。這是一種可以低成本、高效地發現活性高、毒性低以及結構穩定能臨床應用的抗菌肽的新方法。
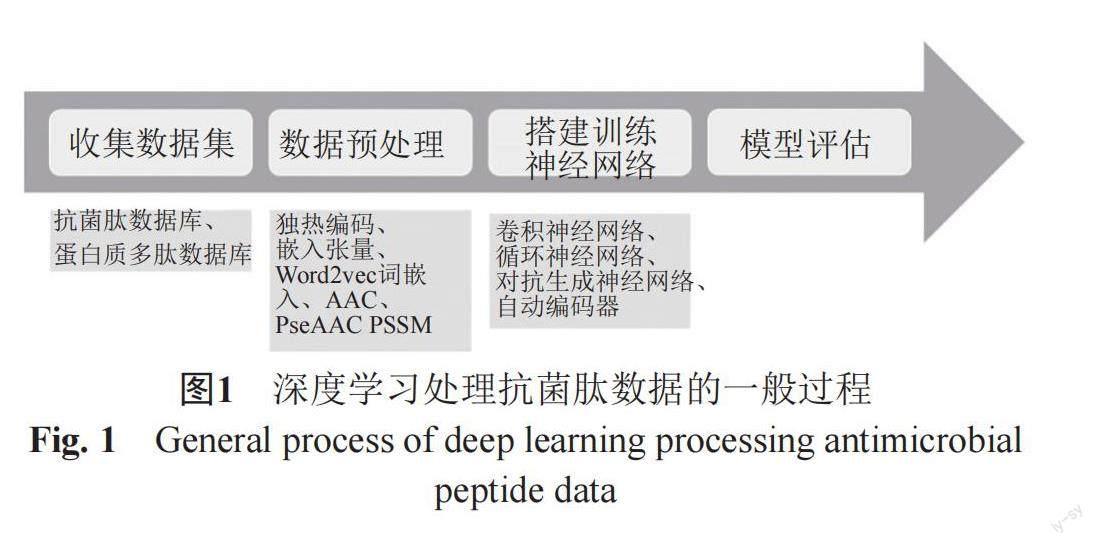
深度學習是根據經驗(數據)自動確定深層網絡參數的一門科學,它主要利用包含多個隱藏層的深層神經網絡學習大量數據中的潛在規律以輸出可靠結果,常見有監督和無監督學習兩種方式。當通過訓練而最小化的損失是網絡輸出和預先指定的期望輸出(即訓練集中的明確標簽)之間的誤差的度量時,訓練被稱為有監督的,如卷積神經網絡(convolutional neural network, CNN)、循環神經網絡(recurrent neural network, RNN)。當通過訓練而最小化的損失不涉及一組樣本輸入的預先指定的期望輸出時,該訓練被稱為無監督,如對抗生成網絡(generative adversarial networks, GAN)、自動編碼器(auto encoder, AE)[10]。深度學習方法雖然在大數據分析中具有普適性,但在不同應用中每個節點的處理上又具有特殊性,圖1展示了深度學習處理抗菌肽數據的一般過程,前兩步中數據來源與數據處理方法是明顯區別于深度學習在其他方面的應用,另外,不同的神經網絡模型具有不同的用途,CNN和RNN常被用于抗菌肽的活性預測[11-14],而GAN和AE則多被用于抗菌肽序列生成[15-17],但RNN有時也會被用于序列生成[18],這些都是值得我們特別去關注的。
1 收集數據集
抗菌肽又稱宿主防御肽(host defensin peptide, HDP),廣泛存在于自然界生物中[19]。自1922年發現溶菌酶開始,到1950年代左右發現桿菌肽和萬古霉素,再到1980年代人們掀起一波對AMP的研究熱潮,每年發現的AMP數量從1990年代的約50個增加到2000年的約100個[20],2010—2015年更是平均每年發表12,000篇相關文章[21],各種抗菌肽的序列、結構、活性以及修飾等信息越來越多被公開。研究者對這些信息進行收集整理,構建了許多抗菌肽數據庫。表1展示一些通用抗菌肽數據庫及其相關信息,更多特定的抗菌肽數據庫可以查看文獻[22]。
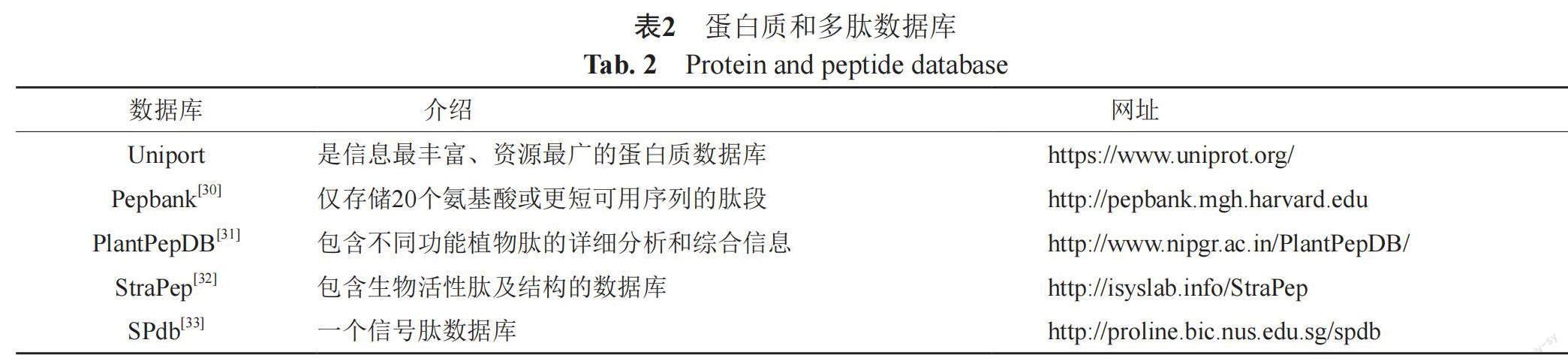
對于有監督學習,數據集中的每一個條目都有一個標簽,作為期望輸出。在抗菌肽活性預測等二分類問題中,這一標簽通常是有活性(陽性)或無活性(陰性),具有不同標簽的數據組成不同的數據集,陽性數據集常在抗菌肽數據庫中收集,陰性數據集則多在其他蛋白質多肽數據庫中收集,兩者作為訓練集和測試集輸入神經網絡。表2展示了一些常見的蛋白質多肽數據庫,更多的多肽數據庫可以查看文獻[29]。而對于無監督學習,數據不需要標注,Das等[17]就設計了可以在UniProt數據庫中報告的所有肽序列(可能無注釋)上訓練的無監督學習模型。
2 數據預處理
數據的特征是決定神經網絡訓練上限的關鍵因素。對于收集到的抗菌肽和其他多肽數據集,往往需要預處理把它轉化為神經網絡可識別的數據,即構建抗菌肽的特征參數或特征描述符。抗菌肽特征構建不僅借鑒計算機科學處理序列問題時使用的獨熱編碼(one-hot encoder)、特征張量嵌入(feature tensor embedding)和Word2vec詞嵌入等方式,同時還伴隨生物信息學和計算生物學的進步產生更復雜和更具描述性的特征,這些特征不僅與理化性質有關,而且與微觀層面的順序結構以及進化信息等有關,如氨基酸組成(amino acid composition, AAC),偽氨基酸組成(pseudo amino acid composition, PseAAC),位置特異性評分矩陣(position-specific scoring matrix, PSSM)等。
獨熱編碼是一種較為經典的多肽序列編碼方式。它指的是分配20個輸入單元來描述一個蛋白質殘基,在二十維空間中,如用向量[1, 0, 0, 0…0, 0, 0]表示丙氨酸,[0, 0, 0…0, 0, 0, 1]表示纈氨酸[34]。獨熱編碼作為一種多肽序列特征能在一定程度上反應多肽的序列信息,但它數據過于離散,很難捕捉到氨基酸之間的相似之處和不同之處[35]。特征張量嵌入則能較好地解決這一點,它利用概率生成的張量對氨基酸殘基編碼,該編碼成為模型可訓練的一部分,將氨基酸映射到可訓練的實數張量,使用反向傳播算法不斷更新這些實數張量,氨基酸之間的相似性和差異性便可通過張量之間的幾何距離來度量[11,35]。Word2vec詞嵌入是自然語言處理中的一種網絡模型,基于從大量文檔語料庫中收集鄰近的單詞數據,通過訓練數據所學得的參數,即隱層的權重矩陣,生成該詞語具有上下文屬性的嵌入特征向量,其中類似向量往往分配給出現在類似上下文中的單詞。Hamid等[36]把多肽序列中的連續3個氨基酸作為一個“詞”,然后利用Word2vec中的skip-gram模型生成的詞嵌入向量,用于細菌素識別。
氨基酸組成是Nakashima和Nishikawa在1994年提出的,它現在一般指多肽序列中20種氨基酸分別出現的頻率,是一個有20個組分的向量[37-38]。在此基礎上,發展出了偽氨基酸組成,其利用位置間隔為λ的氨基酸的疏水性值、親水性值以及側鏈質量等(都進行歸一化處理)計算λ階相關系數(θλ),若以向量X表示多肽的偽氨基酸組成,那么X中的前20個組分是歸一化處理后第i種氨基酸出現頻率?i(i=20),反映了氨基酸組成的影響,后λ個元素是歸一化處理后有一定權重值ω的θλ,反映了氨基酸順序和理化性質的影響[39]。現人們可以通過網頁服務器http://chou.med.harvard.edu/bioinf/PseAA/生成所需的PseAAC[40]。在PseAAC的基礎上,還發展出了偽K-tuple減少氨基酸組成(pseudo K-tuple reduced amino acids composition, PseKRAAC)[41]等方法。
位置特異性評分矩陣(PSSM)是進化信息的一種常見表示[42],一個長為L的多肽序列中,其每一個位置氨基酸突變為20種氨基酸的概率就構成了大小為L×20的PSSM矩陣[43]。PSSM矩陣可以通過PSI-BLAST程序獲得,被Fu等[11]用于抗菌肽識別并獲得了不錯的結果。
3 深度學習模型及應用
深度學習是由多個處理層組成的計算模型,可學習具有多個抽象特征的數據,并通過反向傳播算法來指示機器應該如何更新內部參數,從而發現大數據集中的復雜結構,它已經在預測潛在藥物分子的活性等方面擊敗了其他機器學習技術[44],同時它在抗菌肽抗菌活性預測以及序列生成等方面也有著不錯的表現。
卷積神經網絡(CNN)的基本結構由輸入層、卷積層(convolutional layer)、池化層(pooling layer,也稱為取樣層)、全連接層及輸出層構成,可以有效地降低網絡的復雜度,減少訓練參數的數目,使模型對平移、扭曲、縮放具有一定程度的不變性,并具有強魯棒性和容錯能力,且也易于訓練和優化[45]。Yan等[12]利用PseKRAAC和卷積神經網絡開發了一個基于序列的短AMP分類模型,稱為Deep-AmPEP30,該模型準確率比現有的基于機器學習的方法提高了77%,并且發現了與氨芐青霉素活性相當的抗菌肽P3(FWELWKFLKSLWSIFPRRRP)。
循環神經網絡(RNN)是一類非常強大的用于處理和預測序列數據的神經網絡模型,通過隱藏層上的回路連接,使得前一時刻的網絡狀態能夠傳遞給當前時刻,當前時刻的狀態也可以傳遞給下一個時刻[46],使得序列中的元素相互關聯,另外人們通過在RNN單元中引入輸入門,輸出門和遺忘門,構建了長短期記憶模型(long short-term memory, LSTM),不僅提高標準循環單元的記憶能力,同時也解決了長期依賴的問題[47]。Wang等[18]搭建基于LSTM和雙向LSTM的模型成功地生成并篩選到可能具有抗大腸埃希菌活性的新型AMPs。
深度學習用于抗菌肽研究時,往往不局限于用單一的神經網絡分析抗菌肽數據集。Daniel等[13]就構建了一個包含嵌入層(embedding layer),卷積層(convolutional layer),最大池化層(max pooling layer)和LSTM層的深層神經網絡模型,可以正確識別超過 98% APD 3數據庫中的對革蘭陽性或革蘭陰性細菌具有活性的AMP。
另外深度生成模型也被用于抗菌肽序列的自動生成。自動編碼器(AE)可通過編碼器和解碼器學習輸入分子特征(及其屬性),然后在潛在數據空間進行雙向映射來生成新的分子,已被用于設計一個完全自動化的計算框架CLaSS。CLaSS使用自動編碼器在多肽分子信息構建的潛在數據空間上進行訓練,再利用線性插值的方法在空間中采樣生成新的多肽序列,然后使用深度學習分類器以及從高通量分子動力學模擬得出的物理化學特征,來篩選生成的多肽分子,可用于廣譜的AMP序列的從頭設計與篩選。Das等[17]使用該方法獲得兩條對各種革蘭陽性和革蘭陰性病原菌(包括多重耐藥的肺炎克雷伯菌)具有較高效力的抗菌肽YI12(YLRLIRYMAKMI)和FK13(FPLTWLKWWKWKK),同時它們在小鼠實驗中也顯示了較低的毒性。除了自動編碼器,生成對抗網絡(GAN)也被用于產生新的抗菌肽,它通過生成模型和判別模型的相互博弈學習,而產生較好的輸出結果。Tucs等[15]設計的PepGAN模型可以控制生成序列的概率分布,使之盡可能多地覆蓋活性肽,用該模型生成了一個最低抑菌濃度僅為氨芐西林一半的高活性抗菌肽AMP4 (GLKKLFSKIKIGSALKNLA) 。表3總結了一些用于抗菌肽研究的深度學習模型。
4 模型的評估及不足之處
對于深度學習模型的評估,通常包括計算和實驗兩大類的方法,在計算上常使用靈敏度(sensitivity)、特異性(specificity)、準確率(accuracy)以及馬修相關系數(matthews correlation coefficient,MCC)等作為評估指標,使用測試數據集來判斷模型的準確性。但由于現有的抗菌肽特征表示方法,尚難以完整地描述抗菌肽特征,也缺乏可以模擬和描述AMP各種結構及物理化學特性的堅實理論[49],其生成和預測結果并不完全可信,因此常常需要與其他方法結合加以驗證,比如Puentes等[50]提出了4種新興技術相結合的抗菌肽設計篩選流程,包括人工智能、分子動力學、微生物表面展示(surface-display in microorganisms)和微流控(microfluidics),前兩個是篩選和設計的計算機策略,而后兩個對應于實驗方法的合成和測試。使用實驗合成并測試設計篩選的新型抗菌肽的活性,可以更準確地評估模型的效果,同時也可以發現一些有潛力的新抗菌肽。
另一方面,限制抗菌肽臨床應用的一個問題是毒副作用相對較大,尤其是溶血性問題,但是深度神經網絡需要大量數據進行學習,目前可收集到抗菌肽相關溶血毒性實驗數據較少,因此對于抗菌肽溶血毒性等深度學習預測模型也較少。在小分子藥物毒性預測方面,人們已經開發了一些數據庫和算法,如ToxAlert[51]和商業軟件Discovery Stadio中的ADMET模塊,這些方法通過統計或機器學習的方法,歸納出潛在的毒性結構基團,用于化合物分子的毒性預測。但是對于多肽,尤其是天然氨基酸組成的多肽,在其組成成分上通常沒有明確的毒性基團,另一方面由于多肽結構的欠缺,也使得基于結構毒性基團預測較為困難。因此,在該領域還需要加強相關的研究,以促進抗菌肽的臨床應用。
5 總結與展望
目前,深度學習和人工智能技術可以加速藥物發現,在很大程度上為抗耐藥菌感染藥物的研發提供了新的化合物。對于只含天然氨基酸的抗菌肽序列,上述多種特征構建方法可用于深度學習,以預測抗菌活性或產生新的抗菌肽,但對于含復雜修飾且未知空間結構的抗菌肽,如訂書肽(即在多肽結構中加入一個碳氫側鏈或其他類型側鏈以穩定其二級結構的多肽)[52],尚缺乏合適的結構表征方法,并且由于相應非天然抗菌肽的數據量較少,難以構建深度學習模型。可喜的是,深度遷移學習以及圖神經網絡等新的算法出現,有希望解決這些難題,前者可以在小數據集數據不足的情況下,先在大數據集上預訓練,然后在特定目標數據集(即小數據集)上微調模型參數以實現模型在小數據集上的良好表現[53],后者則是能將多肽分子中原子和鍵轉變為節點與邊的圖結構進行學習,實現對多肽復雜結構的表征,已被用于多肽毒性的預測[54]。同時抗菌肽等多肽以及蛋白質的數據庫在不斷地完善和豐富,更大的數據源變得公開可用,這些數據可以被進一步挖掘,并用于探索化學空間的新領域[55]。因此隨著計算方法的發展和抗菌肽數據的增加,以深度學習為代表的人工智能方法,有望成為應對多重耐藥菌問題以及發現新型抗菌藥物的重要技術手段。
參 考 文 獻
ONiel J. Tackling drug-resistant infections globally: Final report and recommendations[R]. London: Government of the United Kingdom, 2016: 1.
Rdal C, Balasegaram M, Laxminarayan R, et al. Antibiotic development-economic, regulatory and societal challenges[J]. Nat Rev Microbiol, 2019, 18(5): 267-274.
Tommasi R, Brown D G, Walkup G K, et al. ESKAPEing the labyrinth of antibacterial discovery[J]. Nat Rev Drug Discov, 2015, 14(8): 529.
沙國萌, 陳冠軍, 王祿山. 抗生素耐藥性的研究進展與控制策略[J]. 微生物學通報, 2020, 47(10): 3369-3379.
Lazzaro B P, Zasloff M, Rolff J. Antimicrobial peptides: Application informed by evolution[J]. Science, 2020, 368(6490): eaau5480.
Nagarajan D, Roy N, Kulkarni O, et al. Ω76: A designed antimicrobial peptide to combat carbapenem- and tigecycline-resistant Acinetobacter baumannii[J]. Sci Adv, 2019, 5(7): eaax1946.
Mourtada R, Herce H D, Yin D J, et al. Design of stapled antimicrobial peptides that are stable, nontoxic and kill antibiotic-resistant bacteria in mice[J]. Nat Biotechnol, 2019, 37(10): 1186-1197.
Annunziato G, Costantino G. Antimicrobial peptides (AMPs): A patent review (2015-2020)[J]. Expert Opin Ther Patents, 2020, 30(12): 931-947.
Stokes J M, Yang K, Swanson K, et al. A deep learning approach to antibiotic discovery[J]. Cell, 2020, 180(4): 688-702.e13.
Kriegeskorte N, Golan T. Neural network models and deep learning[J]. Curr Biol, 2019, 29(7): R225-R240.
Fu H, Cao Z, Li M, et al. ACEP: Improving antimicrobial peptides recognition through automatic feature fusion and amino acid embedding[J]. BMC Genomics, 2020, 21(1): 597.
Yan J, Bhadra P, Li A, et al. Deep-AmPEP30: Improve short antimicrobial peptides prediction with deep learning[J]. Mol Ther-Nucl Acids, 2020, 20: 882-894.
Daniel V, Uday K, Amarda S. Deep learning improves antimicrobial peptide recognition[J]. Bioinformatics, 2018, 34(16): 2740-2747.
Müller A T, Hiss J A, Schneider G. Recurrent neural network model for constructive peptide design[J]. J Chem Inf Model, 2018, 58(2): 472-479.
Tucs A, Tran D P, Yumoto A, et al. Generating ampicillin-level antimicrobial peptides with activity-aware generative adversarial networks[J]. ACS Omega, 2020, 5(36): 22847-22851.
Dean S N, Walper S A. Variational autoencoder for generation of antimicrobial peptides[J]. ACS Omega, 2020, 5(33): 20746-20754.
Das P, Sercu T, Wadhawan K, et al. Accelerated antimicrobial discovery via deep generative models and molecular dynamics simulations[J]. Nat Biomed Eng, 2021, 5(6): 613-623.
Wang C, Garlick S, Zloh M. Deep learning for novel antimicrobial peptide design[J]. Biomolecules, 2021, 11(3): 471.
Boparai J K, Sharma P K. Mini review on antimicrobial peptides, sources, mechanism and recent applications[J]. Protein Pept Lett, 2020, 1(27): 4-16.
Wang G. The antimicrobial peptide database provides a platform for decoding the design principles of naturally occurring antimicrobial peptides[J]. Protein Sci, 2019, 29(1): 8-18.
Ageitos J M, Sánchez-Pérez A, Calo-Mata P, et al. Antimicrobial peptides (AMPs): Ancient compounds that represent novel weapons in the fight against bacteria[J]. Biochem Pharmacol, 2017, 133: 117-138.
Liu S, Fan L, Sun J, et al. Computational resources and tools for antimicrobial peptides[J]. J Pept Sci, 2017, 23(1): 4-12.
Wang G, Li X, Wang Z. APD3: The antimicrobial peptide database as a tool for research and education[J]. Nucleic Acids Res, 2016, 44(1): 1087-1093.
Hanif W F, Shankar B R, Pratima G, et al. CAMPR3: A database on sequences, structures and signatures of antimicrobial peptides[J]. Nucleic Acids Res, 2016, 44(1): 1094-1097.
Shi G B, Kang X Y, Dong, F Y, et al. DRAMP 3.0: An enhanced comprehensive data repository of antimicrobial peptides[J]. Nucleic Acids Res, 2021, 50(1): 488-496.
Malak P, Amstrong A A, Maia G, et al. DBAASP v3: Database of antimicrobial/cytotoxic activity and structure of peptides as a resource for development of new therapeutics[J]. Nucleic Acids Res, 2021, 49(1): 288-297.
Jhong J H,Chi Y H,Li W C, et al. dbAMP: An integrated resource for exploring antimicrobial peptides with functional activities and physicochemical properties on transcriptome and proteome data[J]. Nucleic Acids Res, 2018, 47(1): 285-297.
Ye G, Wu H, Huang J, et al. LAMP2: A major update of the database linking antimicrobial peptides[J]. Database, 2020, 2020: baaa061.
Dong F Y, Zhao G L, Tong H, et al. The prospect of bioactive peptide research: A review on databases and tools[J]. Curr Bioinform, 2020, 16(4): 494-504.
Duchrow T, Shtatland T, Guettler D, et al. Enhancing navigation in biomedical databases by community voting and database-driven text classification[J]. BMC Bioinformatics, 2009, 10(1): 317.
Das D, Jaiswal M, Khan F N, et al. PlantPepDB: A manually curated plant peptide database[J]. Sci Rep, 2020, 10(1): 2194.
Wang J, Yin T, Xiao X, et al. StraPep: a structure database of bioactive peptides[J]. Database, 2018, 2018: bay038.
Choo K H, Tan T W, Ranganathan S. SPdb-a signal peptide database[J]. BMC Bioinformatics, 2005, 6: 249.
Lin K, May A, Taylor W R. Amino acid encoding schemes from protein structure alignments: Multi-dimensional vectors to describe residue types[J]. J Theor Biol, 2002, 216(3): 361-365.
ElAbd H, Bromberg Y, Hoarfrost A. Amino acid encoding for deep learning applications[J]. BMC Bioinformatics, 2020, 21(10): 660-668.
Hamid M N, Friedberg I, Hancock J. Identifying antimicrobial peptides using word embedding with deep recurrent neural networks[J]. Bioinformatics, 2018, 35(12): 2009-2016.
Nakashima H, Nishikawa K. Discrimination of intracellular and extracellular proteins using amino acid composition and residue-pair frequencies[J]. J Mol Bio, 1994, 238(1): 54.
Guo Z, Yang S, Hu Q, et al. A transverse and longitudinal encoding of protein sequence and its application[J]. J Comput Theor Nanosci, 2013, 10(2): 271-275.
Chou K C. Prediction of protein cellular attributes using pseudo-amino acid composition[J]. Proteins, 2001, 43(3): 246-255.
Shen H B, Chou K C. PseAAC: A flexible web server for generating various kinds of protein pseudo amino acid composition[J]. Anal Biochem, 2008, 373(2): 386-388.
Zuo Y, Yuan L, Chen Y, et al. PseKRAAC: A flexible web server for generating pseudo K-tuple reduced amino acids composition[J]. Bioinformatics, 2016, 33(1): 122-124.
Liu Y, Gong W, Yang Z, et al. SNB-PSSM: A spatial neighbor-based PSSM used for protein-RNA binding site prediction[J]. J Mol Recognit, 2021, 34(6): e2887.
Ruan X, Zhou D, Nie R, et al. Predictions of apoptosis proteins by integrating different features based on improving pseudo-position-specific scoring matrix[J]. Biomed Res Int, 2020, 2020: 4071508.
Lecun Y, Bengio Y, Hinton G. Deep learning[J]. Nature, 2015, 521(7553): 436.
周飛燕, 金林鵬, 董軍. 卷積神經網絡研究綜述[J]. 計算機學報, 2017, 40(6): 1229-1251.
楊麗, 吳雨茜, 王俊麗, 等. 循環神經網絡研究綜述[J]. 計算機應用, 2018, 38(S2): 1-6, 26.
Yu Y, Si X, Hu C, et al. A review of recurrent neural networks: LSTM cells and network architectures[J]. Neural Comput, 2019, 31(7): 1235-1270.
Dean S N, Alvarez J, Dan Z, et al. PepVAE: Variational autoencoder framework for antimicrobial peptide generation and activity prediction[J]. Front Microbiol, 2021, 12: 725727.
Torres M, Fuente-Nunez C. Reprogramming biological peptides to combat infectious diseases[J]. Chem Commun, 2019, 55(100): 15020-15032.
Puentes P R, Henao M C, Torres C E, et al. Design, screening, and testing of non-rational peptide libraries with antimicrobial activity: In silico and experimental approaches[J]. Antibiotics-Basel, 2020, 9(12): 854.
Sushko I, Salmina E, Potemkin V A, et al. ToxAlerts: A web server of structural alerts for toxic chemicals and compounds with potential adverse reactions[J]. J Chem Inf Model, 2012, 52(8): 2310-2316.
Tan Y S, Lane D P, Verma C S. Stapled peptide design: Principles and roles of computation[J], Drug Discov Today, 2016, 21(10): 1642-1653.
Cai C, Wang S, Xu Y, et al. Transfer learning for drug discovery[J]. J Med Chem, 2020, 63(16): 8683-8694.
Wei L, Ye X, Xue Y, et al. ATSE: A peptide toxicity predictor by exploiting structural and evolutionary information based on graph neural network and attention mechanism[J]. Brief Bioinform, 2021, 22(5):? bbab041.
Melo M, Maasch J, De La Fuente-Nunez C. Accelerating antibiotic discovery through artificial intelligence[J]. Commun Biol, 2021, 4(1): 1050.

