轉錄組分析人工配合飼料養殖對鱖魚腸道組織基因表達的影響
基金項目:國家自然科學基金項目(32303030);河北省自然科學基金(C2021204089;V1654588161890;236Z6701G);河北省高等學校科學技術研究項目(BJK2024012);河北省淡水養殖創新團隊名優特種類繁育及綠色高效養殖(HBCT2023230205)。
作者簡介:羅佳興(2000-),男,碩士研究生,研究方向:魚類遺傳育種。E-mail: bhcdljx@163.com。
通訊作者:孫硯峰(1981.04-),男,副教授,研究方向:水域環境生態。E-mail: sunyanfeng@hebau.edu.cn。
DOI:10.3969/j.issn.1004-6755.2024.05.001
摘" 要:為探究鱖魚(Siniperca chuatsi)腸道組織對飼料喂養的適應性,以飼料馴化30 d不吃飼料的鱖魚和飼料馴化30 d吃飼料的鱖魚為試驗群體,基于RNA-seq技術對鱖魚腸道組織的轉錄組數據進行分析。RNA-seq測序共獲得41.65 Gb Clean Data,Q30堿基百分比在90%以上。在FDR≤0.05且︱log2FoldChange︱≥1的篩選條件下轉錄組測序分析共鑒定出955個差異表達基因。GO功能富集分析結果顯示,差異表達基因主要在有機酸代謝過程上顯著富集。KEGG通路富集分析結果顯示,差異基因主要在細胞周期、PPAR信號通路等通路中被顯著富集。綜上所述,鱖魚為適應飼料養殖調動了與飲食有關的信號通路基因的表達,提高鱖魚腸道組織對人工配合飼料的消化、吸收能力。
關鍵詞:翹嘴鱖(Siniperca chuatsi);飼料養殖;轉錄組
翹嘴鱖(Siniperca chuatsi),別名桂魚、季花魚,肉味鮮美、營養豐富,屬于兇猛、肉食性淡水魚類。2022年,我國鱖魚養殖總產量為40.1萬t,相比于2021年增長7.36 %。鱖魚在養殖中主要以投喂的高蛋白、高脂肪的活魚活蝦為食。因此,鱖魚養殖過程中充足活餌料魚的捕撈、寄生蟲疾病的高效預防,成為制約鱖魚產業快速高效發展的主要因素。為此,國內學者從飼料馴化遺傳改良上來改變鱖魚攝食習性,降低鱖魚養殖成本。飼料鱖魚養殖相比于投喂活魚餌養殖鱖魚,養殖成本低、疾病預防可控,可以有效減少對養殖水體的環境污染。但是,鱖魚獨特的生物學特性(自開口起以活魚餌為食),以及“天生糖尿病患者”,導致飼料養殖過程易得脂肪肝,腸道壁變薄,造成飼料鱖魚養殖中代謝紊亂引發疾病,從而嚴重制約鱖養殖產業中發展。本研究運用轉錄組學相關技術,對飼料馴化30 d的翹嘴鱖腸道組織進行轉錄測序,分析飼料鱖魚腸道組織基因表達模式,探究調控鱖魚腸道消化吸收飼料的關鍵功能基因。旨在為飼料鱖魚新品種的選育提供理論基礎。
1" 材料與方法
1.1" 試驗材料
選用安徽省池州一絕生態養殖有限公司種苗基地飼料馴化30 d的鱖魚:對照組(C)是飼料馴化30 d不吃飼料的鱖魚;試驗組(T)是飼料馴化30 d吃飼料的鱖魚。分別選取對照組和試驗組各3尾麻醉解剖,取出腸道組織進行清洗后放入液氮中速凍30 min,之后置于-80 ℃保存。
1.2" 分析方法
將采集的腸道組織樣品送至武漢臻閱生物有限公司進行高通量測序。測序完成后,使用fastp對下機Rawdata(原始數據)進行質控、去除接頭,得到Cleandata(過濾后數據)。在NCBI(https://www.ncbi.nlm.nih.gov/)下載參考基因組(GCF_020085105.1),使用HISAT2進行比對得到映射數據,featureCounts進行基因表達定量。DESeq2和edgeR進行差異表達分析,選擇FDR<0.05和︱log2FoldChange︱≥1的基因作為差異基因。使用clusterProfile對差異基因進行GO和KEGG富集分析。
1.3" 熒光定量PCR驗證
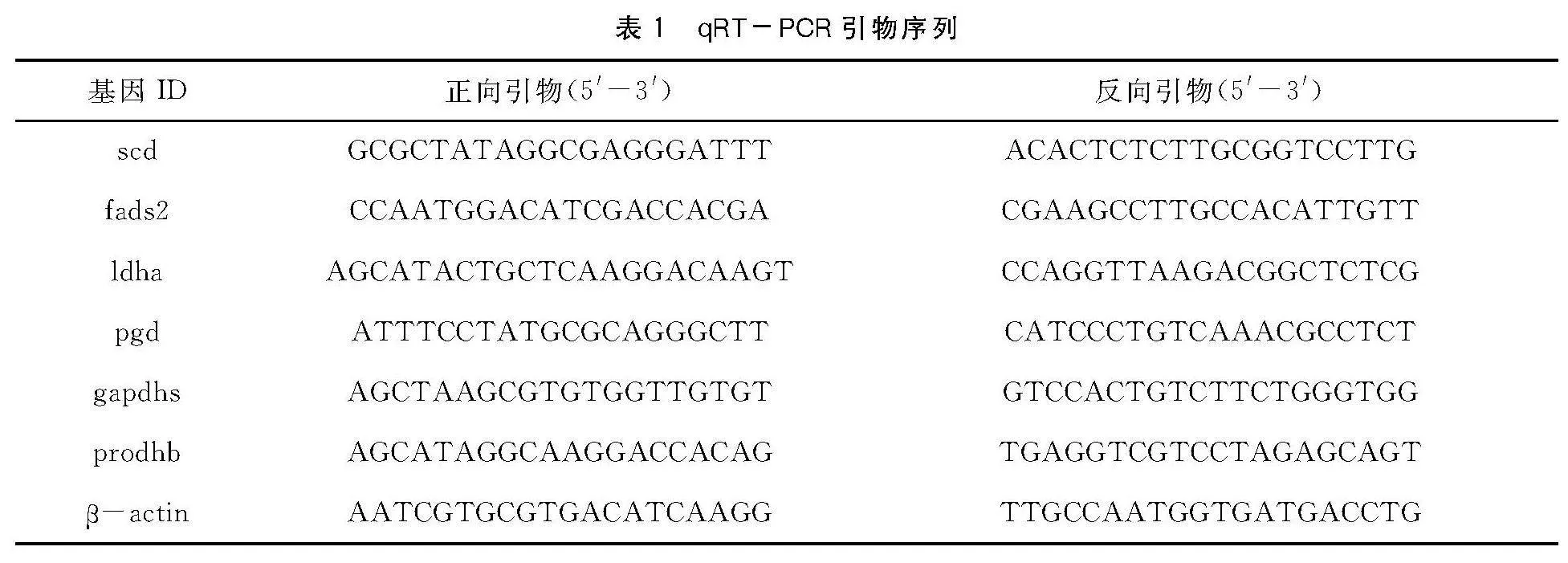
選取6個關鍵差異表達基因進行qRT-PCR 分析驗證,分別提取腸道組織的總RNA,隨后反轉錄合成cDNA,使用NCBI-blast在線設計引物(表1)。以β-actin為內參基因,熒光定量 PCR 儀是Bio-Rad(CFX96),反應程序:95 ℃預變性30 s,然后 95 ℃變性10 s、60 ℃退火30 s(39個循環),65 ℃延伸5 s。采用2-△△Ct值計算每個基因的相對表達量。
1.4" 數據分析
qRT-PCR所得數據用平均值±標準差表示,后用單因素方差分析區別組織和胚胎的差異,用T檢驗來進行顯著性判斷,P<0.01表示差異極顯著。
2" 結果
2.1" 轉錄組測序結果
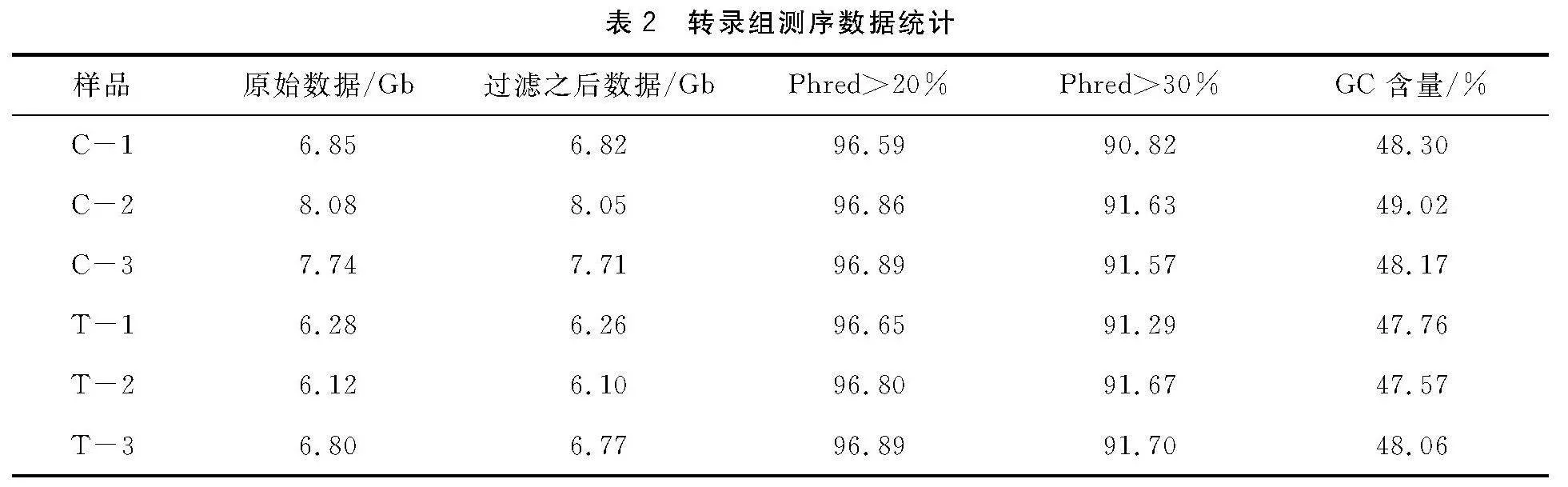
為了探究飼料養殖對鱖魚腸道基因表達的影響,對飼料鱖魚和未馴化成功鱖魚腸道組織進行了轉錄組測序。轉錄組測序共獲得41.65 Gb Clean Data,Q30堿基大于90%,GC含量在47%~50%,轉錄組測序質量較好,測序數據符合后續的基因功能注釋和功能分析要求(表2)。
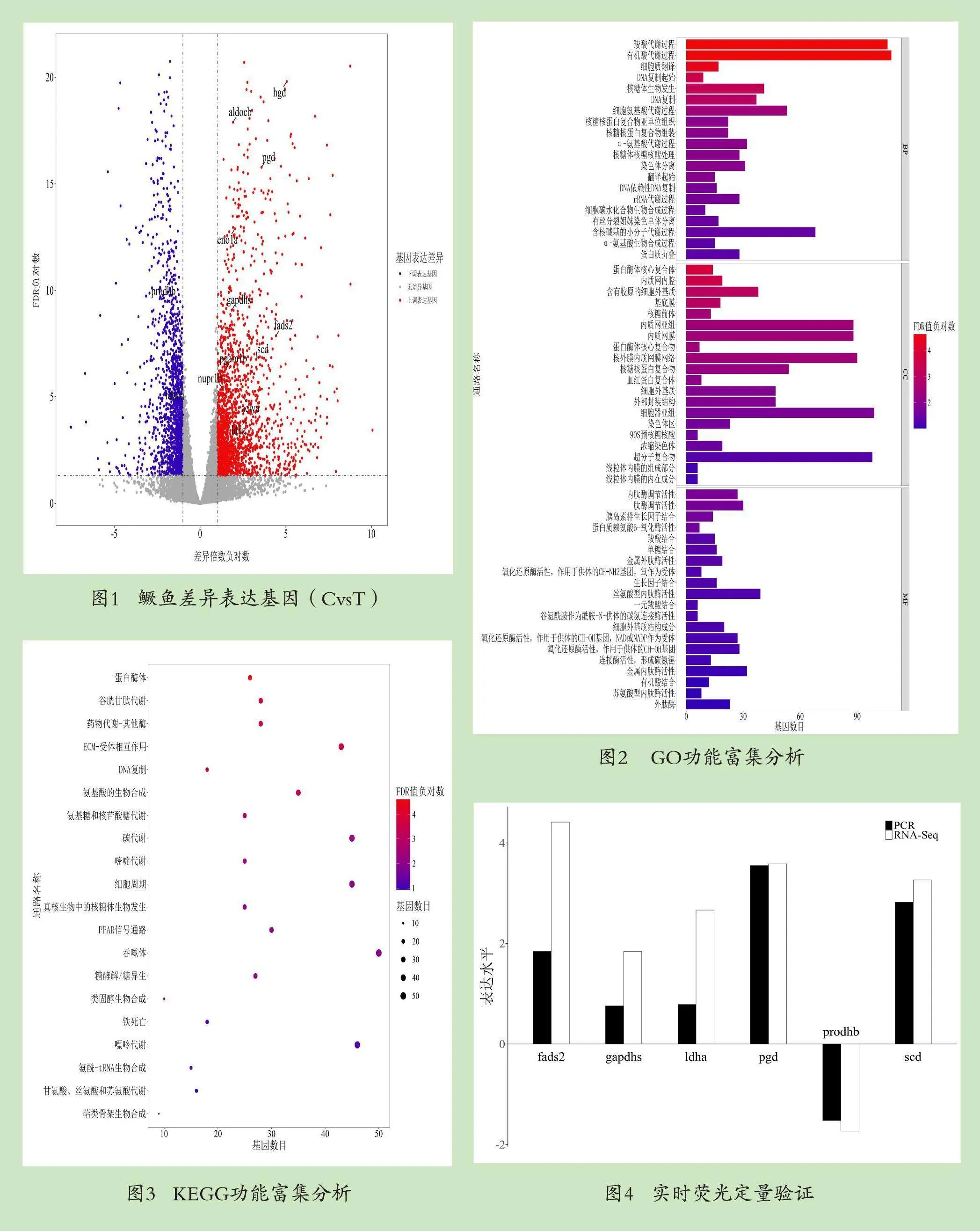
通過對照組和試驗組養殖鱖魚對比,按差異倍數(FoldChange≥2)和校正后的P值(FDR<0.05)篩選差異表達基因,得到955個差異表達基因,包括531個下調基因和424個上調基因(見封三圖1)。對差異表達基因進行GO富集分析,共映射到3 340條GO terms(GO條目)中,使用FDR多重檢驗對P值進行校正,共得到72條顯著富集的GO terms(FDR<0.05)。顯著富集的通路中在分子功能(Molecular Function,MF)中富集較多,共24條,在生物過程(Biological Process,BP)和細胞成分(Cellular Component,CC)中分別富集30和18條(見封三圖2)。其中有機酸代謝過程、羧酸代謝過程等通路富集最為顯著。
2.2" KEGG功能富集及功能基因表達量驗證
通過KEGG富集分析得到了150個富集通路,經過FDR多重檢驗對P值進行校正,共得到17條顯著的富集通路(FDR<0.05)。其中包含了細胞周期(Cell cycle,ko04110)、碳代謝(Carbon metabolism,ko01200)、PPAR信號通路(PPAR signaling pathway,ko03320)、糖酵解/糖異生(Glycolysis /Gluconeogenesis,ko00010)等與飲食有關的信號通路(見封三圖3)。
為了驗證轉錄組數據的準確性,使用實時熒光定量PCR技術檢測了6個基因的相對表達情況。實時熒光定量PCR結果與轉錄組測序分析結果趨勢相同(見封三圖4),說明轉錄組測序結果可靠。
3" 討論與分析
鱖魚屬于典型肉食性魚類,通過人工飼料馴化可以使鱖魚攝食人工配合飼料,馴化后的飼料鱖魚養殖可以極大程度提高鱖魚養殖效益。鱖魚腸道是對食物進行消化、吸收的主要場所,而幽門盲囊是增加消化吸收面積所形成的特殊結構。此外,腸道作為消化食物、吸收營養的器官,對魚類食性具有結構和功能上的適應。鱖魚的消化系統與典型的肉食性魚類相同,腸壁較厚,消化道較短。近幾年,對于飼料鱖魚的基因組學、轉錄組學、代謝組學研究逐漸增多。通過對大西洋鮭魚(Salmo salar)、鱖魚(Siniperca chuatsi)、淡水石首魚(Aplodinotus grunniens)等肉食性魚類的研究發現,經過飼料馴化后其不同組織的基因表達會發生顯著變化。因此,本研究通過對飼料鱖魚和未馴化成功鱖魚腸道組織進行轉錄組分析,以探究翹嘴鱖適應攝食飼料的腸道功能基因調控機制。
本研究發現scd基因在調控翹嘴鱖適應飼養養殖過程中發揮重要作用。Qiang等研究發現scd基因的高表達會使羅非魚的體重更快地增長。Wu等研究表明敲除scd基因可以讓斑馬魚表現出抑制攝食的行為。Guo等通過喂食不同的飼料發現,金鯧幼魚在攝食不同飼料的情況下scd基因的表達量不同。此外,科研人員通過研究發現飼料中添加多不飽和脂肪酸會抑制scd基因的表達。scd基因調控著單不飽和脂肪酸的合成,在多種魚中都參與著調控脂肪酸合成、脂質代謝等重要生理過程。本研究通過轉錄組測序來研究飼料馴化后翹嘴鱖scd基因發現,在飼料馴化后scd基因表達量顯著升高,因此scd基因在鱖魚適應攝食飼料過程中也發揮了關鍵作用。
通過GO功能富集分析結果發現,有機酸代謝過程(organic acid metabolic process)、羧酸代謝過程(carboxylic acid metabolic process)、氧代酸代謝過程(oxoacid metabolic process)、細胞外和內質網(Extracellular and endoplasmic reticulum network)、內質網膜(endoplasmic reticulum membrane)、內質網亞組(endoplasmic reticulum subcompartment)信號通路具有顯著差異。通過KEGG富集分析發現谷胱甘肽代謝(Glutathione metabolism)、氨基酸的生物合成(Biosynthesis of amino acids)、碳代謝(Carbon metabolism)、糖酵解/糖異生(Glycolysis / Gluconeogenesis)、PPAR信號通路(PPAR signaling pathway)等信號通路被顯著富集。內質網是細胞內脂類合成和處理的場所。目前已有研究表明PPAR信號通路與脂質代謝有關。在本研究中差異基因在PPAR信號通路上顯著富集(FDR<0.01),其中scd基因與fads2基因顯著高表達。其中fads2基因的高表達與Li等的研究結果一致。糖酵解/糖異生是脂質代謝的關鍵途徑,本研究中糖酵解/糖異生信號通路顯著富集,且該通路上的基因ldha、gapdhs顯著高表達。
此外,在GO富集分析中發現與scd、pgd、fads2、gapdhs基因在同一通路內的prodhb基因顯著下調,且prodhb基因富集在酸代謝、內質網等信號通路中,但在目前的研究中并未發現此基因的明顯變化。prodhb基因調控著脯氨酸脫氫酶(Proline dehydrogenase,Prodhb)的合成。正常濃度的脯氨酸可以清除細胞中的活性氧,調控細胞生長發育和細胞凋亡,在被催化過程中還可以產生ATP來提供能量。因此,我們推測prodhb基因的下調也對翹嘴鱖適應飼料養殖發揮了關鍵作用,其調控機制值得進一步研究。
綜上,我們推測翹嘴鱖飼料馴化過程中scd、pgd、fads2、gapdhs、ldha基因高表達,prodhb基因被抑制是翹嘴鱖適應飼料養殖的關鍵基因。
4" 結論
通過對飼料馴化后的翹嘴鱖腸道組織進行轉錄組測序分析發現,穩定攝食飼料的翹嘴鱖基因發生顯著變化。我們共篩選出3 686個差異基因,其中差異最顯著的是酸代謝相關基因。關鍵通路為糖酵解/糖異生和PPAR信號通路。我們的研究表明,scd、fads2、ldha、gapdhs、pgd、prodhb等功能基因在鱖魚適應飼料養殖過程中調控腸道組織對飼料營養物質的吸收方面共同產生調控,使翹嘴鱖適應攝食飼料。本研究為探索翹嘴鱖的飼料馴化以及后續的基因功能研究提供了一定的參考。
參考文獻:
農業農村部漁業漁政管理局,全國水產技術推廣總站,中國水產學會.2023中國漁業統計年鑒.北京:中國農業出版社,2023:15-36.
郭薇,尹恒,莫愛杰,等. 飼料蛋白水平對鱖生長、消化和代謝的影響. 華中農業大學學報,2023,42(4):215-224.
王貴英,曾可為,鄭翠華,等. 飼料脂肪水平對鱖魚生長的影響研究.淡水漁業,2003(2):11-12.
HE S,LIANG X F,SUN J,et al.Insights into food preference in hybrid F1 of Siniperca chuatsi (♀)×Siniperca scherzeri (♂) mandarin fish through transcriptome analysis.BMC genomics,2013,14(1): 1-11.
游俊杰.鱖魚馴食飼料相關組蛋白甲基化修飾研究.武漢:華中農業大學,2021.
LIU S,XU P,LIU X,et al.Production of neo‐male mandarin fish Siniperca chuatsi by masculinization with orally administered 17α-methyltestosterone.Aquaculture,2021,530:735904.
國家特色淡水魚產業技術體系.中國鱖魚產業發展報告.中國水產,2021(4):23-32.
汪福保,程光兆,孫成飛,等.池塘鱖魚人工配合飼料生態養殖技術.中國水產,2022(11):82-84.
趙亮亮,施琳瀾,趙金良,等.鱖幽門盲囊早期發育的組織學特征.上海海洋大學學報,2021,30(4):684-690.
MARTIN S A M,DEHLER C E,KRL E.Transcriptomic responses in the fish intestine.Developmental amp; Comparative Immunology,2016,64:103-117.
張三珊,劉海粟,林妙華,等.大蒜和茯苓對草魚幼魚腸道結構、功能及腸道微生物群落的影響.飼料工業,2021,42(22):6-13.
楊秀平,鄒海,趙愛平.鱖魚消化道組織學的初步研究.華中農業大學學報,1993(1):58-63+95-96.
LI L,HE S,LIN M H,et al.Whole-genome resequencing and bisulfite sequencing provide new insights into the feeding habit domestication in mandarin fish (Siniperca Chuatsi).Frontiers in Genetics,2023,13:1088081.
HE S,YOU J J,LIANG X F,et al.Transcriptome sequencing and metabolome analysis of food habits domestication from live prey fish to artificial diets in mandarin fish (Siniperca Chuatsi).BMC Genomics,2021,22(1):129.
JIN Y,OLSEN R E,HARVEY T N,et al.Comparative transcriptomics reveals domestication‐associated features of Atlantic salmon lipid metabolism.Molecular Ecology,2020,29(10):1860-1872.
XIAO Q,LI J,LIANG X F,et al.Programming of high-glucose diet acceptance in Chinese perch (Siniperca Chuatsi) following an early exposure.Aquaculture Reports,2020,18:100534.
SONG C,WEN H,LIU G,et al.Gut microbes reveal pseudomonas medicates ingestion preference via protein utilization and cellular homeostasis under feed domestication in freshwater drum, Aplodinotu sgrunniens.Frontiers in Microbiology,2022,13:861705.
QIANG J,TAO Y F,BAO J W,et al.High fat diet-induced miR-122 regulates lipid metabolism and fat deposition in genetically improved farmed tilapia (GIFT, Oreochromis niloticus) liver.Frontiers in Physiology,2018,9:1422.
WU W,SUN B,HE H,et al.scd knockout activates β-oxidation of fatty acids via accumulating stearic acid (18:0) and induces anorexia in zebrafish.Gene,2023,871:147431.
GUO H,CHEN C,YAN X,et al.Effects of different dietary oil sources on growth performance, antioxidant capacity and lipid deposition of juvenile golden pompano Trachinotus ovatus.Aquaculture,2021,530:735923.
REN H T,ZHANG X N,WANG Q Z,et al.Diet high in α-linolenic acid up-regulates fatty acids biosynthesis-relate gene expression in the hepatopancreas of Yellow River carp (Cyprinus carpio).Israeli Journal of Aquaculture - Bamidgeh, 2022,74(3):1-8.
MONIRUJJAMAN M,RENANI L B,ISESELE P,et al.Increased expression of hepatic stearoyl-CoA desaturase (SCD)-1 and depletion of eicosapentaenoicacid (EPA) content following cytotoxic cancer therapy are reversed by dietary fish oil.International Journal of Molecular Sciences,2023,24(4):3547.
DOBRZYN P,DOBRZYN A,MIYAZAKI M,et al.Stearoyl-CoA desaturase 1 deficiency increases fatty acid oxidation by activating AMP-activated protein kinase in liver. Proceedings of the National Academy of Sciences,2004,101(17):6409-6414.
MAEDA H,HOSOMI R,KOIZUMI M,et al.Dietary cod protein decreases triacylglycerol accumulation and fatty acid desaturase indices in the liver of obese type-2 diabetic KK-Ay mice.Journal of Functional Foods,2015,14:87-94.
QIN Y,HE L,WANG Y,et al.Growth performance, fatty acid composition, and lipid metabolism are altered in groupers (Epinephelus coioides) by dietary fish oil replacement with palm oil.Animal Nutrition,2022,8:102-113.
FU Z,ZHOU S,YU G,et al.Influence of non-protein diets on hepatic metabolism and endocrine in barramundi (Lates calcarifer).Frontiers in Marine Science,2021,8:649747.
GMEZ A M,BARTELTA.Endocrine communication of endoplasmic reticulum stress.BioEssays,2023,45(8):2300093.
ZHENG W,SUN Q,LI L,et al.Role of endoplasmic reticulum stress in hepatic glucose and lipid metabolism and therapeutic strategies for metabolic liver disease.International Immunopharmacology,2022,113:109458.
WU N,WEN H,XU P,et al.PPAR Signaling maintains metabolic homeostasis under hypothermia in freshwater drum (Aplodinotus grunniens).Metabolites,2023,13(1):102.
WANG Y,PAN Y,HOU M,et al.Danggui Shaoyao San ameliorates the lipid metabolism via the PPAR signaling pathway in a Danio rerio (zebrafish) model of hyperlipidemia.Biomedicine amp; Pharmacotherapy,2023,168:115736.
LI L,LIANG X F,CAI W,et al.Dietary with proper ratio of alpha-linolenic acid to linoleic acid enhanced the unsaturated fatty acids deposition of Chinese perch (Siniperca Chuatsi).Aquaculture Nutrition,2021,27(S1):73-85.
GE K,GENGZ.Proteomic analysis of the liver regulating lipid metabolism in Chaohu ducks using two-dimensional electrophoresis.Open Life Sciences,2022,17(1):960-972.
LIU W,LE A,HANCOCK C,et al.Reprogramming of proline and glutamine metabolism contributes to the proliferative and metabolic responses regulated by oncogenic transcription factor c-MYC.Proceedings of the National Academy of Sciences,2012,109(23):8983-8988.
劉玉霞,竇世娟,王秀伶.細菌中脯氨酸的生物合成、降解及功能.微生物學報,2021,61(11):3351-3362.
XU X,ZHANG G,CHEN Y,et al.Can proline dehydrogenase—a key enzyme involved in proline metabolism—be a novel target for cancer therapy?.Frontiers in Oncology, 2023,13:1254439.
Transcriptomic analysis of the impact of artificial compound feed cultivation on gene expression in the intestinal tissue of Mandarin fish
LUO Jiaxing1,GAO Xiaotian2,ZHANG Lihan1,SUN Yanfeng1,WU Chengbin1
(1. Ocean College, Hebei Agricultural University, Qinhuangdao 066003, China; 2. Hebei Academy of Marine and Fishery Sciences (Hebei Marine Fisheries Ecological Environment Monitoring Station), Qinhuangdao 066003, China)
Abstract:To investigate the adaptability of mandarin fish (Siniperca chuatsi) intestinal tissue to feed intake, two groups of fish were fed with feed for 30 days.The control group had not been fed with artificial feed before. The experimental group of fish had been tamed to feed. Utilizing RNA-seq technology, transcriptomic data from the intestinal tissues of mandarin fish were analyzed. The RNA-seq sequencing yielded a total of 41.65 Gb of clean data, with a Q30 base percentage exceeding 90%. Transcriptome sequencing analysis identified 955 differentially expressed genes under the screening criteria of FDR≤0.05 and ︱log2FoldChange︱≥1. GO functional enrichment analysis revealed significant enrichment of differentially expressed genes primarily associated with organic acid metabolism processes. Additionally, KEGG pathway enrichment analysis demonstrated significant enrichment of differential genes in pathways such as the cell cycle and the PPAR signaling pathway. In summary, to adapt to aquaculture feeding, mandarin fish activated the expression of genes related to dietary signaling pathways, thereby enhancing the digestive and absorptive capacities of intestinal tissue for artificial compound feeds.
Key words:Siniperca chuatsi;feed breeding;RNA-seq
(收稿日期:2024-04-04)

