梅花鹿源彭氏變形桿菌F2d 株基因組的測序分析
摘 要:為探究鹿源彭氏變形桿菌(Proteus penneri)致病及耐藥機制,利用二代測序技術對從云南野生動物園病死梅花鹿(Cervus nippon)肺組織中分離鑒定出的1株彭氏變形桿菌F2d株進行基因組測序,將獲得的F2d株基因組序列注釋到毒力因子、耐藥基因和代謝通路等數據庫中。毒力因子數據庫注釋結果顯示,菌株含黏附、營養代謝、運動和免疫調節等類型毒力因子;耐藥基因數據庫注釋結果顯示,F2d株對β-內酰胺類、四環素類、黏菌素類和磺胺類等多種抗生素耐藥,并且攜帶多種抗生素外排機制編碼基因;代謝通路數據庫注釋結果顯示,注釋的基因富集在42個通路上,其中新陳代謝相關注釋的基因數量最多,共有2 250個基因分布在12個通路上。利用基因組測序技術分析鹿源彭氏桿菌F2d株致病性、耐藥情況及代謝特點,可為預防彭氏變形桿菌感染和相關藥物的研發提供理論依據。
關鍵詞:基因組測序;梅花鹿;彭氏變形桿菌;毒力與耐藥基因;功能分析
中圖分類號:S852. 61
文獻標識碼:A
文章編號:2310 - 1490(2024)- 03 - 0513 - 08
DOI:10.12375/ysdwxb.20240307
彭氏變形桿菌(Proteus penneri)隸屬于腸桿菌科(Enterobacteriaceae)變形桿菌屬(Proteus)[1],是一種人獸共患條件性致病菌,侵染人可引起原發性或繼發性食管、呼吸道、腸道和尿路感染并引發敗血癥[2?3]。在動物上,Cao 等[4]首次在凡納濱對蝦(Penaeus vannamei)中分離鑒定出1株高致病性彭氏變形桿菌。曾晨爔等[5]研究發現彭氏變形桿菌可引起暹羅鱷(Crocodylus siamensis)幼鱷食管結節和敗血癥,并造成部分幼鱷死亡,該菌對巴西龜(Trache?mys scripta)的致病力較強,與感染暹羅鱷幼鱷的結果一致。
2021年11月云南省野生動物園1頭6月齡梅花鹿(Cervus nippon)突然死亡,死前主要表現為抽搐和神經癥狀。剖檢發現心包積液,肺臟淤血、充血和實化,肝和腎腫大、充血,其他器官無肉眼可見的病理變化。病死鹿的病理學觀察發現:肺泡毛細血管充血,肺泡壁增厚,肺泡內有脫落的組織碎片,肺間質水腫;肝毛細血管充血,明顯水腫,肝小葉結構紊亂,出現假小葉;腎小球毛細血管充血,腎小管上皮發生壞死、脫落。此后,對其肺、腎和肝等組織進行病原菌分離,經16S rDNA鑒定為彭氏變形桿菌。
細菌感染是影響梅花鹿保護和馴養的重要因素之一,現已報道的梅花鹿源致病菌有化膿隱秘桿菌(Trueperella pyogenes)[6]、肺炎克雷伯氏菌(Klebsiellapneumoniae)[7]、多殺性巴氏桿菌(Pasteurella multo?cida)[8]、產氣莢膜梭菌(Clostridium perfringens)[9]、布氏桿菌(Brucella sp.)[10]和大腸埃希氏菌(Esch?erichia coli)[11]等,但尚未有梅花鹿感染彭氏變形桿菌的報道。
目前,NCBI數據庫中共有12條彭氏變形桿菌基因組數據記錄,其來源包括人(Homo sapiens)、馬來穿山甲(Manis javanica)、紅原雞(Gallus gallus)和豬(Sus sp.)等,數據庫中有相應基因組的注釋信息,但未搜索到相關基因組學文獻的報道。全基因組測序可有效整合利用細菌的全基因組信息,在研究細菌重要特性和變異進化方面有很高的分辨率[12]。本研究利用全基因組測序技術,對云南野生動物園病死梅花鹿肺中分離鑒定的彭氏變形桿菌F2d株進行基因組測序分析,以期獲得該菌株的完整基因組信息,確定其攜帶的毒力基因、耐藥基因等生物學特征,進一步揭示致病機制、耐藥機制,為預防彭氏變形桿菌感染及相關藥物的研發提供理論基礎。
1 材料與方法
1. 1 材料
彭氏變形桿菌分離自云南省野生動物園病死梅花鹿的肺臟組織,由西南林業大學動物科學實驗室保存。
1. 2 菌種處理
在LB液體培養基中,37 ℃、180 r/min培養24 h得到彭氏變形桿菌F2d株菌懸液,經4 ℃、6 000 r/min離心10 min獲得菌體,用于后期研究。
1. 3 菌株基因組測序、組裝和分析
將凍存樣品置于干冰中,送至上海派森諾生物科技股份有限公司進行全基因組測序,將獲得的原始測序數據過濾優化[13],采用A5-miseq[14]和SPAdes[15]對去除接頭序列的測序數據進行從頭拼裝,構建contig和scaffold。對得到的拼裝結果進行評估和比較,并使用Pilon[16]軟件進行堿基校正。
1. 4 功能元件預測
采用CRISPRCasFinder、GeneMarkS、tRNAscan-SE、Barrnap和Rfam對F2d株中的非編碼RNA、CRISPRs等功能元件進行預測。
1. 5 細菌毒力因子及耐藥基因檢測
將F2d 株基因組序列與毒力基因數據庫(virulencefactor database,VFDB)和抗生素耐藥基因數據庫(the comprehensive antibiotic research database,CARD)進行比對[17?18],檢測基因組中存在的與毒力因子和抗生素抗性相關的基因。
1. 6 基因功能分析
將F2d 株基因組序列分別與CAZy、COG、KEGG、GO、TCDB和PHI等數據庫進行BLAST比對,從而獲得預測基因的注釋信息。
2 結果
2. 1 基因測序與組裝
基于Illumina NovaSeq測序平臺,使用第二代測序技術對彭氏變形桿菌F2d進行基因組測序,去除接頭及低質量的reads后從頭拼裝,總共獲得226個scaffolds。F2d 株基因組序列總長為7 238 711 bp,N20、N50、N90長度依次為170 398、88 108、23 492 bp,最長序列和最短序列分別為259 928 、525 bp,GC值為47. 04%。
2. 2 功能元件分析
對獲得的F2d株基因組進行功能元件分析,采用GeneMark. hmm建立相應的統計模型,對基因組中蛋白質編碼基因預測,共獲得7 024個蛋白質編碼基因,總大小為6 264 870 bp,占基因組總大小的86. 55%,平均大小為891. 92 bp。
對F2d株中主要的非編碼RNA類型進行預測,其中rRNA 7 個(3 個5S rRNA,2 個16S rRNA,2 個23S rRNA),tRNA 104個,其他ncRNA 212個。
CRISPRs存在于原核生物基因組中,能夠給宿主提供某種獲得性免疫。本次預測結果發現,F2d株中共存在8條CRISPRs結構。
2. 3 毒力因子及耐藥基因分析
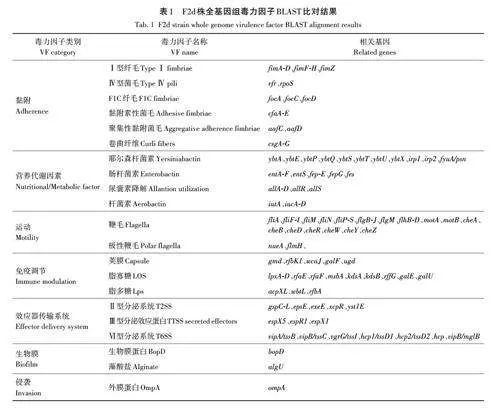
將F2d株全基因組在毒力因子數據庫VFDB中進行BLAST比對。從注釋結果可知,F2d株的毒力由多種類型的毒力因子介導,包括營養代謝、黏附、運動和免疫調節等(表1)。
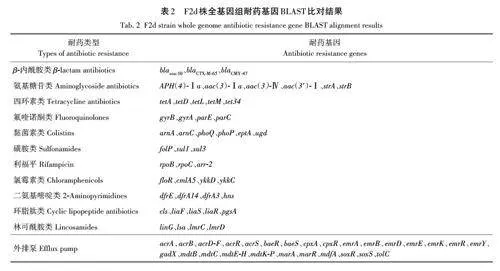
將F2d株基因組在耐藥基因數據庫CARD中進行比對,檢索出多種抗生素耐藥基因,包括β-內酰胺類(blaoxa-10、blaCTX-M-65、blaCMY-47)、四環素類(tetA、tetD、tetL、tetM、tet34)、黏菌素類(arnA、arnC、phoQ、phoP、eptA、ugd)、磺胺類(folP、sul1、sul3),以及多種外排泵編碼基因等(表2)。
2. 4 基因功能注釋
2. 4. 1 CAZy數據庫注釋
將F2d 株基因組序列與碳水化合物活性酶CAZy數據庫進行比對,共檢測出209個CAZy 酶類基因,其中糖苷水解酶類基因有86個,糖基轉移酶類基因有60個,碳水化合物酯酶類基因有37個,輔助活性酶類基因有12個,多糖裂解酶類基因有4個,此外,還包含與碳水化合物結合相關的酶類基因10個。
2. 4. 2 COG數據庫注釋
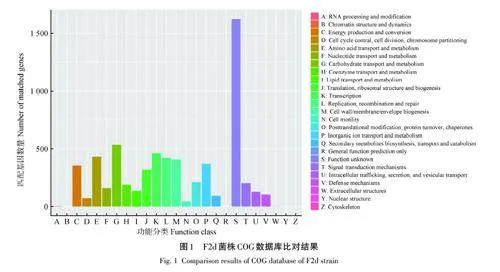
將F2d株基因組序列與COG蛋白數據庫進行比對,預測與已知蛋白具有同源性的蛋白,結果顯示共有6 228個基因注釋到20組COG 的功能中(圖1),4 610個蛋白與已知蛋白或假設蛋白同源,1 627個蛋白功能未知,匹配數量大于400的基因有:碳水化合物運輸與代謝相關蛋白535個,轉錄相關蛋白460個,氨基酸轉運與代謝相關蛋白432個,復制、重組和修復相關蛋白421個,細胞壁/膜/包膜生物發生相關蛋白405個。
2. 4. 3 KEGG數據庫注釋
將F2d 株基因組序列與KEGG 數據庫進行比對,預測基因編碼蛋白參與的生物通路。KEGG注釋結果顯示注釋的基因富集在六大分類的42個通路上。其中,細胞過程共有295個基因分布在4個通路上,環境信息處理共有589個基因分布在3個通路上,遺傳信息處理共有394個基因分布在4個通路上,人類疾病共有217個基因分布在11個通路上。新陳代謝注釋的基因數量最多,共有2 250個基因分布在12個通路上,遺傳信息處理方面共有81個基因分布在8個通路上。
2. 4. 4 GO數據庫注釋
將F2d株基因組序列與GO數據庫進行比對,共注釋出5 247個蛋白編碼基因。生物過程相關基因數量最多,為4 891個;其次是分子功能相關基因,共4 580個;最少的是細胞組分,為2 043個基因。
生物過程層面共有44種功能注釋,細胞氮化合物代謝過程、生物合成過程功能相關基因最多,分別為1 788、1 615個。細胞組分層面共有13種功能注釋,細胞組成及胞內組分相關基因最多,分別為1 604、1 168個。分子功能層面共有26種功能注釋,離子結合和DNA結合相關基因最多,分別為1 540、828個。
2. 4. 5 TCDB注釋
將F2d株基因組序列與TCDB數據庫進行比對,共有1 308個基因注釋到TCDB數據庫中,分別編碼通道與孔道(226個)、電化學勢驅動轉運體(380個)、主要活性轉運蛋白(576個)、轉錄組(142個)、跨膜電子載體(48個)、運輸時涉及的附屬因素(54個)和不完全表征的運輸系統(186個)。
2. 4. 6 PHI數據庫注釋
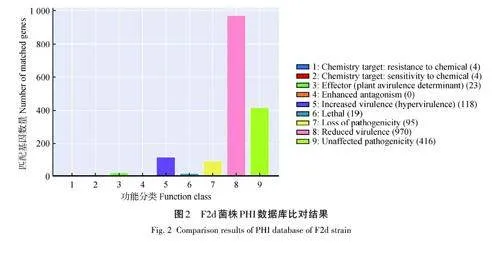
將F2d株基因組序列與PHI數據庫進行比對,共注釋到1 649個基因(圖2)。其中,970個基因突變后毒力減弱,416個突變后不受影響,118個基因突變后毒力增加,95個突變后喪失致病性,23個突變成效應子,19個突變后致死。
3 討論
全基因組測序分析是揭示菌株的潛在功能及相關分子機制的重要手段[19]。盡管在當前的NCBI數據庫中記錄了12 條彭氏變形桿菌的基因組數據(2 條完整),并有較詳細的基因組注釋,但基于PubMed 和ScienceDirect 未搜索到有關該菌基因組方面的文獻報道。基于基因組的注釋數據可分析F2d株基因組中的基因分布情況,進而對分離株的毒力和耐藥表型作出相應地推斷,故本研究利用二代測序技術,對鹿源彭氏變形桿菌進行基因組測序,將得到的基因組序列與VFDB、CARD、CAZy、COG、KEGG、GO、TCDB和PHI等多個數據庫進行比對,對其參與的代謝過程進行分析,重點對毒力因子和耐藥基因進行了分析。
微生物的致病性主要由毒力因子決定,毒力因子是致病的重要因素[20]。由毒力因子VFDB數據庫比對的結果可知,F2d株包含多種毒力因子,包括侵襲因子、黏附因子、營養代謝和免疫調節等。Ⅰ型菌毛在F2d株黏附相關的毒力因子中占比最高,包括fimA、fimC、 fimD、 fimF、 fimG 和fimH 等,這些基因所編碼的Ⅰ型菌毛可增強變形桿菌的黏附、定殖能力。有報道顯示,Ⅰ型菌毛是多種細菌的黏附因子,通過侵襲引發機體的免疫應答以及促進對宿主黏膜表面的黏附等途徑增強細菌的致病力,在病原菌侵襲宿主并引發感染的過程中起重要作用[21]。鐵離子是多種生化反應及電子傳遞等生物過程的輔助因子,是生物體必需的金屬元素,在自然界中,鐵元素主要以不溶性的氫氧化物或氧化物形式存在,且生物體內游離鐵的濃度較低,致病菌很難直接利用宿主體內的鐵離子。在F2d株中檢測出多種與鐵轉運相關的毒力因子,包括耶爾森桿菌素(Yersiniabactin)、腸桿菌素(Enterobactin)等,這類毒力基因編碼的YBT、ENT鐵轉運系統與鐵的結合力較高,能夠從轉鐵蛋白和乳鐵蛋白中螯合鐵,在致病菌獲取鐵離子的過程中起重要作用[22]。
微生物的抗生素耐藥機制復雜多樣,是由多個生理生化過程共同協作形成,包括形成改變藥物作用靶位基因、細胞膜滲透性變化或孔蛋白孔徑變窄、滅活酶和外排泵排出抗生素等[23]。耐藥基因數據庫CARD 比對結果顯示,F2d 株可能擁有多種耐藥機制。F2d株中含有β-內酰胺酶編碼基因,如頭孢菌素酶編碼基因blaCMY-47、超廣譜β-內酰胺酶編碼基因blaCTX-M-65等,破壞抗生素分子的內部結構,使抗生素降解從而產生耐藥性[24?25]。氨基糖苷類耐藥基因在F2d株中也有所表達,包括氨基糖苷磷酸轉移酶編碼基因APH(4)-Ⅰa 以及氨基糖苷乙酰轉移酶編碼基因aac(3)-Ⅰa、aac(3)-Ⅳ、aac(3')-Ⅰ等。F2d株中含有多種多藥外排機制相關的編碼基因,其中,marR、acrR 基因不僅參與編碼RND外排泵,還改變抗生素靶點產生耐藥性,marA 編碼了RND外排泵蛋白的同時降低抗生素的通透性。soxR、tolC 基因編碼的蛋白與ABC、RND 和MFS 3 種外排泵功能有關;soxS 編碼蛋白不僅具有上述3種外排泵功能,同時還能改變抗生素靶點、降低抗生素通透性。多種耐藥基因共存會導致多重耐藥甚至泛耐藥的發生,降低獸用抗生素的治療效果,使細菌性感染更加難以治療,甚至在人與動物或環境接觸等途徑中傳播至人類,威脅人類的健康。
對彭氏變形桿菌F2d株基因功能分析,有助于預測該菌的生物學功能。微生物碳水化合物的結合結構域與CAZy的底物相結合后,可以提高CAZy催化底物的活性,加快反應速率[26]。從CAZy數據庫注釋結果得知,在F2d株碳水化合物活性酶中,糖苷水解酶和糖基轉移酶的數量最多,結構多糖可以被特定的糖苷水解酶水解,而合成天然糖基結構需要糖基轉移酶。高效的糖基轉移酶是天然產物糖基化的關鍵因素,為藥物的研發提供了可能性[27]。
將F2d株蛋白質編碼基因序列與COG、KEGG和GO數據庫注釋分析發現,代謝過程的基因富集程度最高,3個數據庫的注釋結果一致,這說明了代謝過程對于維持細菌的生命至關重要[28]。此外,COG注釋結果中發現F2d株有較多的與基因重組、轉移有關的基因,這提示毒力因子與抗性基因的整合會導致變異株的出現,進一步加強菌株的毒力或耐藥性。因此,為了研發對應藥物或提高疫苗的高效性,需要充分了解菌株的生物學信息[29]。
4 結論
本研究對云南野生動物園病死梅花鹿肺中分離的彭氏變形桿菌F2d株進行了基因組測序與生物信息學分析,并分析基因組攜帶的毒力因子、耐藥元件和代謝通路,發現分離株基因組中攜帶大量的毒力基因和耐藥基因,這暗示分離株具有高致病性和強耐藥性,研究結果豐富了梅花鹿源分離菌的資料。彭氏變形桿菌作為一種人獸共患條件性致病菌,在人和動物上的病例均有報道,了解該菌株基因組的結構和功能,分析與致病和耐藥機制中的相關基因,為預防和治療彭氏變形桿菌的感染提供理論基礎。
參考文獻:
[1] DRZEWIECKA D. Significance and roles of Proteus spp. bacteria
in natural environments[J]. Microbial Ecology, 2016, 72(4):
741-758.
[2] SIWI?SKA M, LEVINA E A, OVCHINNIKOVA O G, et al.
Classification of a Proteus penneri clinical isolate with a unique Oantigen
structure to a new Proteus serogroup, O80[J]. Carbohydrate
Research, 2015, 407: 131-136.
[3] 蔡晶, 楊一兵, 叢林海. 兩種腸道條件致病菌同時引起的喉部
感染一例[J]. 聽力學及言語疾病雜志, 2012, 20(4):
398-399.
CAI J, YANG Y B, CONG L H. A case of laryngeal infection
caused by two intestinal conditional pathogens at the same time
[J]. Journal of Audiology and Speech Pathology, 2012, 20(4):
398-399.
[4] CAO H P, HE S, LU L Q, et al. Identification of a Proteus
penneri isolate as the causal agent of red body disease of the cultured
white shrimp Penaeus vannamei and its control with Bdellovi?
brio bacteriovorus[J]. Antonie van Leeuwenhoek, 2014, 105(2):
423-430.
[5] 曾晨爔, 林茂, 李忠琴, 等. 暹羅鱷食道結節病病原彭氏變形
桿菌的分離與鑒定[J]. 微生物學通報, 2019, 46(7): 1629-
1635.
ZENG C X, LIN M, LI Z Q, et al. Isolation and identification of
pathogen Proteus penneri from Crocodylus siamensis suffering nodulous
esophagitis[J]. Microbiology China, 2019, 46(7): 1629-
1635.
[6] 李俊鋒, 趙自亮, 朱桓奕, 等. 1株梅花鹿源化膿隱秘桿菌的
分離鑒定及其生物學特性分析[J]. 中國獸醫學報, 2023, 43
(4): 720-725;740.
LI J F, ZHAO Z L, ZHU H Y, et al. Isolation, identification and
biological characteristics analysis of a strain of Trueperella pyo?
genes from sika deer[J]. Chinese Journal of Veterinary Science,
2023, 43(4): 720-725;740.
[7] 王煒強, 孫浩云, 楊志松. 動物園梅花鹿源肺炎克雷伯氏菌的
分離鑒定及耐藥性[J]. 野生動物學報, 2022, 43(1):
220-223.
WANG W Q, SUN H Y, YANG Z S. Isolation, identification and
drug resistance of Klebsiella pneumoniae isolated from sika deer
(Cervus nippon) in zoo[J]. Chinese Journal of Wildlife, 2022, 43
(1): 220-223.
[8] 劉守振, 任志華, 楊康, 等. 一起梅花鹿巴氏桿菌病的診治
[J]. 畜牧與獸醫, 2011, 43(10): 106-107.
LIU S Z, REN Z H, YANG K, et al. Diagnosis and treatment of
pasteurellosis in sika deer[J]. Animal Husbandry amp; Veterinary
Medicine, 2011, 43(10): 106-107.
[9] 羅柳, 湯德元, 曾智勇, 等. 梅花鹿感染產氣莢膜梭菌的診斷
及中西醫結合治療[J]. 野生動物學報, 2022, 43(1):
184-187.
LUO L, TANG D Y, ZENG Z Y, et al. Diagnosis and integrated
treatment of Clostridium perfringens infection in sika deer(Cervus
nippon)[J]. Chinese Journal of Wildlife, 2022, 43(1) :
184-187.
[10] 李文平, 郭延輝, 張鴻, 等. 一例梅花鹿布氏桿菌病的診治
[J]. 吉林畜牧獸醫, 2023, 44(7): 137-138.
LI W P, GUO Y H, ZHANG H, et al. Diagnosis and treatment
of a case of Cervus nippon brucellosis[J]. Jilin Animal Husbandry
and Veterinary Medicine, 2023, 44(7): 137-138.
[11] 冷闖, 張文波, 鄧舜洲, 等. 一例梅花鹿致病菌的分離、鑒定
及藥敏試驗[J]. 中國畜牧獸醫, 2012, 39(4): 207-211.
LENG C, ZHANG W B, DENG S Z, et al. Isolation, identification
and drug sensitive test of pathogenic bacterium from a spotted
deer[J]. China Animal Husbandry amp; Veterinary Medicine,
2012, 39(4): 207-211.
[12] 周江林, 彭小川, 胡明達, 等. 基于全基因組測序的細菌進
化研究進展[J]. 生物技術通訊, 2018, 29(6): 844-850.
ZHOU J L, PENG X C, HU M D, et al. Advances in bacterial
evolution study based on whole genome sequencing[J]. Letters
in Biotechnology, 2018, 29(6): 844-850.
[13] SCHUBERT M, LINDGREEN S, ORLANDO L. AdapterRemoval
v2: rapid adapter trimming, identification, and read merging
[J]. BMC Research Notes, 2016, 9: 88.
[14] COIL D, JOSPIN G, DARLING A E. A5-miseq: an updated
pipeline to assemble microbial genomes from Illumina MiSeq data
[J]. Bioinformatics, 2015, 31(4): 587-589.
[15] BANKEVICH A, NURK S, ANTIPOV D, et al. SPAdes: a new
genome assembly algorithm and its applications to single-cell sequencing
[J]. Journal of Computational Biology, 2012, 19(5):
455-477.
[16] WALKER B J, ABEEL T, SHEA T, et al. Pilon: an integrated
tool for comprehensive microbial variant detection and genome assembly
improvement[J]. PLoS One, 2014, 9(11): e112963.
[17] CHEN L H, YANG J, YU J, et al. VFDB: a reference database
for bacterial virulence factors[J]. Nucleic Acids Research,
2005, 33(Database issue): D325-D328.
[18] JIA B F, RAPHENYA A R, ALCOCK B, et al. CARD 2017:
expansion and model-centric curation of the comprehensive antibiotic
resistance database[J]. Nucleic Acids Research, 2017,
45(Database issue): D566-D573.
[19] BECK B R, PARK G S, LEE Y H, et al. Whole genome analysis
of Lactobacillus plantarum strains isolated from kimchi and
determination of probiotic properties to treat mucosal infections
by Candida albicans and Gardnerella vaginalis[J]. Frontiers in
Microbiology, 2019, 10: 433.
[20] KONG C, NEOH H M, NATHAN S. Targeting Staphylococcus
aureus toxins: a potential form of anti-virulence therapy[J]. Toxins,
2016, 8(3):72.
[21] 郭志燕, 周明旭, 朱國強. 細菌Ⅰ型菌毛結構及其致病性研
究進展[J]. 中國預防獸醫學報, 2014, 36(7): 577-581.
GUO Z Y, ZHOU M X, ZHU G Q. Research progress on the
structure and pathogenicity of bacterial type Ⅰ fimbriae[J]. Chinese
Journal of Preventive Veterinary Medicine, 2014, 36(7):
577-581.
[22] FETHERSTON J D, KIRILLINA O, BOBROV A G, et al. The
yersiniabactin transport system is critical for the pathogenesis of
bubonic and pneumonic plague[J]. Infection and Immunity,
2010, 78(5): 2045-2052.
[23] 杜靜怡, 王鋮, 郭君慧, 等. 細菌RND外排泵的結構與作用
機制研究進展[J]. 中國抗生素雜志, 2022, 47(10): 994-
1001.
DU J Y, WANG C, GUO J H, et al. Research progress in the
structure and function of RND efflux pump[J]. Chinese Journal
of Antibiotics, 2022, 47(10): 994-1001.
[24] MEINI S, TASCINI C, CEI M, et al. AmpC β -lactamaseproducing
Enterobacterales: what a clinician should know[J]. Infection,
2019, 47(3): 363-375.
[25] WILSON H, T?R?K M E. Extended-spectrum β -lactamaseproducing
and carbapenemase-producing Enterobacteriaceae
[J]. Microbial Genomics, 2018, 4(7): e000197.
[26] COSTA T R D, FELISBERTO-RODRIGUES C, MEIR A, et al.
Secretion systems in Gram-negative bacteria: structural and
mechanistic insights[J]. Nature Reviews Microbiology, 2015,
13: 343-359.
[27] BOSSé J T, LI Y W, ROGERS J, et al. Whole genome sequencing
for surveillance of antimicrobial resistance in Actinobacillus
pleuropneumoniae[J]. Frontiers in Microbiology, 2017, 8: 311.
[28] BOYCE J D, ADLER B. How does Pasteurella multocida respond
to the host environment?[J]. Current Opinion in Microbiology,
2006, 9(1): 117-122.
[29] LIN H L, FENG C L, ZHU T T, et al. Molecular mechanism of
the β-lactamase mediated β-lactam antibiotic resistance of Pseu?
domonas aeruginosa isolated from a Chinese teaching hospital
[J]. Frontiers in Microbiology, 2022, 13: 855961.

