檉柳根際土壤鏈霉菌Streptomyces sp. TRM76323基因簇分析及代謝產物分離
摘要:目的 利用鏈霉菌Streptomyces sp. TRM76323的基因組序列信息,分析其次級代謝產物生物合成基因簇并預測其代謝產物,為發現潛在新抗生素奠定基礎。方法 采用Illumina Novaseq對TRM76323菌株進行全基因組測序,結合antiSMASH、CARD、OrthoFinder、Peppan軟件進行基因組分析,進行代謝潛力、抗性基因和基因組差異預測,為指導新的微生物來源的天然產物的發現提供重要的理論指導,并利用現代分離技術獲得代謝產物。結果 TRM76323菌株的基因組序列由7,338,911 bp組成,G+C含量為72.1%,共31個生物合成基因簇,有糖肽類耐藥等抗性基因、分離獲得化合物β-羰基-1H-吲哚-3-丙醛、環二肽和鄰苯二甲酸二丁酯。結論 檉柳根際土壤鏈霉菌Streptomyces sp. TRM76323有豐富的次級代謝產物生物合成基因簇,能產生多種次級代謝產物,具有進一步發掘新抗生素的價值。
關鍵詞:檉柳根際;鏈霉菌;全基因組測序;次級代謝產物
中圖分類號:R978.1,Q939.1 文獻標志碼:A
Gene cluster analysis and metabolic product isolation of Streptomyces sp. TRM76323 in the rhizosphere soil of Tamarix
Abstract Objective This study analyzed the secondary metabolite biosynthesis gene clusters of Streptomyces sp. TRM76323 on the basis of its genomic sequence information, laying the foundation for the discovery of potential new antibiotics. Method The whole genome sequencing of the strain TRM76323. was performed by Illumina Novaseq. Additionally, antiSMASH, CARD, OrthoFinder and Pepperan softwares were used for genome analysis to predict metabolic potential, resistance genes, and genomic differences. These findings provided important theoretical guidance for the discovery of new natural products from microbial sources. Furthermore, modern isolation techniques were utilized to obtain metabolites. Results The genome sequence of the TRM76323 strain consisted of 7,338, 911-base pairs, with a G+C content of 72.1%. A total of 31 biosynthetic gene clusters were identified, including resistance genes such as glycopeptide resistance. β-Carbonyl-1H-indole-3-propanal, cyclic dipeptide, and dibutyl phthalate were isolated from this strain. Conclusion Streptomyces sp. TRM76323, found in the rhizosphere soil of Tamarix, possessed a diverse clusters of secondary metabolite biosynthesis genes. This strain had the potential to produce various secondary metabolites and was worth further exploration for the discovery of new antibiotics.
Key words Rhizosphere of Tamarix chinensis; Streptomyces; Whole genome sequencing; Secondary metabolites
臨床68.3%抗感染和79.8%抗腫瘤的小分子化學藥來源于天然產物或者其衍生物,而放線菌作為抗生素的寶庫[1-4],從中已分離出鏈霉素、新霉素等結構多樣的抗生素[5]。放線菌抗生素種類多、活性廣、應用十分廣泛,除發揮常規的殺菌、抑菌和抗感染等作用,還可用于抗蟲劑、抗腫瘤劑、除草及免疫抑制。而隨著放線菌物種和代謝產物研究的長期積累,新物種和新活性物質的發現越來越難,科學家們將目光轉向極端環境[6-7]。相較于普通生境,極端環境微生物在長期適應生存中,產生了有別于普通生境微生物的代謝產物,如一些特殊功能的酶類或一些結構特異的代謝產物,以應對環境中生長制約因素對其自身生長的影響,因此更有可能從中發現對人類有用的次級代謝產物。
新疆地處我國西北邊陲,獨特的地理環境造就了干旱、鹽堿化程度高的極端環境。檉柳作為一種廣布的鹽生植物,具有很強的耐鹽、抗旱、耐淹的生物學特性,廣泛分布在新疆內陸鹽堿地,有能量轉換、氣候與水分調節、水質凈化等環境功能[8-9]。本研究開展新疆檉柳根際鹽堿土微生物的分離和多樣性研究[10],根據菌株形態和16S rRNA鑒定,獲得了一株與Streptomyces luozhongensis TRM49605相似性較高的鏈霉菌,命名為Streptomyces sp. TRM76323。兩者的DNA-DNA雜交的dDDH值為30.60%,ANI值為86.09%,為一潛在新物種。本研究以挖掘新疆檉柳根際鏈霉菌的次級代謝產物為目標,結合全基因組測序技術與現代分離純化技術,開展了TRM76323菌種次級代謝產物的預測和挖掘。
1 材料與方法
1.1 儀器、材料及試劑
1.2 菌種來源
Streptomyces sp. TRM76323菌株分離自檉柳根際鹽堿性土壤(東經83°205',北緯38°412'),菌種分別保存于塔里木大學生命科學與技術學院、塔里木大學省部共建國家重點實驗室和中國典型培養物保藏中心(China Center for Type Culture Collection,CCTCC),編號為CCTCC No. M2023092。
1.3 培養基
ISP4培養基:淀粉10.0 g,硫酸鎂2.0 g,碳酸鈣2.0 g,硫酸銨4.0 g,磷酸氫二鉀2.0 g,瓊脂16 g,蒸餾水1.0 L,pH 7.0。
高氏培養基:可溶性淀粉20 g,硝酸鉀1 g,硫酸氫二鉀 0.5 g,硫酸鎂0.5 g,氯化鈉0.5 g,硫酸亞鐵0.01 g,瓊脂20 g,pH 7.0。
1.4 方法
1.4.1 基因組測序
將提取的DNA送到南京派森諾生物信息科技有限公司進行全基因組Illumina Novaseq測序[11]。對獲得的基因組采用AbySS軟件[12]進行拼接。將拼接好的基因組數據上傳到NCBI數據庫(https://pubmed.ncbi.nlm.nih.gov/),基于Snapgene軟件進行分析。
1.4.2 比較基因組分析
基于比較基因組的方法,本研究使用Ezbiocloud (https://www.ezbiocloud.net/)中相似性最高的19個菌株基因組進行下載,將所得基因組進行antiSMASH[13]進行預測,將預測得到的不同基因簇進行匯總分類整理,分析TRM76323菌株的次級代謝潛力,為次級代謝產物的挖掘提供理論指導。
1.4.3 泛基因組分析
對TRM76323菌株及相似菌株進行核心基因組分析,將掃描獲得的數據,同時將數據進行ITOL[14]作圖。利用orthoFinder對所有菌株進行直系同源蛋白分析,將文件使用ITOL進行作圖。基于PEPPAN算法對所有菌株進行泛基因組掃描,將文件利用 ITOL進行作圖。并將通過Peppan掃描得到的peppan.PEPPAN.gene_content文件利用imgeGP在線軟件構建泛基因組曲線圖進行泛基因組分析。
1.4.4 抗性基因分析
使用綜合抗菌研究數據庫(CARD)[15]預測TRM76323的耐藥基因[16],預測其抗性潛力。
1.4.5 代謝產物發酵、分離及鑒定
發酵:選擇ISP4作為種子培養基,配制3 L,接種培養3 d,30 ℃,150 r/min。選擇小米作為發酵培養基,將50 L小米培養基(每升15 g小米,5 g葡萄糖,4 g蛋白胨和3 g氯化鈉,pH7)的置于100 L發酵罐中。同時配3 mol/L NaOH溶液以及HCl溶液調節pH值,將發酵罐進行121 ℃高溫滅菌30 min,滅菌后待溫度冷卻至30 ℃,將種子液接種培養7 d。使用80%甲醇對樣品進行萃取,獲得發酵粗提取物。
分離:將干燥完的樣品進行分段萃取,通過TLC檢測確定洗脫劑,選擇二氯甲烷-甲醇(10:1, V/V)對甲醇萃取后的樣品進行展層,確定濃度后選擇樣品-硅膠(1:8, W/W)進行柱層析分離,柱層析的上樣量為212 g,洗脫劑為二氯甲烷-甲醇(10:1, V/V),洗脫4個柱體積,每200 mL收集為一瓶,將其旋干,點板,確定樣品真空濃縮至干燥,通過TLC薄層色譜分析,確定樣品純度,得到純品化合物。
鑒定:將純品化合物溶解于適宜的氘代溶劑,置于潔凈干燥的核磁管中進行核磁共振分析,得到碳氫譜數據,將碳譜信號輸入上海微譜化合物數據庫(http://www.nmrdata.com/)查詢,將結果與已發表文獻中的碳氫譜數據進行對比,確定化合物結構。
2" " 結果
2.1 菌株基因組特征
TRM76323菌株的基因組序列由7,338,911 bp組成,包含127個重疊群、5個rRNA、103個tRNA和6330個編碼序列,G+C含量為72.1%。全基因組登入號:JAWCTQ000000000,數據庫為NCBI數據庫(https://www.ncbi.nlm.nih.gov/)。
2.2 生物合成相關基因簇分析及比較基因組學分析
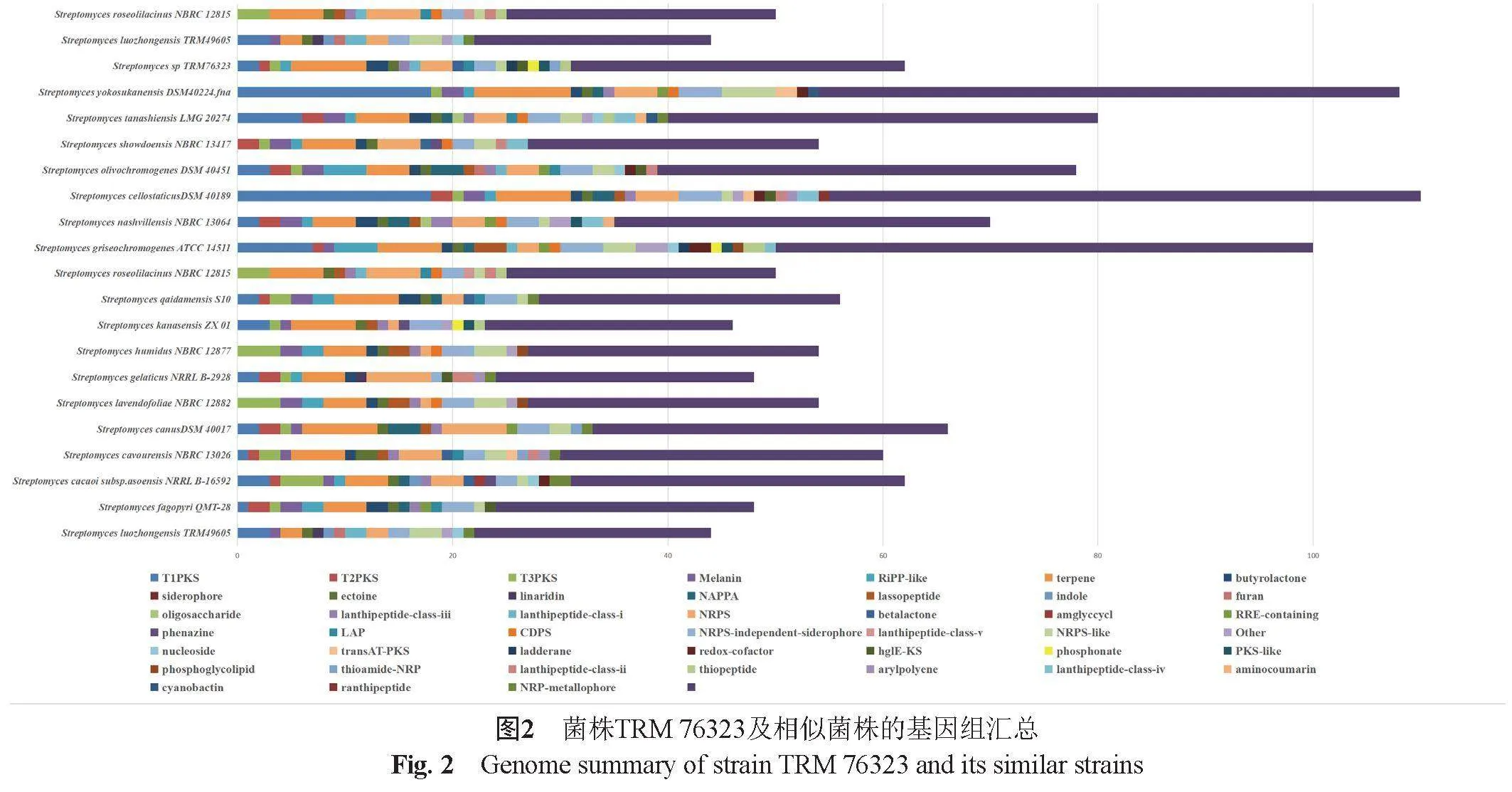
本研究通過對菌株TRM 76323及相似菌株進行antiSMASH分析,將結果匯總進行比較基因組學分析(圖1)。基于比較基因組學分析可知所有菌株都有Terpene和NRPS-independent-siderophore(非核糖體肽合成酶-獨立鐵載體合成酶)基因簇和促植物生長因子。所有菌株的主要次級代謝產物還有T1PKS、T2PKS、T3PKS、melanin、RiPP-like和butyrolactone等。相似菌株S. cellostaticus DSM 40189有55個基因簇,代謝潛力最豐富,S. yokosukanensis DSM 40224次之,有54個基因簇,S. luozhongensis TRM 49605只有22個基因簇。其中該菌株TRM 76323中Cluster 8為膦酸生物合成基因簇,基因簇全長96,815 kb,該基因簇在其相似菌株中較為獨特,其可能產生相應的抗生素,草甘膦是該類化合物的主要代表之一。
本研究利用antiSMASH對菌株Streptomyces sp. TRM76323基因組的次級代謝產物合成基因簇進行預測及分析,檢測到31個與次級代謝產物合成相關的基因簇,主要包含6個terpene、6個NRPS、5個lanthipeptide、2個siderophore、3個butyrolactone和1個phosphonate。從次級代謝產物類型看,其代謝能力較為豐富,其中可能包含抗真菌的大環類IPC合酶抑制劑rustmicin、抗腫瘤neocarzinostatin、胰腺癌干細胞抑制劑BE-43547A1、抗結核ecumicin、酶抑制劑RP-1776、抗病毒soraphen A、抗細菌ficellomycin和抗腫瘤zorbamycin等。該菌株具有比較獨特的膦酸生物合成基因簇,與已發表的代謝產物的基因簇相似性較低,表明其很大的合成新型抗生素潛力。
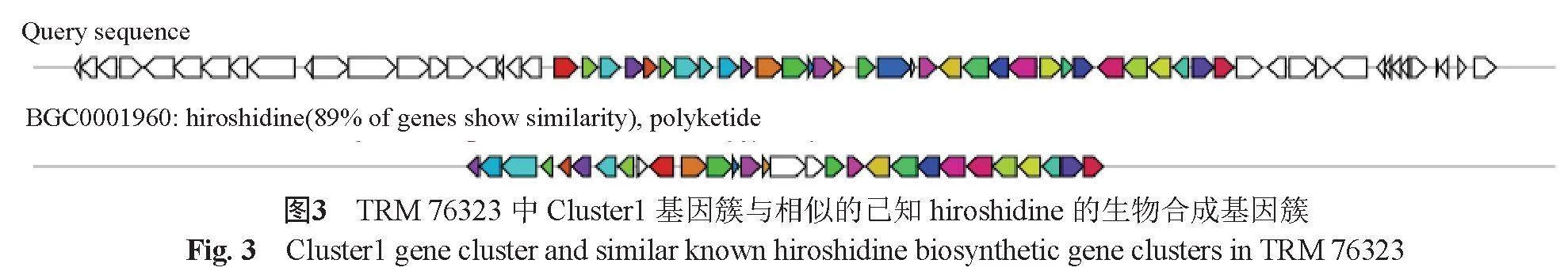
TRM 76323 中Cluster 1共有基因65個基因構成,與已知S. hiroshimensis中的hiroshidine的生物合成基因簇(MIBiGaccession: BGC0001960)具有同源性,相似度為89%,說明菌株TRM 76323的Region1基因簇極有可能具有合成這類化合物的潛力,且可能為新抗生素。
2.3 菌株的泛基因組分析
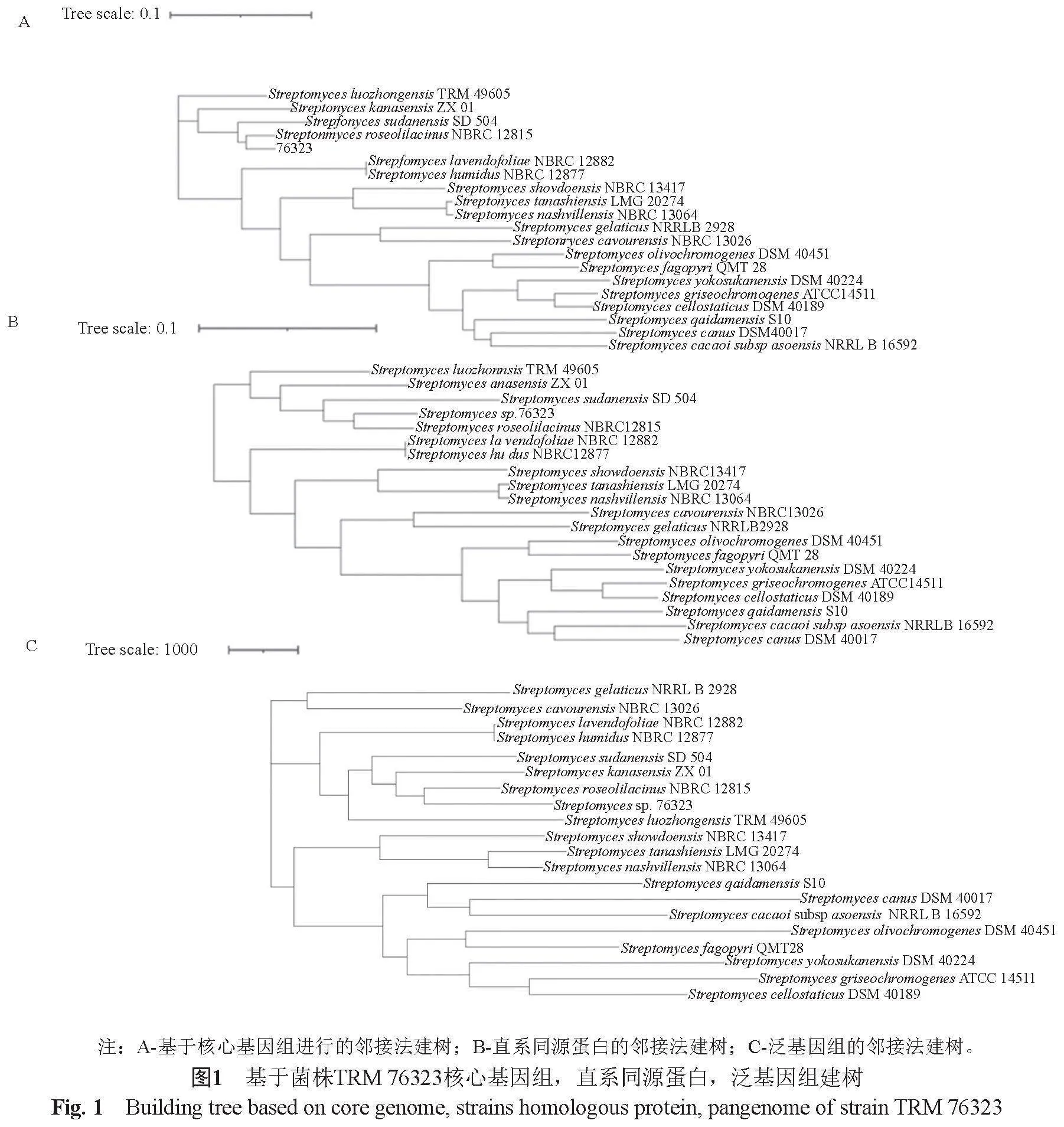
菌株TRM 76323與其相似菌株建立系統發育樹,基于核心基因組,同源蛋白,泛基因組建樹(圖1)可知,TRM 76323與其最相似的兩株菌株都聚類到一起,說明了菌株的同源性。菌株TRM 49605和菌株S. roseoliacinus NBRC 12851都具有明顯分支,說明菌株的物種特異性。通過同源蛋白建樹的同源性揭示這些蛋白質控制的基因表達之間的相互關系,也可以預測在特定基因組織中蛋白質的表達和調控水平,說明菌株TRM 76323與相似菌株具有較好的同源性。泛基因組代表該物種所有的基因庫,菌株TRM 76323與相似菌株聚類到一起,其生物合成代謝具有相似性,通過圖2發現大多數菌株具有共同的基因簇,它們分別是terpene、ectoine、NRPS、NRPS-like、NRPS-independent-siderophore和lanthipeptide-class-iii等,泛基因組的分析結果與比較基因組一致。
TRM 76323中Cluster 1共有基因65個基因構成,與已知Streptomyces hiroshimensis中的hiroshidine生物合成基因簇(MIBiGaccession:BGC0001960)具有同源性,相似度為89%,說明菌株TRM 76323的Cluster 1基因簇極有可能具有合成這類化合物的潛力(圖3)。
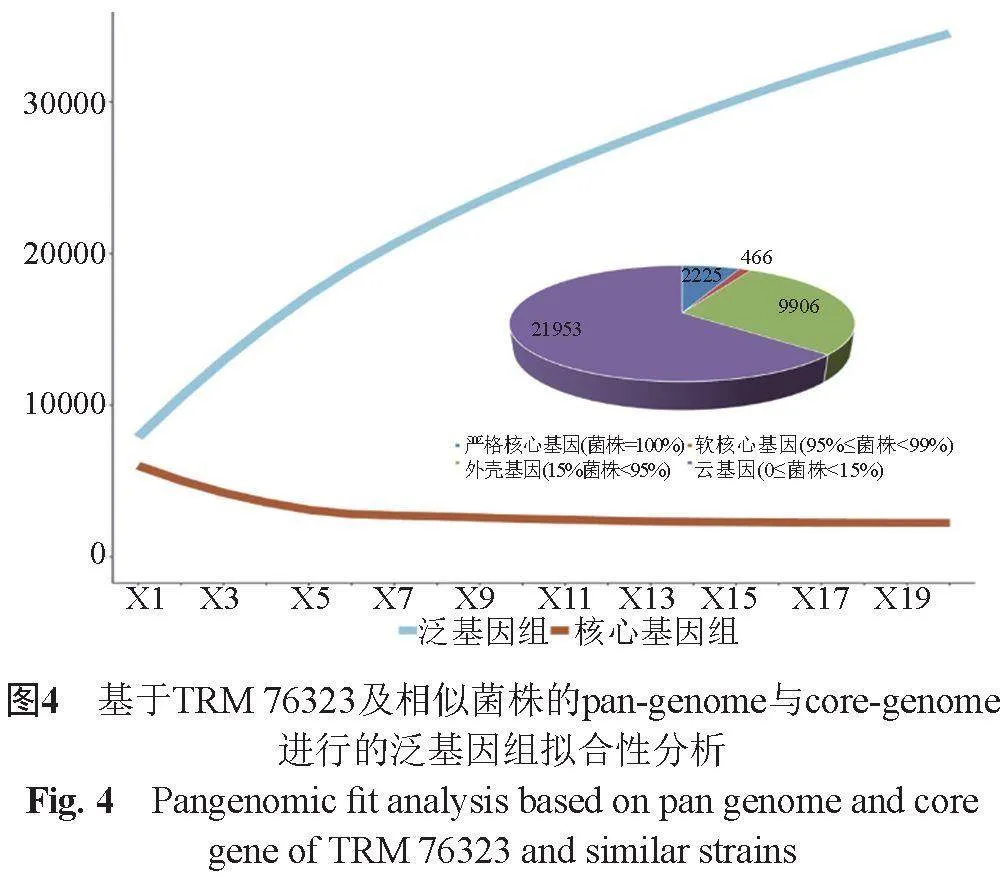
泛基因組關聯研究(PanGWAS)是通過使用Peppan算法來定義鏈霉菌中的生物基因組。只有敏感性和特異性≥95%的基因,存在于≤25%的對照組中,才被認為是有意義的結果。通過泛基因組基因和核心基因擬合性分析,顯示了添加最后一個基因組后的明顯增加趨勢,支持鏈霉菌的泛基因組是開放的(α=0.534±0.015,γ=0.049±0.004)。核心基因組大小估計表明,核心基因的數量接近恒定值。泛基因組基因家族的數量隨著鏈霉菌基因組數量的增加而增加,最終有34,550個基因。核心基因組中的基因家族數量隨著鏈霉菌基因組數量的增加而減少。共有2,225個核心基因,占6.4%,軟核心基因共有446個,占比1.3%,共有9,906個外殼基因,占28.7%,共有21953個云基因,占63.6%(圖4)。泛基因組結果模型是開放的,更多的非核心基因促進了物種的多樣性,使它們能夠適應各種環境條件。
2.4 菌株抗性基因預測
基于耐藥基因數據庫CARD對菌株TRM 76323進行抗性分析,分析可知菌株抗性包含利福平單加氧酶、糖肽類耐藥基因簇和解旋酶樣RNA聚合酶保護蛋白基因家族,序列相似性分別為75.52%、35.38%和81.24%,說明菌株可能具有此類抗性,已另文發表[16]。
2.5 菌株代謝產物鑒定
將獲得的單體化合物利用核磁共振分析。首先為樣品選擇適宜的氘代試劑,旋蒸干后使用相應的試劑進行核磁共振分析,并結合相關文獻確定化合物的單體結構,解析如下。
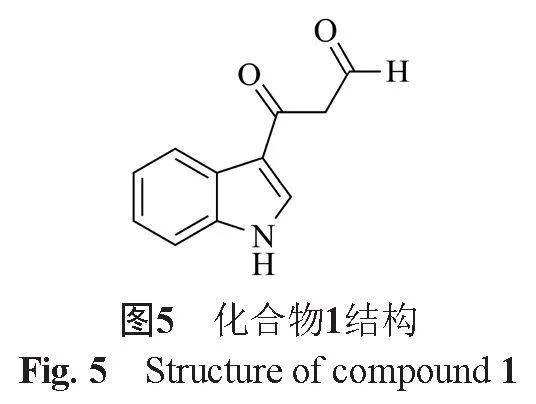
化合物1:淡黃色無定形粉末,5 mg,溶于DMSO,化合物結構鑒定為β-羰基-1H-吲哚-3-丙醛。1H NMR(500 MHz, DMSO-d6): 8.34(1H, s, H-10), 8.18(1H, d, J=7.5 Hz, H-4), 7.22~7.25(2H, m, H-5, H-6), 4.54(1H, d, J=5.0 Hz, C-9), 13C NMR(125 MHz, DMSO-d6): 134.1(d, C-2), 114.4(s, C-3), 125.8(s, C-3a), 121.6(d, C-4), 122.3(d, C-5), 123.4(d, C-6), 112.6(d, C-7), 136.9(s, C-7a), 190.0(s, C-8), 44.8(t, C-9), 161.8(s, C-10)。經文獻查閱,該化合物與文獻報道[17]基本對應,鑒定該化合物為β-羰基-1H-吲哚-3-丙醛(圖5)。
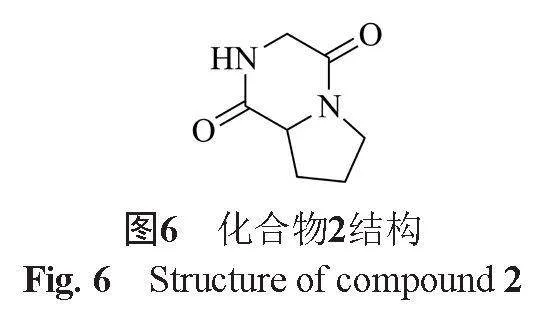
化合物2:白色粉末,5 mg,溶于DMSO,化合物結構鑒定為甘氨酸-L-脯氨酸。1H NMR(500 MHz, DMSO-d6): 4.01(1H, dd, J =16.8, 1.6 Hz, H-3a), 3.98(1H, d, J =16.8 Hz, H-3b), 4.12(1H, ddd, J=7.2, 6.6, 1.8 Hz, H-6), 2.55/2.13(each1H, m, H-7), 1.86(2H, m, H-8), 3.54(2H, m, H-9),13C NMR(125 MHz, DMSO-d6): 163.9(C-2), 58.1(C-6), 169.3(C-5), 58.1(C-6), 45.9(C-3), 22.1(C-8), 27.9(C-7).經過微譜數據庫比對,文獻查閱,該化合物與文獻報道基本對應[18],鑒定該化合物為環二肽(甘氨酸-L-脯氨酸)即cyclo-(Gly-L-Pro)(圖6)。
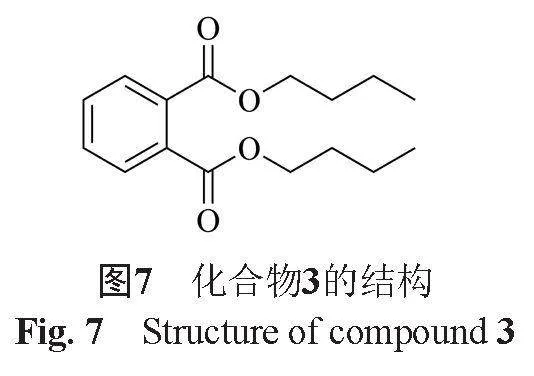
化合物3:無色透明液體,4 mg,溶于MeOD。化合物結構鑒定為鄰苯二甲酸二丁酯。1H NMR(500 MHz, DMSO-d6): 1H NMR(500 MHz, Methanol-d4): 0.99(6H, t, J =5, CH3 5', 5\"), 1.47(4H, m, CH2-4', 4\"), 1.77(4H, m, CH2-3' ,3\"), 4.32(4H, t, J =5, CH2-2', 2\"), 7.75(2H, dd, J =10, 5, H-2, 5); 7.65(2H, dd, J=10, 5, H-3, 4).13C NMR(125 MHz, Methanol-d4):12.2(q, C-5', 5\"), 18.4(t, C-4', 4\"), 39.9(t, C-3', 3\"), 64.9(t, C-2', 2''), 128.1(d, C-2, 5), 130.5(d, C-3, 4), 131.8(s, C-1, 6), 167.5(s, C-1', 1\"),與文獻相符合[19],見圖7。
3 結論與討論
眾多關于植物根際鏈霉菌分離次級代謝產物的報道說明了植物根際來源的微生物具有豐富的次級代謝產物,如張小霞等[20]從檉柳根際土壤獲得一株鹽單胞菌Bachu 26,具有植物促生的作用。周冬月等[21]對長白山特殊生境藥用植物根際拮抗菌的次級代謝產物進行分離,獲取拮抗金黃色葡萄球菌的化合物,張一等[22]對紅樹林植物木欖根際土壤真菌Penicillium sp. MA-37的化學成分進行研究鑒定了8個天然產物,包括氮-(2-甲氧基-4-羥基苯)-丙酰胺酸(1)等。對于新疆檉柳根際鏈霉菌的次級代謝產物的挖掘的研究報道不多,因此對于檉柳根際來源的微生物代謝產物挖掘具有重要意義。本研究采用第二代測序技術,獲得了新疆檉柳根際鏈霉菌TRM 76323的全基因組序列,并利用antiSMASH(v6.0.1)在線工具預測了該菌株的潛在生物合成基因簇,發現其具有31條生物合成基因簇,相比較其相似菌株,說明其具有較為豐富的代謝潛能。通過對菌株TRM 76323比較基因組學和泛基因組學分析,發現了菌株共同代謝能力和獨特的生物合成基因,如基因組揭示了其含有膦酸合成基因簇的相似菌株,值得進一步挖掘。抗性基因是導致抗生素抗性菌株出現的原因之一,它是由基因突變導致的。因此,通過基因檢測來篩選抗性基因,可以幫助人們更好地了解菌株的抗性,以便更好地利用微生物資源,為此通過預測菌株TRM 76323抗性基因分析可發現其可能具有利福平單加氧酶、糖肽類耐藥的耐藥性,該實驗結果為后續天然產物挖掘提供理論支持,而獲得的β-羰基-1H-吲哚-3-丙醛,環二肽,鄰苯二甲酸二丁酯等化合物豐富了小分子化合物資源庫,對于以上化合物李云鵬等[23]報道了鄰苯二甲酸二丁酯對于4種鐮刀菌[尖孢鐮刀菌(Fusarium oxysporum)、茄病鐮刀菌(F. solani)、三線鐮刀菌(F. tricinctum)、燕麥鐮刀菌(F. avenaceum)]具有較好的拮抗活性;劉睿等[24]發現環二肽類化合物對慢性髓原白血病細胞K562具有細胞毒活性。
本研究對菌株TRM 76323的基因組分析幫助我們準確、快速地識別菌株中的基因和功能,發現菌株獨特的代謝潛力,以及代謝潛力的同一性。針對世界抗生素瀕臨枯竭,大多數菌株具有耐藥性的問題,大多研究根據生物合成基因簇和抗生素抗性基因這兩方面,試著對抗生素展開篩選,用來篩選的這兩類基因中,抗性基因可以通過靶向修飾起作用,易于識別。相關研究通過對基因信息的分析,確定了新抗生素的存在,其中之一是一個已知的化合物complestatin,而另一種則來自采于加拿大亞伯達省的一株鏈霉菌,被命名為corbomycin[25],本研究嘗試通過菌株抗性基因的預測表明了菌株抗性基因包括了糖肽類抗生素抗性基因等,為后續天然產物的挖掘提供理論指導。本研究利用現代分離技術獲得了相關的活性化合物,但未充分獲得該菌株的代謝產物,根據基因組分析發現菌株中存在和已知化合物生物合成基因簇相似度低的基因簇,說明菌株可能具有產生新化合物的潛力,該菌株還值得后續進行挖掘。
參 考 文 獻
陳傳兵, 胡金姍, 于鑫, 等. 深海放線菌Actinomadura cremea中的生物堿類化合物[J]. 中山大學學報(自然科學版)(中英文), 2022, 61(3): 28-34.
萬中義, 方偉, 張亞妮, 等. 放線菌HBERC-39158抗真菌活性化合物的分離鑒定[J]. 湖北農業科學, 2020, 59(S1): 140-144.
劉衛兵, 葉邦策. 放線菌聚酮類化合物的合成生物學研究及生物制造[J]. 化工進展, 2021, 40(3): 1226-1237.
江婷, 蒲洪, 段燕文, 等. 沙漠、火山、極地來源鏈霉菌新天然產物(2009-2020)[J]. 有機化學, 2021, 41(5): 1804-1820.
羅健雅, 黃紫貝, 趙以恒, 等. 抗廣譜耐藥細菌的放線菌篩選及抗菌活性研究[J]. 中國抗生素雜志, 2023, 48(11): 1229-1241.
莊瀅潭, 劉芮存, 陳雨露, 等. 極端微生物及其應用研究進展[J]. 中國科學: 生命科學, 2022, 52(2): 204-222.
顧覺奮, 羅學剛. 極端微生物活性物質的研究進展[J]. 中國天然藥物, 2003, 1(4): 60-64.
寧虎森, 何苗, 羅青紅, 等. 新疆檉柳林生態服務功能及其價值評估分析[J]. 生態科學, 2019, 38(4): 111-118.
陳曉琴, 王婷, 汪建紅. 新疆檉柳屬植物的價值及開發利用建議[J]. 新疆師范大學學報(自然科學版), 2006, 25(3): 100-102.
陳乙煌, 邢利, 東珍珍, 等. 檉柳根際土壤放線菌的分離及拮抗活性篩選[J]. 新疆農業科學, 2023, 60(7): 1790-1797.
任海偉, 李志娟, 劉美琪, 等. 基于高通量測序技術分析不同窖齡窖泥真菌群落多樣性與空間異質性[J]. 食品科學, 2024, 45(2): 178-187.
Jackman S D, Vandervalk B P, Mohamadi H, et al. ABySS 2.0: Resource-efficient assembly of large genomes using a Bloom filter[J]. Genome Res, 2017, 27: 768-777.
Liu Y Y, Xu M J, Tang Y Q, et al. Genome features and AntiSMASH analysis of an endophytic strain Fusarium sp. R1[J]. Metabolites, 2022, 12(6): 521.
Letunic I, Bork P. Interactive Tree Of Life (iTOL): An online tool for phylogenetic tree display and annotation[J]. Bioinformatics, 2007, 23(1): 127-128.
Alcock B P, Raphenya A R, Lau T T Y, et al. CARD 2020: Antibiotic resistome surveillance with the comprehensive antibiotic resistance database[J]. Nucleic Acids Res, 2020, 48(D1): D517-D525.
Chen Y H, Wu Y Z, Liu Q, et al. Streptomyces tamarix sp. nov.: Antagonism against Alternaria gaisen producing streptochlorin, isolated from Tamarix root soil[J/OL]. Front Microbiol, 2023, 14: 1273842. doi: 10.3389/fmicb.2023.
1273842. eCollection 2023.
孫勇, 萬傳星, 張利莉. 胡楊林土壤放線菌TRM 43006分類鑒定及其次級代謝產物研究[J]. 塔里木大學學報, 2015, 27(3): 1-7.
Ferro J N, de Aquino F L, de Brito R G, et al. Cyclo-Gly-Pro, a cyclic dipeptide, attenuates nociceptive behaviour and inflammatory response in mice[J]. Clin Exp Pharmacol Physiol, 2015, 42(12): 1287-1295.
Williams T D, Blanchfield J B. Retention, distribution, excretion, and metabolism of dibutyl phthalate-7-14C in the rat[J]. J Agric Food Chem, 2002, 23(5): 854-858.
張小霞, 陳筱玥, 王秋云, 等. 檉柳根際一株鹽單胞菌Bachu 26的分離、鑒定及其鹽脅迫下的促生作用研究[J]. 微生物學報, 2024, 64(2): 607-622.
周東月, 尹遜哲, 姜爽, 等. 長白山特殊生境藥用植物根際拮抗菌的分離及其代謝產物活性分析[J]. 時珍國醫國藥, 2018, 29(9): 2090-2092.
張一, 李曉明, 尚卓, 等. 紅樹林植物木欖根際土壤真菌Penicillium sp. MA-37的化學成分研究[J]. 海洋科學, 2013, 37(2): 7-11.
李元鵬, 牟曉玲, 馬家易, 等. 鄰苯二甲酸二丁酯對鐮刀菌化感抑制機制研究[J]. 甘肅農業大學學報, 2023, 58(5): 113-121.
劉睿, 朱天驕, 朱偉明, 等. 海洋放線菌S1001中抗腫瘤活性成分的研究[J]. 中國抗生素雜志, 2006, 31(1): 36-38.
Culp E J, Waglechner N, Wang W, et al. Evolution-guided discovery of antibiotics that inhibit peptidoglycan remodelling[J]. Nature. 2020, 578(7796): 582-587.

