沙城產區釀酒酵母多樣性研究
趙靜靜,李 艷,2,*
(1.河北科技大學生物科學與工程學院,河北 石家莊 050018;2.河北省發酵工程技術研究中心,河北 石家莊 050018)
沙城產區釀酒酵母多樣性研究
趙靜靜1,李 艷1,2,*
(1.河北科技大學生物科學與工程學院,河北 石家莊 050018;2.河北省發酵工程技術研究中心,河北 石家莊 050018)
對沙城產區龍眼葡萄相關環境中分離篩選的釀酒酵母進行多樣性研究。在連續3年(2008、2009、2010年)的實驗中,共從葡萄園土壤、葡萄酒廠設備表面、接觸過葡萄酒廠設備的葡萄汁和自然發酵過程中采集菌源樣品227份,共分離得到1358株酵母菌。用5.8S-ITS區域RFLP方法進行分子水平的分類鑒定及賴氨酸培養基復篩,得到了270株釀酒酵母。再利用Interdelta PCR指紋圖譜法將釀酒酵母區分為16類,其中土壤5類,自然發酵過程中第2、3、4期分別得到4類、10類和11類;酒廠設備表面3類;接觸酒廠設備的葡萄汁3類。釀酒酵母的種類因樣品采集時間、采集地點等不同有明顯區別。自然發酵過程中得到的釀酒酵母被認為是本土酵母的可能性最大。
釀酒酵母;5.8S-ITS區域RFLP分析;Interdelta PCR指紋圖譜法;生物多樣性;本土酵母
葡萄酒發酵過程是復雜的微生物代謝過程,涉及細菌、霉菌、酵母菌等多種微生物,其中酵母菌起著關鍵作用。在發酵初期,非酵母屬酵母占主導優勢[1],隨著發酵進行,發酵液中乙醇含量逐漸增加,對乙醇耐受力不高的非酵母屬酵母逐漸被淘汰,取而代之的是酵母屬酵母,最終完成整個發酵過程[2-3]。前人[4-5]的研究發現,釀酒酵母對于葡萄酒中最終揮發性化合物的形成及感官品質有著非常重要的影響。目前國內大多數葡萄酒生產企業都是采用商品活性干酵母進行生產,其優點是性能穩定的酵母菌能夠確保質量統一的產品,卻很難得到獨特風味的葡萄酒。因此,在產區環境范圍內,分離和篩選適合特定葡萄產區和葡萄品種的釀酒酵母菌株具有十分重要的意義。
目前,用于酵母菌屬和種水平區分鑒定的分子方法有許多種,限制性片段長度多態性(RFLP)分析由于其鑒定結果的準確性和操作簡便被廣泛的予以應用[6-7]。對于釀酒酵母種以下水平區分的方法包括Interdelta 序列指紋圖譜分析和線粒體DNA限制性分析等[8-10]。本研究采用5.8S-ITS區域RFLP分析法和Interdelta 序列指紋圖譜分析對對龍眼葡萄酒環境中連續3年分離得到的酵母菌進行屬、種和種以下水平的區分,研究沙城產區釀酒酵母的多樣性,為進一步篩選獲得特色的土著酵母提供參考。
1 材料與方法
1.1 樣品的采集及處理
1.1.1 樣品采集
本研究于2008—2010年3個釀酒季節(10月)和2009年非釀酒季節(4月),從河北沙城釀酒葡萄產區的懷來縣沙城鎮選擇了沙營、夾河和東水泉3個自然村的龍眼葡萄園,以及中國長城葡萄酒有限公司的兩個釀酒車間,采集葡萄園土壤、葡萄果實、葡萄鮮汁、自然發酵過程的發酵液和擦拭釀酒設備內壁等樣品共計227份,用于分離純化酵母菌株。
1.1.2 樣品處理
所有樣品均無菌采集、24h內運輸回實驗室。
土壤:稱取1g土壤,加無菌水進行梯度稀釋,用于純種分離。
葡萄漿果:取新鮮完整的龍眼葡萄漿果(約20g),置于無菌生理鹽水溶液中,輕柔振蕩10min,再進行梯度稀釋。
葡萄鮮汁:實驗室自制:在無菌狀態下,將葡萄壓榨出汁后,進行梯度稀釋;接觸酒廠設備的葡萄汁:從中國長城葡萄酒有限公司釀酒車間取汁,在實驗室無菌狀態下進行梯度稀釋。
自然發酵:取龍眼葡萄,在實驗室無菌條件下破碎壓榨進行自然發酵,按發酵液中殘糖量的不同將發酵分為4個時期:第1個時期為加SO2及果膠酶浸漬24h后取樣,第2個時期為發酵液中殘糖80%(質量分數,下同),第3個時期為發酵液中殘糖30%,第4個時期為發酵液中殘糖4g/L以下。每個發酵時期采集發酵液樣品1mL,進行梯度稀釋。
設備表面:用無菌棉球擦拭設備內壁表面約40cm2,將其中的菌液擠出,定容到20mL后取1mL進行梯度稀釋。
經梯度稀釋后的樣品0.2mL涂布于WL培養基上,25℃條件下培養7d,挑菌經劃線培養得到單菌落。
1.2 釀酒酵母菌株的篩選
1.2.1 WL培養基進行菌落聚類及巨大菌落觀察
將菌株劃線接種于WL培養基上,25℃條件下培養7 d,觀察菌落形態,挑選釀酒酵母菌株。
巨大菌落形態觀察:將供試細胞懸液用0.8%的無菌生理鹽水稀釋至以0.1mL 細胞懸液含10個細胞為佳,涂布在10%的麥汁明膠培養基平板上,在20℃左右培養3~4周。待巨大菌落形成后,觀察菌落的大小、顏色、形態、光澤和周緣形狀等。
1.2.2 酵母菌5.8S rRNA-ITS區域及核糖體26S D1/D2區域RFLP分析
1)DNA提取:SDS裂解法[11]。
2)5.8S rRNA-ITS區域PCR反應體系[6](50μL):引物 ITS1 (5′-TCCGTAGGTGAACCTGCGG-3′)、ITS4 (5′-TCCTCCGCTTATTGATATGC-3′)各 0.5μmol/L;dNTP 0.2mmol/L;Colorless GoTaq Reaction Buffer 10μL;TaqDNA 聚合酶2.5μL;模板DNA 1μL。
PCR反應條件:95℃預變性7min;95℃變性1min,52℃退火2min,72℃延伸2min,循環35次;再72℃延伸10min。PCR產物用2%瓊脂糖凝膠70V電泳檢測。
3)核糖體26S D1/D2區域PCR反應體系(50μL):引物NL1(5′-GCA TAT CAA TAA GCG GAG GAAAAG-3′),NL2(5′-GGT CCG TGT TTC AAG ACG G-3′)各0.2 μ mol/L;dNTP 0.2mmol/L,Colorless GoTaq Reaction Buffer 10μL,TaqDNA聚合酶2.5U,模版DNA 1μL。
PCR反應條件:95℃預變性7min;94℃變性1min,52℃退火1min,72℃延伸80s,循環36次;再72℃延伸8min。PCR產物用2%瓊脂糖凝膠70V電泳檢測。
4)利用3種限制性內切酶HaeIII、Hinf I和cofI對擴增產物進行酶切[12]。酶切反應體系20μL,10×Buffer 2 μL、BSA 0.2μL、PCR產物8μL、限制性內切酶1μL。37℃溫育2h;3%瓊脂糖凝膠電泳檢測,控制電壓70V。
1.2.3 賴氨酸培養基復篩釀酒酵母
賴氨酸培養基(g/L):D-葡萄糖 10、L-組氨酸0.001、DL-蛋氨酸0.002、DL-色氨酸0.002、對-氨基苯甲酸2×104、生物素2×105、葉酸2×106、肌醇0.01、煙酸4×104、泛酸0.002、鹽酸吡哆醇4×104、核黃素2×104、鹽酸硫胺素4×104、硼酸5×104、結晶氯化銅4×105、碘化鉀1×104、結晶氯化鐵2×104、結晶硫酸錳4×104、結晶鉬酸鈉2×104、結晶硫酸鋅4×104、磷酸二氫鉀0.85、磷酸氫二鉀0.15、結品硫酸鎂0.5、氯化鈉0.1、結晶氯化鈣0.1、賴氨酸鹽酸鹽2.5、瓊脂20,pH值自然,121℃滅菌20min[13]。
釀酒酵母菌株的復篩:將經過初篩的菌株,接種到YEPD液體培養基中活化24h后,按照1%接種量接種到5mL無菌水中進行菌株饑餓處理,7d后接種到賴氨酸培養基中,27℃條件下培養15d后觀察酵母生長情況。
1.3 Interdelta PCR指紋圖譜法區分鑒定釀酒酵母菌株
DNA提取:SDS裂解法[11]。
反應體系(50μL):引物δ12、δ2各1μmol/L;dNTP 0.2mmol/L;Colorless GoTaq Reaction Buffer 10μL;TaqDNA聚合酶0.5μL;模板DNA 1μL。
反應條件:97℃預變性4min;94℃變性30s,45℃退火1min,72℃延伸2min,循環30次;再72℃延伸10min。PCR 產物用2%瓊脂糖凝膠70V電泳檢測。
2 結果與分析
2.1 酵母菌的分離
從沙城產區龍眼葡萄相關環境(包括葡萄園土壤、葡萄酒廠設備表面、接觸過葡萄酒廠設備的葡萄汁和自然發酵過程)采集227份樣品,共篩選得到1358株酵母菌。通過WL培養基進行酵母菌的形態聚類和區分得到42種不同的形態類型[14]。
2.2 釀酒酵母的篩選
2.2.1 菌落形態聚類結果
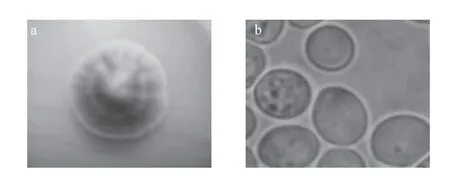
圖1 釀酒酵母的菌落形態(a)和顯微形態(b)Fig.1 Colonial and microscopic morphology of Saccharomyces cerevisiae
由圖1可知,經過WL培養基的培養及巨大菌落的觀察,單菌落形態為邊緣微綠色,中間顏色略深,火山狀凸起,表面光滑有褶皺,邊緣較整齊;顯微鏡觀察其細胞的顯微形態為卵圓形,有的菌株正在進行出芽生殖,此類酵母菌被初步認定為釀酒酵母(Saccharomyces cerevisiae)[15]。
2.2.2 酵母菌的5.8S rRNA-ITS區域及核糖體26S D1/D2區域RFLP分析
在酵母菌形態初步鑒定的基礎上,利用5.8S rRNAITS區域及核糖體26S D1/D2區域RFLP分析的方法對此類酵母進行分子鑒定,分析結果見表1。結果證明這種形態類型的酵母菌的確是釀酒酵母(Saccharomyces cerevisiae)[16],初篩得到300株,其中,2008年釀酒季節54株,2009年170株,2010年76株。而2009年非釀酒季節未篩得該酵母菌株,原因是非釀酒季節只是對土壤進行了采樣分離,而土壤中本來所含酵母菌數量就不多,再經過實驗過程中的定量梯度稀釋,使得篩得酵母菌株的幾率縮小,因而從非釀酒季節的土壤中不易獲得釀酒酵母菌株[17]。另外,沙城產區春季風沙較大,天氣干燥也是未分離到釀酒酵母的原因之一。

表1 5.8S rRNA-ITS區域及核糖體26S D1/D2區域RFLP分析結果Table 1 RFLP analysis result 5.8S rRNA-ITS and 26S D1/D2 of isolated yeasts
隨機挑取兩株進行5.8S rRNA-ITS區域PCR產物進行核苷酸序列測定,通過與NCBI數據庫進行比對來最終確定酵母菌的種屬。通過與Genebank數據庫菌株比對,菌株鑒定為釀酒酵母,結果見表2。

表2 酵母菌5.8S-ITS區域的基因序列分析結果Table 2 Sequence analysis of the 5.8S-ITS of yeast strains
2.2.3 賴氨酸培養基復篩釀酒酵母
釀酒酵母不能采用賴氨酸作為酵母氮源,因而在賴氨酸培養基上不能生長,故可以通過賴氨酸培養基對初步鑒定為釀酒酵母的菌株進行復篩[13]。結果顯示,其中30株菌在培養了3~4周后能夠在賴氨酸培養基上生長,證明其并不是釀酒酵母,因此釀酒酵母菌株共篩選得到了270株。
2.3 Interdelta PCR指紋圖譜法區分鑒定釀酒酵母

圖2 釀酒酵母PCR產物電泳圖Fig.2 Interdelta fingerprinting patterns of Saccharomyce cerevisiae

表3 Interdelta PCR指紋圖譜分析結果Table 3 Results of Interdelta fingerprinting analysis of Saccharomyces cerevisiae
對所得270株釀酒酵母進行Interdelta PCR指紋圖譜法的區分鑒定,分為16類,分析結果如表3所示,電泳圖譜如圖2所示。
2008年篩選得到的36株釀酒酵母經過PCR指紋圖譜分析區分為5類,2009年158株區分為9類,2010年76株區分為4類。其中僅有2類在兩年出現:類型1在2008年和2010年都分離得到;類型5同時出現在2008年和2009年。從菌源分析,土壤中分離得到5類釀酒酵母共25株,占總菌數的9.3%;自然發酵第2期得到4類共26株,占總菌數的9.6%;自然發酵第3期得到10類共97株,占總菌數的35.9%;自然發酵第4期得到11類共80株,占總菌數的29.6%;酒廠設備分離得到3類共26株,占總菌數的9.6%;接觸酒廠設備鮮汁得到3類16株,占總菌數的5.9%。自然發酵過程的第3、4期釀酒酵母的多樣性是最顯著的。由于風、雨等氣候因素的影響,使葡萄漿果表面酵母菌的數量很少,按照本實驗操作在葡萄漿果表面未篩得釀酒酵母。但是葡萄自然發酵過程所出現的酵母菌株應該是來自葡萄果表面,因為在發酵過程中少量菌株自身生長使菌群壯大,再加上這些酵母的發酵能力和酒精耐受力強,在自然發酵中后期成為主導菌群,故篩選到大量菌株,在這些酵母中最有可能篩得本土釀酒酵母。由于企業生產采用商品酵母,發酵完成后設備清洗和滅菌的不徹底,導致從酒廠設備表面及鮮汁中篩選得到的酵母菌多為商用酵母的可能性較大,不易獲得本土菌株。
對區分得到的16類釀酒酵母進行統計分析,可見各類釀酒酵母所占比例和彼此的關系,分別見圖3、4。

圖3 各類型釀酒酵母所占比例Fig.3 Composition of various populations of Saccharomyce cerevisiae
由圖3可知,類型5和9數量最多,占釀酒酵母總數的20%左右,其他類型數量均低于12%。這兩個類型的菌株在2008年和2009年的3個葡萄園土壤和自然發酵過程中篩得。

圖4 釀酒酵母聚類分析樹形圖Fig.4 Phylogenetic tree of Saccharomyce cerevisiae
由圖4可知,各類型菌株的親緣關系。其中,類型3和16親緣關系最近,而類型10與其他類型菌的關系最遠。葡萄酒相關環境受年份、氣候等條件的影響,會導致微生物組成的變化[18],因此釀酒酵母經過區分鑒定后的類型也相對較多。
2.4 本土釀酒酵母的分布
在葡萄酒生產中應用本土釀酒酵母,容易獲得具有地區特色的葡萄酒產品。目前國內葡萄酒生產企業都是應用商用活性干酵母進行生產,難以獲得具有獨特風味的葡萄酒,因此篩選本土優良酵母菌具有十分重要的應用價值。與我國常用的5種商用活性干酵母的PCR指紋圖譜鑒定結果進行比對,其中包括LAFFORT F5、Zymaflore F15、安琪葡萄酒酵母、Lalvin CY-3079、Vitilevure KD[19]。
電泳條帶大小比對的結果顯示,在沙城產區分離到的釀酒酵母菌中存在著商用酵母。其中,篩選得到的釀酒酵母類型1與Lalvin CY-3079一致,這類酵母來自2008年夾河土壤、接觸過設備的葡萄汁還有2010年夾河自然發酵4期。其他15類與商用酵母不同,有可能是本土酵母。特別是在自然發酵第3、4期篩到的14類均為本土酵母。
總的來說,獲得本土釀酒酵母的途徑主要是土壤、葡萄果表面、自然發酵過程。本研究土壤中分離到4種類型的本土酵母。自然發酵過程中共分離到14種類型的釀酒酵母,其中13種類型為本土酵母。
3 結 論
本實驗研究了沙城產區龍眼葡萄酒相關環境中分離得到的釀酒酵母種類及多樣性。利用Interdelta PCR指紋圖譜法將分離得到的270株釀酒酵母區分為16類。其中土壤中分離得到5類;自然發酵2、3、4期分別為4類、10類和11類;酒廠設備3類;接觸酒廠設備鮮汁3類。其中有一類釀酒酵母屬于商用酵母CY-3079,其余15類均為沙城產區的本土釀酒酵母菌株。自然發酵過程中釀酒酵母的多樣性復雜,是分離本土優良酵母的重要來源。本研究為針對特定葡萄產區和葡萄品種進行優良本土酵母的篩選奠定了基礎。后續對篩得菌株的生理生化特性及釀酒性能的研究還在進行中。
[1] ZOTT K, MIOT S C, CLAISSE O, et al. Dynamics and diversity of non-Saccharomycesyeasts during the early stages in winemaking[J].International Journal of Food Microbiology, 2008, 125(2): 197-203.
[2] PINA C, SANTOS C, COUTO J A, et al. Ethanol tolerance of five non-Saccharomyceswine yeasts in comparison with a strain ofSaccharomycescerevisiae: influence of different culture conditions[J]. Food Microbiology, 2004, 21(4): 439-447.
[3] 楊瑩, 徐艷文, 薛軍俠, 等. WL營養瓊脂對葡萄酒相關酵母的鑒定效果驗證[J]. 微生物學雜志, 2007, 29(5): 75-78.
[4] REGODON M J A, PEREZ N F, FERNANDEZ M R, et al. Influence ofSaccharomyces cerevisiaeyeast strain on the major volatile compounds of wine[J]. Enzyme and Microbial Technology, 2006, 40(1): 151-157.
[5] DOMIZIO P, LENCIONI L, CIANI M, et al. Spontaneous and inoculated yeast populations dynamics and their effect on organoleptic characters of Vinsanto wine under different process conditions[J]. International Journal of Food Microbiology, 2007, 115(3): 281-289.
[6] ESTEVE Z B, BELLOCH C, URUBURUL F, et al. Identification of yeasts by RFLP analysis of the 5.8S rRNA gene and the two ribosomal internal transcribed spacers[J]. International Journal of Systematic Bacteriology, 1999, 49(1): 329-337.
[7] SABATE J, CANO J, ESTEVE Z B, et al. Isolation and identification of yeasts associated with vineyard and winery by RFLP analysis of ribosomal genes and mitochondrial DNA[J]. Microbiological research,2002, 157(4): 267-274.
[8] MERCADOA L, DALCERO A, MASUEKKI R, et al. Diversity ofSaccharomycesstrains on grapes and winery surfaces: analysis of their contribution to fermentative flora of Malbec wine from Mendoza (Argentina)during two consecutive years[J]. Food Microbiology, 2007, 24(4): 403-412.
[9] CIANI M, MANNAZZU I, MARINANGELI P, et al. Contribution of winery-residentSaccharomyces cerevisiaestrains to spontaneous grape must fermentation[J]. Food Microbiology, 2004, 85(2): 159-164.
[10] JEUNE L C, ERNY C, DEMUYTER C, et al. Evolution of the population ofSaccharomyces cerevisiaefrom grape to wine in a spontaneous fermentation[J]. Food Microbiology, 2006, 23(8): 709-716.
[11] 薩姆布魯克 J, 弗里奇 E F, 曼尼阿蒂斯 T. 分子克隆實驗指南[M].金冬雁, 譯. 北京: 科學出版社, 1996: 465-467.
[12] 王澤舉. 新疆、甘肅地區葡萄酒相關酵母菌的鑒定及多樣性研究[D]. 楊凌: 西北農林科技大學, 2008.
[13] 龐紅勛, 崔艷, 劉金福, 等. 本土葡萄酒酵母的選育及發酵性能[J].食品研究與開發, 2010(6): 169-174.
[14] 薛軍俠, 徐艷文, 楊瑩, 等. WL培養基在釀酒酵母篩選中的應用[J].中國釀造, 2007(9): 36-39.
[15] 焦紅茹, 劉樹文, 祖顯生, 等. 赤霞珠相關酵母菌的分離及其分子生物學鑒定[J]. 釀酒科技, 2007(12): 17-20.
[16] GUILLAMON J M, SABATE J, BARRIO E, et al. Rapid identification of wine yeast species based on RFLP analysis of the ribosomal ITS regions[J]. Archives of microbiology, 1998, 169(5): 387-392.
[17] PRETORIUS I S, WESTHUIZEN T J, AUGUSTYN O. Yeast biodiversity in vineyards and wineries and its importance to the South African wine industry[J]. South African Journal for Enology & Viticulture,1999, 20(2): 61-74.
[18] 李艷, 盧君, 張利中, 等. 沙城龍眼葡萄自然發酵過程相關酵母生物多樣性研究[J]. 食品科學, 2009, 30(21): 237-240.
[19] 李艷, 盧君, 崔建東, 等. 分子生物學方法區分鑒定五種常用葡萄酒活性干酵母[J]. 食品科學, 2009, 30(19): 200-202.
Diversity ofSaccharomyces cerevisiaein Different Habitats in Shacheng Region
ZHAO Jing-jing1,LI Yan1,2,*
(1. College of Bioscience and Bioengineering, Hebei University of Science and Technology, Shijiazhuang 050018, China;
2. Research Center for Fermentation Engineering of Hebei Province, Shijiazhuang 050018, China)
In this study, the biodiversity ofSaccharomyces cerevisiaeisolated from different environments associated with Longan grape in Shacheng wine-producing region was investigated. In the continuous three years 2008, 2009 and 2010, 227 samples were collected from vineyard soil, the surface of winery equipments, grape juice contacted with winery equipments and naturally fermented grape and 1358 yeast strains were isolated from them. Molecular-level identification based on a PCR-RFLP analysis of 5.8S ITS rRNA and secondary screening using lysine medium were carried out to obtain 270Saccharomyces cerevisiaestrains. Further, the strains were divided using Interdelta PCR into 16 classes, including 5 from vineyard soil, 4, 10 and 11 from at the 2nd, 3rdand 4thstages of natural fermentation respectively, 3 from the surface of winery equipments and 3 from grape juice contacted with winery equipments.Saccharomyces cerevisiaespecies notably varied with sampling time and habitat.Saccharomyces cerevisiaestrains isolated from naturally fermented grape had the greatest possibility of being indigenous strains.
Saccharomyces cerevisiae;restriction pattern of amplified 5.8S-ITS rRNA gene (RFLP);Interdelta PCR;biodiversity;indigenous yeast
Q78
A
1002-6630(2012)05-0224-05
2011-10-24
河北省科技支撐計劃項目(092210003D);河北省自然科學基金項目(C2011208028)
趙靜靜(1985—),女,碩士研究生,研究方向為傳統發酵工程創新技術。E-mail:576556889@qq.com
*通信作者:李艷(1958—),女,教授,本科,研究方向為葡萄酒科學技術。E-mail:lymdh5885@163.com

