高羊茅FaHsfC1b啟動子克隆及外源激素處理下FaHsfC1b的表達分析
曹 薇, 楊志民, 莊黎麗, 王 劍, 孫 巖
(南京農業大學草業學院,江蘇南京 210095)
作為一種多年生冷季型草坪草,高羊茅(Festucaarundinacea)具有抗病蟲害、成坪快、耐修剪等多種優點,因此在我國溫帶南部和亞熱帶地區得到了廣泛的種植。在溫帶以北,如北京地區,高羊茅在越冬時遭受冷脅迫,導致其通常活不過2年,被稱為“短命草”,而在亞熱帶地區,夏季高溫以及高溫帶來的缺水也嚴重影響著高羊茅的生長,提高高羊茅對非生物脅迫的抵抗能力有益于節約草坪管理成本,進一步擴大高羊茅草坪草在我國的使用范圍。
植物熱激轉錄因子(heat stress transcription factors,Hsfs)是存在于植物細胞內調節熱激蛋白表達的一類轉錄調節基因,在植物熱脅迫信號轉導和耐熱性的產生過程中具有重要作用[1]。熱激轉錄因子在結構上可以分為3類:A類、B類、C類。A類熱激轉錄因子是熱激基因表達的激活劑,在不同的植物中具有不同的應激反應[1],多項研究表明,此類熱激轉錄因子在植物抗逆境尤其是抗熱方面起到重要作用[2-4]。B類熱激轉錄因子本身沒有轉錄激活功能,在番茄抗熱性形成的不同階段中,HsfB1和HsfA1a可以形成一個增效的共激活子[5]。另外,在熱脅迫下HsfB1可以跟其他熱激轉錄因子結合控制或抑制看家基因的表達[6]。與A類Hsfs相類似,熱脅迫下的轉錄組分析顯示,C類熱激轉錄因子HsfCs在水稻(Oryzasativa)、小麥(Triticumaestivum)、白菜(Brassicapekinensis)、椪柑(CitrusreticulataBlanco)、胡蘿卜(Daucuscarota)、豌豆(Pisumsativum)、簸箕柳(Salixsuchowensis)中普遍上調表達,在其中某些物種中,HsfCs也響應滲透脅迫、冷脅迫、ABA[7-13]。到現在為止,對于C類熱激轉錄因子的研究較少,僅發現水稻缺失OsHsfC1b后,對鹽脅迫、干旱脅迫及ABA敏感,在非脅迫條件下,OsHsfC1b對水稻的生長是不可缺少的[14]。目前對多年生草坪草尤其是高羊茅中C類熱激轉錄因子的功能及其上游調控因子缺乏研究。
啟動子是DNA分子與RNA聚合酶特異結合的部位,負責下游基因的轉錄激活,對于基因的表達非常重要。伊淑瑩[15]等研究獲得了番茄線粒體小分子熱激蛋白基因LeMTsHSP起始密碼子上1 915 bp的調控序列,可能對該基因的表達具有增強作用。高溫下小麥(Triticumaestivum)Hvhsp17啟動子能驅動Hvhsp17在小麥中表達[16]。在不同類型的啟動子中,有一類被稱為激素誘導型啟動子,激素首先與植物體內的特定受體相結合,受體蛋白被激活后與啟動子結合,從而激活下游基因的表達,使植物發生一系列生理反應。如果植物缺少相應的受體基因或啟動子,就不會對激素起反應[17]。目前研究較多的有水楊酸(SA)、脫落酸(ABA)、赤霉素(GA)、生長素(IAA、NAA等)等激素響應啟動子。不同激素誘導型啟動子需要具有相應的激素響應元件,比如在擬南芥RD29B啟動子中存在2個ABA響應元件ABRE,只有這2個元件相互作用形成ABA應答復合體,才能使其所在的啟動子表現出ABA誘導活性[18]。
植物激素在植物抗逆性中發揮著重要的作用,ABA含量在大豆熱激恢復階段會明顯快速升高[19-20],而ABA不敏感型突變體abi1和abi2則表現為耐熱性變差[21]。水稻中的OsHsfC1b被證實依賴ABA提高水稻對鹽脅迫的抗性[14],并且已經在A類的熱激轉錄因子中發現了富集ABA應答原件(ABRE)的順式調控模塊,這表明了ABA在一些熱激轉錄因子轉錄調控中的介導作用[22]。SA能夠在植物遭受逆境時及時清除ROS,維持抗氧化酶的活性,從而保護脅迫下細胞膜的穩定性[23]。生長素和赤霉素作為植物激素普遍參與了植物在正常條件和脅迫條件下的各種生理機制[24-27]。研究發現,大麥和擬南芥在熱脅迫下,伴隨著內源生長素含量的降低,編碼生長素合成的關鍵基因表達量也出現下調,并導致雄性不育,這些性狀可以通過外施生長素得到恢復[28]。赤霉素還參與調解生長素/細胞分裂素的拮抗作用[29]。
基于已有高羊茅品種凌志的全長序列,我們設計 5′RACE 引物,通過染色體步移法分離FaHsfC1b上游啟動子并克隆,隨后利用啟動子分析網站對該基因啟動子區域的順式作用調控元件及其功能進行分析,并根據啟動子中出現的激素響應元件,對激素處理下FaHsfC1b的表達量進行了測定,從而為高羊茅C類熱激轉錄因子的分子調控機制研究提供科學依據。
1 材料與方法
1.1 植物材料與菌株
試驗于2015年9—12月在南京農業大學草業科學學院草坪分子生物學實驗室完成。高羊茅(Festucaarundinacea)品種凌志材料為筆者所在實驗室草坪種質資源圃自有品種。選取實驗室資源圃高羊茅植株,洗去根部泥土并去掉枯葉后將植株分蘗從整株上分離下來,剪去根部,將分蘗種植于霍格蘭營養液[30]中,營養液上置泡沫板以海綿包裹分蘗節支撐植株,按照1/3原則修剪葉片,培養條件為白天、夜晚各12 h,白天溫度25 ℃,夜晚溫度20 ℃。其后每周換1次營養液,待 2~3周后,分蘗節發育成較大植株,側根也長出10~15 cm,即可更換營養液并對高羊茅進行處理。
pMD19SimpleT載體購自TaKaRa公司,大腸桿菌菌株DH5α為筆者所在實驗室凍存。引物合成及DNA測序均在南京思普金生物公司完成。
1.2 方法
1.2.1 高羊茅FaHsfC1b啟動子的克隆 高羊茅FaHSFC1b啟動子的克隆采用染色體步行法,具體步驟參考李廣平等的方法[31]和BD Genome WalkerTMUniversal Kit User Manual說明書。2條基因特異反向引物分別為:NGSP1:5′-TCGT-GCTCTGCTCGGCGTGGT-3′;NGSP2:5′-TACCTCGCTCTGT-GCGTGCCG-3′。重新驗證啟動子所設計的引物序列為:F:5′-CAGAGCAGAAGTGGTGATGGT-3′;R:5′-GAGGAG-GAAGTCGGAGAAGGC-3′。
1.2.2FaHsfC1b啟動子序列分析 采用GenBank在線比對工具BLASTn(http://blast. ncbi. nlm.nih. gov/)[32]進行了序列同源性分析,啟動子序列調控元件分析利用植物順勢調控元件數據庫PlANTCARE(http://bio informatics. psb. ugent. be/webtools/plantcare/html/)[33]和PLACE(http://www. dna. affrc. go. jp/PLACE/signalscan.html)[34]完成。
1.2.3 外源激素處理 將水培2周左右的高羊茅植株轉移到分別含有ABA(100 μmol/L)、GA(90 mg/L)、IAA(60 mg/L)、SA(0.5 mmol/L)、ZT(0.5 mg/L)的營養液中處理24 h,在0、4、12、24 h取根部和葉片,每個處理取3個重復,培養條件與處理前相同。
1.2.4 高羊茅總RNA的提取與基因表達分析 總RNA提取采用試劑盒Plant RNA Kit(50)(Omega Bio-Tec,American),用于熒光定量PCR的cDNA采用試劑盒PrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)(TaKaRa,Otsu,Japan)合成,通過qRT-PCR檢測FaHsfC1b在不同激素處理下的表達情況。采用SYBR Green Ⅰ Master反應體系(Roche Diagnostic,Rotkreuz,Switzerland),在LightCycler 480 Ⅱ(Roche,Rotkreuz,Switzerland)儀器中進行。以FaTublin作為內參基因,使用如下引物:RT-FaHsfC1b-F:ACTTCAAGCACCGCAACTTT;RT-FaHsfC1b-R:TGAACCGGCCTTCTTCTTCT;RT-FaTublin-F:ATGCTTT-CGTCTTATGCCC;RT-FaTublin-R:CTCTTGGTTTTGATGG-TTGC。
2 結果與分析
2.1 FaHsfC1b啟動子克隆
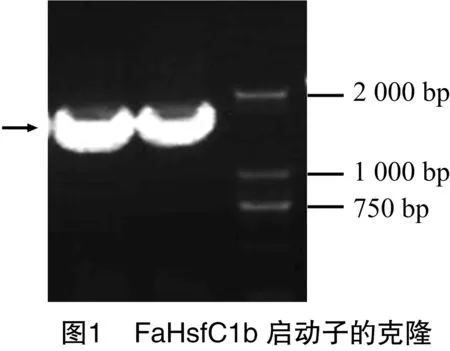
構建的獲得克隆經測序后與已有序列進行拼接和比對,以構建的高羊茅葉片基因組酶解DNA片段文庫為模板,經過2輪PCR后,從高羊茅的EcoRⅠ酶切體系中擴增到1條 1 562 bp 左右的片段(圖1)。將該片段進行克隆,連接到pMD19SimpleT并測序,結果顯示,獲得的片段實際長度為 1 514 bp。與已有的高羊茅FaHSFC1b基因(登錄號:KY475613)序列比對后發現所得到的序列與高羊茅FaHSFC1b序列有95 bp的重疊區,表明所得到的序列是其上游的DNA序列。進一步設計1對驗證引物,克隆分析整理后,共得到高羊茅FaHSFC1b基因上游長度為1 657 bp的DNA序列。
2.2 FaHsfC1b啟動子序列生物信息學分析
通過順式作用元件數據庫PLACE和PlANTCARE對FaHsfC1b啟動子的分析,結果發現FaHsfC1b啟動子很多個位點上都有啟動子核心元件TATA-box,即RNA聚合酶結合位點,確保了轉錄起始位點的高度靈活性,多個CAAT-box則控制著轉錄起始的頻率(表1)。
除了核心元件,FaHsfC1b啟動子中包含的元件按照功能大致可以分為6類(表1)。第一類為光信號相關元件,在啟動子中數量最多、范圍最廣,多達16種,有-10PEHVPSBD、box Ⅱ、chs-Unit 1 m1、G-box、GAG-motif、GBOXLERBCS、I-box、INRNTPSADB、MNF1、MRE、PRECONSCRHSP70A、REBETALGLHCB21、SORLIP2AT、SORLIP1AT、Sp1、TBOXATGAPB。第二類是與抵抗逆境相關的元件,集中在干旱、冷/寒、鹽及病害方面,包括綜合性防御元件(同時具備抵抗2種或2種以上逆境相關元件):DOFCOREZM(多種脅迫防御)、DPBFCOREDCDC3(病原與高鹽脅迫)、GT1GMSCAM4(病害與鹽脅迫)包括脫水響應元件:DRECRTCOREAT、MYCATRD22;低溫響應元件:LTRECOREATCOR15、LTRE1HVBLT49;抗病相關元件:GT1CONSENSUS、SEBFCONSSTPR10A、WBOXATNPR1。還有一種缺氧相關的缺氧特異性誘導增強元件:GC-motif,猜測可能與澇害相關。第三類是組織特異性元件,它們參與調控基因在不同植物組織中的表達,包括分生組織:CAT-box;葉肉細胞:CACTFTPPCA1;花粉:GTGANTG10、POLLEN1LELAT52;根瘤組織:OSE1ROOTNODULE、OSE2ROOTNODULE;根毛:RHERPATEXPA7;種子:RYREPEATBNNAPA;胚乳:Skn-1_motif。第四類為特殊物質響應元件,有蔗糖應答元件:RAV1AAT、SREATMSD;銅應答元件:CURECORECR;硫應答元件:SURECOREATSULTR11。第五類是一些與基因、酶及蛋白結合的位點。
此次研究重點觀察到的第六類是激素響應元件,包括細胞分裂素調節基因ARR1結合位點:ARR1AT;脫落酸響應元件:ABRE、MYB2CONSENSUSAT;生長素響應元件:CATATGGMSAUR;乙烯響應元件:GCCCORE、WBOXNTERF3;水楊酸響應元件:TCA-element;赤霉素響應元件:PYRIMIDINEBOXOSRAMY1A、WRKY710S,另外WRKY710S元件也與創傷有關;調節類黃酮生物合成元件:MYBCORE。
綜合上述分析,可知克隆到的啟動子序列可能與光照周期、光照度有關,并且可能通過響應激素誘導,調控下游基因表達,在植物生長、抗病、抗逆方面發揮作用。
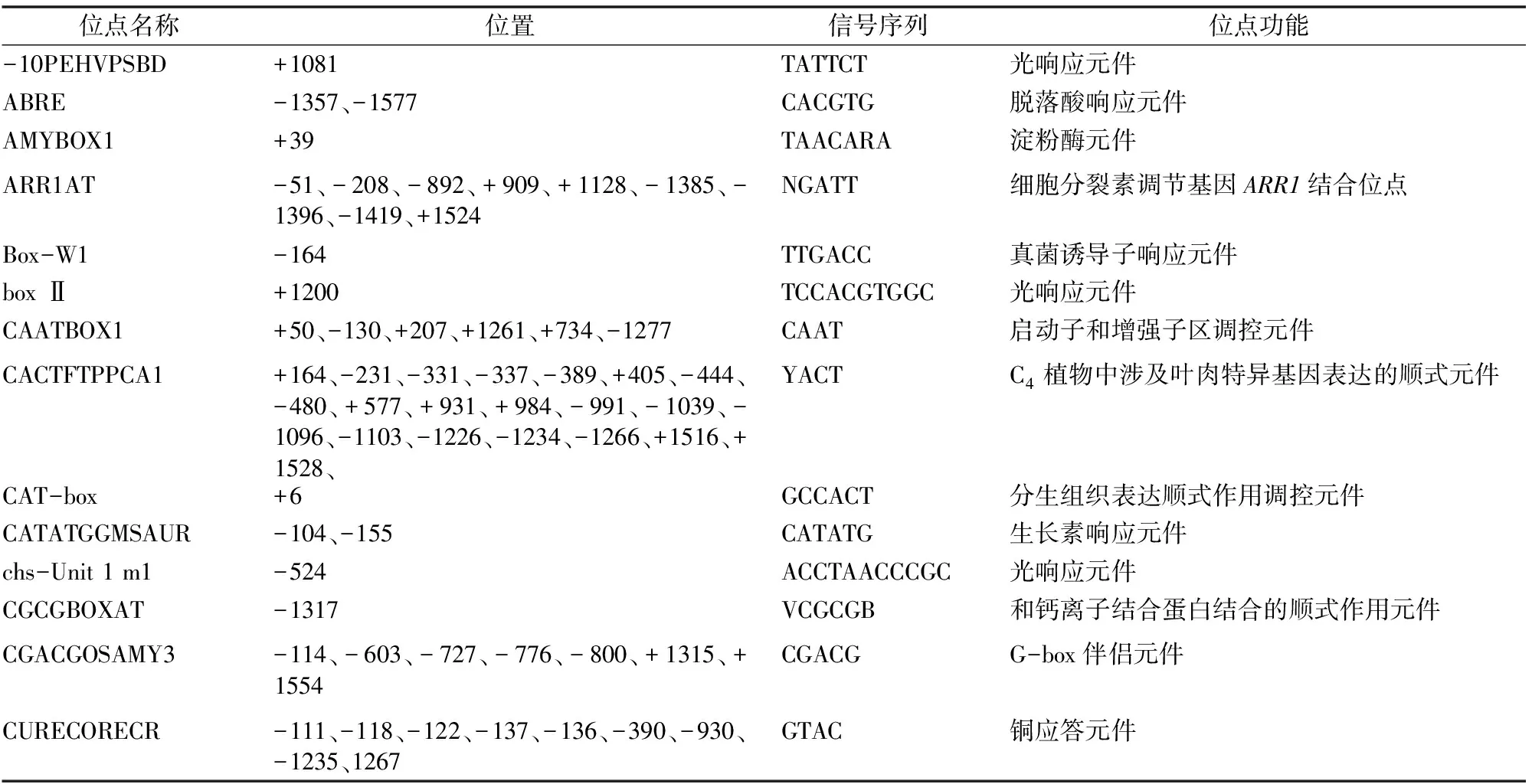
表1 應用PLANTCARE預測FaHsfC1b啟動子順式作用元件

位點名稱位置信號序列位點功能DOFCOREZM+22、+238、-407、+857、-1078、+1101、-1134、-1144、+1285、-1296、+1460、-1623、AAAG防御與脅迫響應元件DPBFCOREDCDC3+147、-875、-1250、+1343、+1357、+1574ACACNNG病原和高鹽脅迫響應元件DRECRTCOREAT-801、-1481RCCGAC脫水響應元件EBOXBNNAPA-96、-104、-989、-1242、-1344、1346、-1358、-1432、-1486、CANNTG儲藏蛋白napA的E-boxEECCRCAH1+128、+340、+748、-1120、-1521GANTTNCMyb轉錄因子LCR1結合位點ELRECOREPCRP1-320TTGACC激發響應元件EMBP1TAEM+1358CACGTGGC抗旱、耐寒相關的反式作用因子GCCCORE-860、-882、+899GCCGCC乙烯響應元件G-Box-1202、+1421CACGTG光順式作用調控元件GATABOX-47、-844、-1158、+1514、-1563、-1629GATAASF-2結合位點GAG-motif+310、+1149、471GGAGATG光響應元件GBOXLERBCS+1357MCACGTGGC光響應基因的上游保守序列GT1CONSENSUS-45、+1150、+1276、+1282、+1558GRWAAW調控抗病基因PR-1a表達,影響水楊酸誘導的基因表達GT1GMSCAM4+17、+1282GAAAAA在病原或鹽誘導的SCaM-4基因表達中發揮作用GTGANTG10+30、+68、-87、+99、+315、-328、+338、-412、-821、-959、+1227、-1409GTGA花粉晚期基因啟動子的元件GC-motif+1394CCCCCG缺氧特異性誘導增強元件Ⅰ-box-1626、-46GATAA部分光響應元件INRNTPSADB+355、-1220、-1277YTCANTYY光響應元件LECPLEACS2-191TAAAATATLeCp結合的順式元件LTRE1HVBLT49+854CCGAAA低溫響應元件LTRECOREATCOR15-810、-812、+1482、-1491、+1553CCGAC低溫響應元件MNF1+56GTGCCC(A/T)(A/T)光響應元件MRE-341AACCTAA光響應MYB結合元件MYBCORE-561、-1464CNGTTR調節類黃酮(一類植保素)生物合成MYB2CONSENSUSAT+561YAACKG脫落酸信號轉錄激活因子MYCATRD22-96、+1344CACATG脫水響應元件OSE1ROOTNODULE+238AAAGAT根瘤組織特異性表達元件OSE2ROOTNODULE+427、+1076CTCTT根瘤組織特異性表達元件RAV1AAT+915、-1116、-1436CAACA蔗糖應答元件REBETALGLHCB21-1629CGGATA光敏色素調控元件RHERPATEXPA7-210、-605、+1606KCACGW根毛特異元件POLLEN1LELAT52+16、-408、-507、-674、-712、-1071、-1131、-1145、-1642AGAAA花粉特異表達的順式作用元件ROOTMOTIFTAPOX1+191、-1561ATATTrolD的啟動子元件PRECONSCRHSP70A-758、+886、+1372、+1553SCGAYNRNNNNNNNNNNNNNNNHD光響應元件PROLAMINBOXOSGLUB1+1098TGCAAAG葡聚糖酶1醇溶谷蛋白元件PYRIMIDINEBOXOSRAMY1A-1284CCTTTT赤霉素響應元件RYREPEATBNNAPA+1034CATGCA在種子特異性表達中發揮作用SEBFCONSSTPR10A+761、-1324YTGTCWC抗病相關元件SEF4MOTIFGM7S+263、-1402RTTTTTRSEF4結合位點Skn-1_motif+956GTCAT胚乳高水平表達順式作用調控元件SORLIP2AT+160、-863、-947、+1030、+1139、-1363GGGCC光響應元件SORLIP1AT+162、-272、-550、-633、-689、-734、-880、+1311、-1349、-1361、+1538GCCAC光響應元件
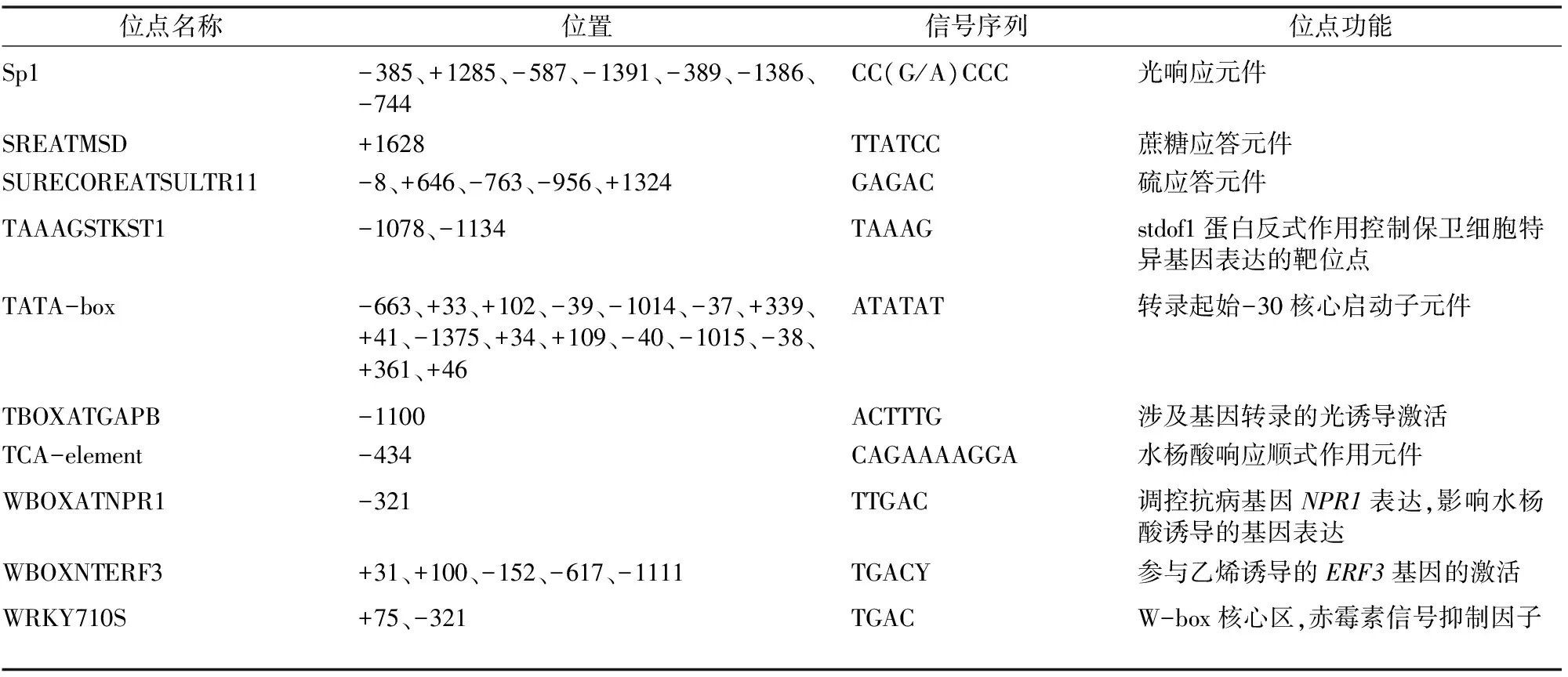
位點名稱位置信號序列位點功能Sp1-385、+1285、-587、-1391、-389、-1386、-744CC(G/A)CCC光響應元件SREATMSD+1628TTATCC蔗糖應答元件SURECOREATSULTR11-8、+646、-763、-956、+1324GAGAC硫應答元件TAAAGSTKST1-1078、-1134TAAAGstdof1蛋白反式作用控制保衛細胞特異基因表達的靶位點TATA-box-663、+33、+102、-39、-1014、-37、+339、+41、-1375、+34、+109、-40、-1015、-38、+361、+46ATATAT轉錄起始-30核心啟動子元件TBOXATGAPB-1100ACTTTG涉及基因轉錄的光誘導激活TCA-element-434CAGAAAAGGA水楊酸響應順式作用元件WBOXATNPR1-321TTGAC調控抗病基因NPR1表達,影響水楊酸誘導的基因表達WBOXNTERF3+31、+100、-152、-617、-1111TGACY參與乙烯誘導的ERF3基因的激活WRKY710S+75、-321TGACW-box核心區,赤霉素信號抑制因子
2.3 不同激素處理下FaHsfC1b的表達水平分析
為了驗證FaHsfC1b啟動子序列上存在的激素響應元件是否與FaHsfC1b在激素下的表達調控有關,我們對高羊茅進行了激素處理試驗(圖2),發現在ABA、GA、IAA、SA、ZT處理下,24 h內FaHsfC1b表達量均呈現上升再下降的趨勢。ABA處理下,高羊茅根部FaHsfC1b表達量在4 h開始上升并在 12 h 達到峰值,為對照的49倍,葉片表達水平比根部低,同樣在4 h開始上升并在12 h達到峰值,為對照的19倍。GA處理下根部和葉片中FaHsfC1b表達量都在4 h達到峰值,為對照的7~8倍,隨后葉片中基因表達量雖有下降但依舊顯著高于對照,而根部表達量則下降到處理前水平。IAA處理下高羊茅根部FaHsfC1b表達量明顯快于也高于葉片中基因表達量,根部基因表達量在4 h達到峰值,并且是對照的42倍,而葉片則在12 h開始出現基因表達量的上升,與根部相比上升幅度較小,峰值為對照的11倍。SA和ZT處理下FaHsfC1b表達量在根部和葉片中的上升/下降趨勢很相似,但SA處理下高羊茅根部基因表達量為所有處理中最高水平,在4 h達到峰值,為對照的65倍,葉片中FaHsfC1b表達量則在12 h開始上升并在24 h達到峰值,為對照的11倍;在ZT處理下,高羊茅根部FaHsfC1b表達量同樣在4 h達到峰值,為對照的15倍,葉片在24 h達到峰值,是對照基因表達量的24倍。

FaHsfC1b對以上5種激素的響應與啟動子元件中5種激素響應元件相吻合,證明啟動子所包含的響應元件與下游基因的調控表達有一定聯系。
3 討論與結論
FaHsfC1b啟動子分析表明,該啟動子序列中含有豐富的順勢作用元件和其他應答元件。參與低溫響應的元件有6個,抗旱相關元件有4個,鹽脅迫相關元件有8個,抗病相關元件有3個,推測FaHsfC1b可能與高羊茅抗寒、抗旱及抗鹽相關,而已有的研究結果表明,HsfC1在滲透脅迫、鹽脅迫和冷脅迫中的表達都很突出[35,37],將水稻中的OsHsfC1b敲除后,轉基因水稻對滲透脅迫的抗性明顯弱于野生型[14]。
在所有順式作用元件中,數量最多的是涉及C4植物葉肉特異表達的順式元件CACTFTPPCA1,此元件是維持C4植物葉肉特異性,因而得以更高效率地利用光照和氮資源的關鍵元件[38],另一個發現是關于光照的元件,在所有元件中光響應元件無論在種類還是數量上都是最多的,比較前人對其他基因啟動子的分析,我們可以推測FaHsfC1b可能存在復雜的光應激反應表達模式,在植物正常的光合作用中起到重要作用。此外,FaHsfC1b啟動子中還包含有多個蔗糖應答元件、銅應答元件、硫應答元件、真菌誘導子元件,猜測外源施加營養、金屬元素或真菌感染可能會對FaHsfC1b的轉錄發生影響。啟動子中含有的其他基因、酶及蛋白結合的位點說明植物對外界的應激反應是一個復雜的機制,需要多種物質共同協同作用。
對FaHsfC1b啟動子序列分析的另一結果顯示,激素響應元件所響應的激素不僅涵蓋了五大類激素:生長素、細胞分裂素、赤霉素、脫落酸、乙烯,還包括水楊酸、類黃酮,其中細胞分裂素調節基因ARR1結合位點多達8個,在啟動子的末梢還有個分生組織表達順式作用調控元件CAT-box,猜測FaHsfC1b的轉錄表達可能對植物的生長有影響。大多數干旱應答基因都受脫落酸的誘導,已經在A類熱激轉錄因子中發現了富集脫落酸應答元件的啟動子序列[39],這表明脫落酸在C類熱激轉錄因子轉錄調控中可能也有一定的介導作用。組織特異性元件的富集可推測FaHsfC1b的轉錄調控遍布高羊茅植株的各個部位。
在本試驗中,FaHsfC1b響應IAA、ZT、GA、ABA和SA,在FaHsfC1b啟動子中,生長素、赤霉素、脫落酸響應元件都有不止1個,而研究表明,啟動子中多拷貝應答元件可能參與下游基因的增強表達[40]。當干旱脅迫應答基因上游啟動子序列中含有ABRE元件時,該基因的表達調節依賴ABA調控途徑,本課題組前期對FaHsfC1b的研究表明FaHsfC1b響應干旱脅迫、冷脅迫和鹽脅迫,而對OsHsfC1b的研究表明其維持水稻耐旱性依賴于ABA調控途徑,因此進一步猜測FaHsfC1b可能也具有相同功能[14]。另外研究指出生長素誘導型啟動子往往同時含有干旱脅迫或其他脅迫相關的作用元件,表明啟動子下游基因的調控依賴于激素和逆境脅迫的共同作用[41]。
目前除ABA以外,C類熱激轉錄因子在其他植物外源激素處理下的表達分析尚無報道,因此猜測植物激素可能通過啟動子來調控FaHsfC1b的表達,進而在植物抗逆性中發揮作用。由于此類激素尤其是生長素、細胞分裂素和赤霉素和植物生長密切相關,結合水稻中的OsHsfC1b在水稻生長中所扮演的不可缺少的角色[14],進一步猜測FaHsfC1b可能也在高羊茅的生長發育中發揮重要作用。
本研究通過對高羊茅FaHsfC1b啟動子序列的分析,推測FaHsfC1b可能在植物激素介導的抗逆性中發揮重要作用,對于植物生長發育及正常的光合作用可能也是必不可少的。
參考文獻:
[1]黃小云,陶 鵬,李必元,等. 植物熱激轉錄因子基因家族的研究進展[J]. 浙江農業科學,2014(9):1323-1332,1336.
[2]Chen H,Hwang J E,Lim C J,et al. Arabidopsis DREB2C functions as a transcriptional activator of HsfA3 during the heat stress response[J]. Biochemical and Biophysical Research Communications,2010,401(2):238-244.
[3]Czarneckaverner E,Yuan C X,Fox P C,et al. Isolation and characterization of six heat shock transcription factor cDNA clones from soybean[J]. Plant Molecular Biology,1995,29(1):37-51.
[4]Yokotani N,Ichikawa T,Kondou Y,et al. Expression of rice heat stress transcription factor OsHsfA2e enhances tolerance to environmental stresses in transgenicArabidopsis[J]. Planta,2008,227(5):957-967.
[5]Bharti K,Koskull-D?ring P V,Bharti S,et al. Tomato heat stress transcription factor HsfB1 represents a novel type of general transcription coactivator with a histone-like motif interacting with the plant CREB binding protein ortholog HAC1[J]. The Plant Cell,2004,16(6):1521-1535.
[6]Scharf K D,Berberich T,Ebersberger I,et al. The plant heat stress transcription factor (Hsf) family: structure,function and evolution[J]. Biochimica et Biophysica Acta,2012,1819(2):104-119.
[7]Hu W,Hu G,Han B. Genome-wide survey and expression profiling of heat shock proteins and heat shock factors revealed overlapped and stress specific response under abiotic stresses in rice[J]. Plant Science,2009,176(4):583-590.
[8]Xue G P,Sadat S,Drenth J,et al. The heat shock factor family fromTriticumaestivumin response to heat and other major abiotic stresses and their role in regulation of heat shock protein genes[J]. Journal of Experimental Botany,2014,65(2):539-557.
[9]Huang X Y,Tao P,Li B Y,Wang W H,et al. Genome-wide identification,classification,and analysis of heat shock transcription factor family in Chinese cabbage (Brassicarapapekinensis)[J]. Genetics and Molecular Research,2015,14(1):2189-2204.
[10]Lin Q,Jiang Q,Lin J,et al. Heat shock transcription factors expression during fruit development and under hot air stress in Ponkan (CitrusreticulataBlanco cv. Ponkan) fruit[J]. Gene,2015,559(2):129-136.
[11]Huang Y,Li M Y,Wang F,et al. Heat shock factors in carrot: genome-wide identification,classification,and expression profiles response to abiotic stress[J]. Molecular Biology Reports,2015,42(5):893-905.
[12]Li P S,Yu T F,He G H,et al. Genome-wide analysis of the Hsf family in soybean and functional identification of GmHsf-34 involvement in drought and heat stresses[J]. BMC Genomics,2014,15:998-1009.
[13]Zhang J,Li Y,Jia H X,et al. The heat shock factor gene family inSalixsuchowensis:a genome-wide survey and expression profiling during development and abiotic stresses[J]. Frontiers in Plant Science,2015,6:735-748.
[14]Schmidt R,Schippers J H,Welker A,et al. Transcription factor OsHsfC1b regulates salt tolerance and development inOryzasativassp.japonica[J]. AoB Plants,2012,2012(1):11-23.
[15]伊淑瑩,孫愛清,趙春梅,等. 番茄多脅迫誘導型LeMTshsp啟動子的分子克隆及其功能分析[J]. 云南植物研究,2007,29(2):223-230.
[16]Freeman J,Sparks C A,West J,et al. Temporal and spatial control of transgene expression using a heat-inducible promoter in transgenic wheat[J]. Plant Biotechnology Journal,2011,9(7):775-788.
[17]孫芳芳,宋洪元. 植物誘導型啟動子研究進展[J]. 南方園藝,2014(2):51-56.
[18]Yamaguchi-Shinozaki K,Shinozaki K. Organization of cis-acting regulatory elements in osmotic- and cold-stress-responsive promoters[J]. Trends in Plant Science,2005,10(2):88-94.
[19]Larkindale J,Huang B. Thermotolerance and antioxidant systems inAgrostisstolonifera:involvement of salicylic acid,abscisic acid,calcium,hydrogen peroxide,and ethylene[J]. Plant Physiology,2004,161(4):405-413.
[20]Liu N,Ko S,Yeh K C,et al. Isolation and characterization of tomatoHsa32 encoding a novel heat-shock protein[J]. Plant Science,2006,170(5):976-985.
[21]Larkindale J,Hall J D,Knight M R,et al. Heat stress phenotypes ofArabidopsismutants implicate multiple signaling pathways in the acquisition of thermotolerance[J]. Plant Physiology,2005,138(2):882-897.
[22]Jin G H,Gho H J,Jung K H. A systematic view of rice heat shock transcription factor family using phylogenomic analysis[J]. Journal of Plant Physiology,2013,170(3):321-329.
[23]Sakata T,Oshino T,Miura S,et al. Auxins reverse plant male sterility caused by high temperatures[J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(19):69-74.
[24]Sun T P,Gubler F. Molecular mechanism of gibberellin signaling in plants[J]. Annual Review of Plant Biology,2004,55(1):197-223.
[25]Fleet C M,Sun T P. A dellacate balance: the role of gibberellin in plant morphogenesis[J]. Current Opinion in Plant Biology,2005,8(1):77-85.
[26]Colebrook E H,Thomas S G,Phillips A L,et al. The role of gibberellin signalling in plant responses to abiotic stress[J]. Journal of Experimental Biology,2014,217(1):67-75.
[27]Branlard T. Proteomic analysis of the effect of heat stress on hexaploid wheat grain : characterization of heat-responsive proteins from non-prolamins[J]. Proteomics,2004,(4):505-513.
[28]Oshino T,Abiko M,Saito R,et al. Premature progression of anther early developmental programs accompanied by comprehensive alterations in transcription during high-temperature injury in barley plants[J]. Molecular Genetics and Genomics,2007,278(1):31-42.
[29]Vanstraelen M,Benková E. Hormonal interactions in the regulation of plant development[J]. Annual Review of Cell & Developmental Biology,2012,28(28):451-463.
[30]Hoagland D R,Arnon D I. The water-culture method for growing plants without soil[J]. Califagricexpstncirc,1937,347(5406):357-359.
[31]李廣平,張長青,章 鎮,等. 中國李pgip啟動子的克隆及調控元件分析[J]. 園藝學報,2009,36(10):1425-1430.
[32]Altschul S F,Madden T L,Sch?ffer A A,et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs[J]. Nucleic Acids Research,1997,25(17):389-402.
[33]Lescot M,Déhais P,Thijs G,et al. PlantCARE,a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research,2002,30(1):325-327.
[34]Higo K,Ugawa Y,Iwamoto M,et al. Plant cis-acting regulatory DNA elements (PLACE) database[J]. Nucleic Acids Research,1999,27(1):297-300.
[35]Kilian J,Whitehead D,Horak J,et al. The AtGenExpress global stress expression data set: protocols,evaluation and model data analysis of UV‐B light,drought and cold stress responses[J]. Plant Journal,2007,50(2):347-363.
[36]Wang J,Sun N,Deng T,et al. Genome-wide cloning,identification,classification and functional analysis of cotton heat shock transcription factors in cotton (Gossypiumhirsutum)[J]. BMC Genomics,2014,15:955-961.
[37]Reddy P S,Kavi Kishor P B,Seiler C,et al. Unraveling regulation of the small heat shock proteins by the heat shock factor HvHsfB2c in barley: its implications in drought stress response and seed development[J]. PloS One,2014,9(3):119-125.
[38]Udo Gowik J B,Meryem A,Ute S,et al. cis-Regulatory elements for Mesophyll-Specific gene expression in the C4plant flaveria trinervia,the promoter of the C4phosphoenolpyruvate carboxylase gene[J]. The Plant Cell,2004,16(5):1077-1090.
[39]Bechtold U,Albihlal W S,Lawson T,et al.ArabidopsisHEAT SHOCK TRANSCRIPTION FACTORA1b overexpression enhances water productivity,resistance to drought,and infection[J]. Journal of Experimental Botany,2013,64(11):3467-3481.
[40]Nishiuchi T,Shinshi H,Suzuki K. Rapid and transient activation of transcription of theERF3 gene by wounding in tobacco leaves: possible involvement of NtWRKYs and autorepression[J]. Journal of Biological Chemistry,2005,279(53):355-361.
[41]Liu Z B,Hagen G,Guilfoyle T J. A G-Box-Binding protein from soybean binds to the E1 auxin-response element in the soybean GH3 promoter and contains a proline-rich repression domain[J],Plant Physiology,1997,115(2):397-407.

