1spo0A基因缺失對Bacillus clausii發(fā)酵生產性能的影響
原梨萍1,肖靜2,王瑞明2,路福平1*,汪俊卿2,李玉1
(1.天津科技大學生物工程學院工業(yè)微生物教育部重點實驗室,天津300457;2.齊魯工業(yè)大學生物工程學院山東省微生物酶技術重點實驗室,山東濟南250353)
克勞氏芽孢桿菌(Bacillus clausii)是一類好氧型、內生抗逆孢子的革蘭氏陽性菌,具有很好的耐受性,能在極端環(huán)境下生長。近年來,芽孢形成及萌發(fā)過程中關鍵基因的研究[1-2]備受青睞,而克勞氏芽孢桿菌由于其生理特性及基因信息不明確,使其在芽孢方面的研究及工業(yè)上的應用較為落后。目前,國內外科研人員以模式微生物枯草芽孢桿菌為主對芽孢形成過程及關鍵基因的調控做了大量深入的研究[3-7],其中,以Spo0A、sigma因子及五種磷酸激酶基因的研究居多[8-13]。芽孢的生成是一個多層次分階段的過程,其中Spo0A是細胞從營養(yǎng)生長期進入芽孢形成時期的關鍵應答調節(jié)蛋白(responseregulatorprotein,RSP),Spo0A-PO4通過結合到基因“OA-box”序列上可調節(jié)500多種基因的表達。
目前國內外研究發(fā)現(xiàn),spo0A基因缺失對枯草芽孢桿菌、梭狀芽孢桿菌、蘇云金芽孢桿菌等多種芽孢桿菌的芽孢生成均產生有效的抑制作用[14-16],而且該基因一定程度上影響菌體生物量及代謝產物的產量,SANDOVAL N R[17]敲除梭狀芽孢桿菌spo0A基因后不僅對芽孢生成產生影響,而且有效提高了菌體生物量及丁醇產量。目前國內外關于spo0A基因缺失影響淀粉酶產量的報道少見。本實驗通過基因敲除技術構建spo0A基因缺失菌株,研究發(fā)現(xiàn)spo0A基因的缺失對菌體生物量、芽孢生成率及淀粉酶活力均產生影響,該基因在菌種改良上可以作為提高淀粉酶產量的靶點,對后續(xù)芽孢缺失型工業(yè)生產菌株的構建具有重要意義,為酶工業(yè)遺傳育種提供了新的選擇,也為高產淀粉酶芽孢桿菌工程菌株的構建奠定了基礎。
1 材料與方法
1.1 材料與試劑
1.1.1 菌株及質粒
克勞氏芽孢桿菌(Bacillus clausii)QL-1、含pHT01質粒的大腸桿菌(Escherichia coli)JM109:山東省微生物工程重點實驗室保存。
1.1.2 試劑
限制性核酸內切酶:日本TakaRa寶生物公司;氯霉素、Ezup柱式基因組脫氧核糖核酸(deoxyribonucleicacid,DNA)抽提試劑盒、SanPrep柱式DNA膠回收試劑盒:生工生物工程(上海)有限公司;高純度質粒小量快速抽提試劑盒:北京艾德萊生物科技有限公司;其他試劑均屬于國產分析純。引物由生工生物工程(上海)有限公司合成。
1.1.3 培養(yǎng)基
LB培養(yǎng)基:酵母浸粉5g/L、蛋白胨10g/L、氯化鈉10g/L、pH 7.0。
可溶性淀粉培養(yǎng)基:牛肉膏5 g/L、蛋白胨10 g/L、氯化鈉5 g/L、可溶性淀粉 2 g/L、pH 7.0~7.2、瓊脂20 g/L。
菌體增殖培養(yǎng)基:酵母浸粉5 g/L、蛋白胨10 g/L、氯化鈉10 g/L、山梨醇91 g/L。
菌體復蘇培養(yǎng)基:酵母浸粉5 g/L、蛋白胨10 g/L、氯化鈉10 g/L、山梨醇69 g/L、甘露醇91 g/L。
上述培養(yǎng)基均于121℃滅菌20 min。
1.2 儀器與設備
SW-CJ-2FD型雙人單面凈化工作臺:蘇州凈化設備有限公司;WFJ7200型可見分光光度計:尤尼柯(上海)儀器有限公司;4380C型電轉儀、5804R低溫冷凍離心機:德國Eppendorf公司;GNP-9080型隔水式恒溫培養(yǎng)箱:上海精宏實驗設備有限公司;Veriti96孔熱循環(huán)梯度聚合酶鏈式反應(polymerase chain reaction,PCR)儀:美國應用生物系統(tǒng)公司;MD2000核酸超微量分光光度計:美國BioFure公司;ZQYZ-CS型恒溫振蕩培養(yǎng)箱:上海知楚儀器有限公司;DYY-12電泳儀:北京市六一儀器廠;UVI Essential V6凝膠成像儀:英國Uvitec公司。
1.3 實驗方法
1.3.1 引物設計
采用Oligo軟件設計引物,引物見表1。

表1 本實驗所用引物Table 1 Primers used in the study
1.3.2 同源重組片段spo0A-Cmr的制備
同源重組片段spo0A-Cmr的制備流程見圖1。
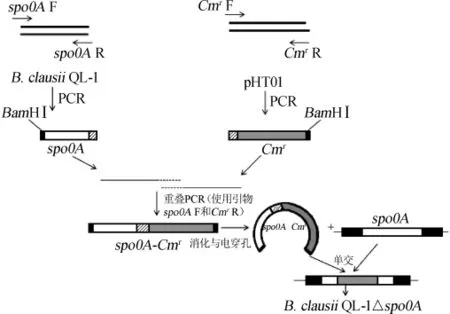
圖1 重組菌B.clausii QL-1△spo0A構建流程圖Fig.1 Flowchart of construction of recombinant strain B.clausii QL-1△spo0A
spo0A基因的調取:挑取B.clausiiQL-1單菌落轉接于50 mL LB液體培養(yǎng)基,37℃,200 r/min培養(yǎng)12 h,使用Ezup柱式基因組DNA抽提試劑盒對B.clausiiQL-1菌體基因組進行提取。1 μL基因組模板、1 μLspo0AF和1 μLspo0AR引物、12.5μL2×HiFi-PCRmaster、9.5μLddH2O組成的25μL反應體系進行PCR擴增,獲得堿基長度為418bp的spo0A基因片段,PCR擴增條件為:(1)95℃預變性5 min,95℃變性30 s,56 ℃退火30 s,72 ℃延伸45 s,30個循環(huán);(2)72 ℃再延伸10 min,4℃保存。
片段Cmr基因的調取:挑取大腸桿菌JM109單菌落轉接于50 mL LB液體培養(yǎng)基,37℃,200 r/min培養(yǎng)12 h,使用高純度質粒小量提取試劑盒提取pHT01質粒。1μLpHT01質粒模版、1 μLCmrF和1 μLCmrR引物、12.5 μL2×HiFi-PCR master、9.5 μL ddH2O組成的25 μL反應體系進行PCR擴增,獲得長度為1264bp的Cmr抗性基因片段,PCR擴增條件為:(1)95℃預變性5 min,95℃變性30 s,58℃退火30 s,72℃延伸2min35s,30個循環(huán);(2)72℃再延伸10min,4℃保存。
同源重組片段spo0A-Cmr的制備:使用SanPrep柱式DNA膠回收試劑盒回收所得的spo0A基因片段和Cmr抗性基因片段,首先進行第一步重疊PCR,2μLspo0A基因片段和2μLCmr抗性基因片段互為模板及引物、12.5 μL 2×HiFi-PCR master及3.5 μL ddH2O組成的25 μL反應體系進行重疊PCR擴增,PCR擴增條件為:(1)95℃預變性5min;95℃變性30s,58℃退火30s,72℃延伸2 min 35 s,8個循環(huán);向第一步反應液中加入1 μLspo0AF和1 μLCmrR引物、12.5 μL 2×HiFi-PCR master及10.5 μL ddH2O進行第二步大量擴增PCR,PCR擴增條件為:(1)95℃預變性5min;95℃變性30 s,58℃退火30 s,72 ℃延伸3 min 30 s,30個循環(huán);(2)72℃再延伸10 min,4℃保存。經(jīng)過上述兩步重疊延伸PCR擴增得到1648bp的同源重組片段spo0A-Cmr。使用SanPrep柱式DNA膠回收試劑盒回收所得的spo0A-Cmr片段,-20℃保存?zhèn)溆谩?/p>
1.3.3 同源重組片段spo0A-Cmr回收產物的酶切及濃縮
采用限制性核酸內切酶BamHⅠ對所得的spo0A-Cmr片段進行酶切,30 μL酶切體系為:25 μL PCR產物、1.5 μLBamHⅠ、3.5 μL 10×K Buffer,37 ℃靜置1.5 h。向酶切產物中加入醋酸鈉(1/10體積,3mol/L)和無水乙醇(25倍體積),混合后-20℃處理20 min,12 000 r/min離心8 min得DNA沉淀;加入體積分數(shù)為70%乙醇重懸DNA沉淀,12 000 r/mim離心8 min,除去乙醇,37℃風干,加入ddH2O重懸,制得300-500 ng/μL的spo0A-Cmr濃縮DNA溶液。
1.3.4 克勞氏芽孢桿菌B.clausiiQL-1感受態(tài)的制備
電轉緩沖液配制:山梨醇91 g/L、甘露醇91 g/L、甘油100 g/L。
將B.ClausiiQL-1單菌落接種于15 mL的LB液體培養(yǎng)基中,37℃、200 r/min振蕩培養(yǎng)12 h;將上述菌液以2%接種量轉接到50 mL LB培養(yǎng)基中,37℃、200 r/min振蕩培養(yǎng)至OD600nm值為1.0;4 ℃、8 000 r/min離心8 min收集菌體;電轉緩沖液洗滌3~4次后重懸、分裝,即為制備好的感受態(tài)。感受態(tài)制備過程中均保證4℃條件下操作。
1.3.5 重組菌B.clausiiQL-1△spo0A的構建
25 mg/mL氯霉素的制備:準確稱取氯霉素溶解于無水乙醇,溶解后過0.22 μm濾膜過濾除菌,分裝后于-20℃中保存。
感受態(tài)與spo0A-Cmr濃縮片段以10∶1(V/V)比例混合,1500V、5ms條件下進行電轉化,立即加入1 mL的菌體復蘇培養(yǎng)基,37℃,180 r/min復蘇4~5 h后涂布于氯霉素終質量濃度為25 μg/mL的LB固體培養(yǎng)基中培養(yǎng)18~24 h,篩選具有氯霉素抗性菌株。
1.3.6 陽性重組菌株的鑒定
以提取到的陽性菌株DNA為模板,spo0AF和CmrR為引物PCR擴增,擴增產物進行1%瓊脂糖凝膠電泳分析,最終獲得陽性重組菌株B.clausiiQL-1△spo0A。B.clausiiQL-1△spo0A重組菌于LB液體培養(yǎng)基中,在37℃、200r/min振蕩培養(yǎng)條件下傳代15次,進行菌落PCR檢測。
1.3.7 出發(fā)菌株及重組菌株芽孢形成檢測
將菌株B.clausiiQL-1與B.clausiiQL-1△spo0A分別以2%接種量接種于LB液體培養(yǎng)基,37℃、200 r/min振蕩培養(yǎng)28 h,將菌液于80℃水浴處理10 min。將80℃水浴處理過的出發(fā)菌株菌液稀釋10-6倍、重組菌株菌液不稀釋,同樣分別做三個平行,取100 μL涂布于LB固體平板上,將未處理的出發(fā)菌株及重組菌株的菌液分別稀釋10-6倍,做3個平行,取100 μL涂布于LB固體平板上,上述平板放置于37℃培養(yǎng)12~18 h,用平板計數(shù)法觀察并清點100 μL處理菌液及未處理菌液的芽孢數(shù)及菌體數(shù)(CFU/mL)。芽孢生成率計算公式如下:

1.3.8B.clausiiQL-1△spo0A重組菌生長曲線及干質量的測定
生長曲線的繪制:挑取菌株B.clausiiQL-1與B.clausiiQL-1△spo0A單菌落于15mLLB液體培養(yǎng)基,37℃、200r/min振蕩培養(yǎng)12h,將菌液轉接至100 mLLB液體培養(yǎng)基至初始OD600nm值為0.1左右,繼續(xù)培養(yǎng),每隔4 h取樣4 mL,使用紫外分光光度計在波長600 nm處測定吸光光度值。
干質量的測定:每隔12 h取樣10 mL,離心得菌體沉淀,于70℃烘箱烘干至恒質量后測其干質量。
1.3.9B.clausiiQL-1△spo0A重組菌淀粉酶酶活的測定
分別將出發(fā)菌株B.clausiiQL-1和重組菌株B.clausiiQL-1△spo0A單菌落點涂于可溶性淀粉平板中央,于37℃條件下靜置培養(yǎng)36 h,觀察并比較透明圈大小。分別以2%接種量接種在LB液體培養(yǎng)基中,37℃、200 r/min振蕩培養(yǎng),每隔6 h取樣5 mL,4℃、12 000 r/min離心8 min取上清,按照國標GB/T 24401—2009《α-淀粉酶制劑》中所示方法測定淀粉酶酶活,并按干質量進行換算為U/g。
淀粉酶酶活定義:于60℃,pH為6.0條件下,1 h液化1 g可溶性淀粉的酶量,即為一個酶活力單位(U/g)。
2 結果與分析
2.1 spo0A基因缺失菌株B.clausiiQL-1△spo0A的構建
以B.clausiiQL-1出發(fā)菌基因組為模板,spo0AF和spo0AR為引物進行PCR擴增,1%瓊脂糖凝膠電泳檢測,結果見圖2a。由圖2a可知,擴增產物大小為500 bp左右,與理論長度418 bp相符,說明成功獲得spo0A片段;以質粒pHT01為模板,CmrF和CmrR為引物PCR擴增,1%瓊脂糖凝膠電泳檢測,結果見圖2b。由圖2b可知,擴增產物大小為1000bp左右,與理論長度1 264 bp相符,說明成功獲得抗性基因Cmr片段;以膠回收所得的spo0A片段和Cmr片段為模板,spo0AF和CmrR為引物進行重疊延伸PCR擴增,1%瓊脂糖凝膠電泳檢測,結果見圖2c。由圖2c可知,擴增產物大小為1 500 bp左右,與理論長度1 648 bp相符,說明成功獲得同源重組片段spo0A-Cmr。將spo0A-Cmr片段經(jīng)BamHⅠ酶切后進行濃縮,測其質量濃度為450 ng/μL。將10 μL回收片段與100 μL的B.clausii感受態(tài)混合進行電轉化,涂布于氯霉素抗性LB平板上,挑取陽性轉化子,以spo0AF和CmrR為引物進行菌落PCR驗證,1%瓊脂糖凝膠電泳檢測結果見圖2d。由圖2d可知,出現(xiàn)特異性目的條帶,表明成功獲得B.clausiiQL-1△spo0A重組菌。
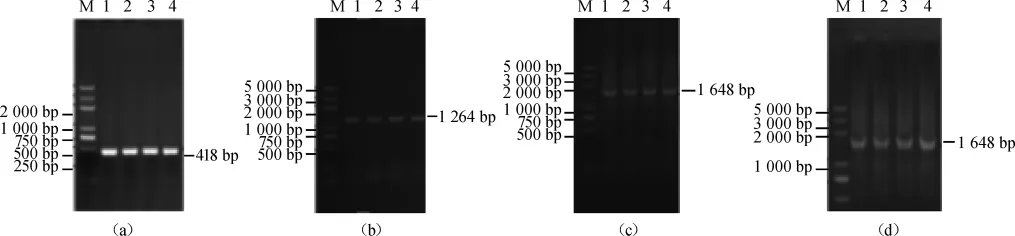
圖2 spo0A-Cmr片段PCR擴增產物及菌落PCR擴增產物凝膠電泳圖Fig.2 Electrophoresis of PCR amplified products of spo0A-cmr and colony
通過連續(xù)傳代15次,以CmrF和CmrR為引物進行菌落PCR擴增,1%瓊脂糖凝膠電泳檢測,結果見圖3。由圖3可知,得到擴增產物大小為1 000 bp左右,與理論長度1 264 bp相符,說明B.clausiiQL-1△spo0A成功導入Cmr抗性基因,以spo0AF和CmrR為引物得到的擴增產物大小同樣于1648bp出現(xiàn)特異性目的條帶,表明重組菌株遺傳穩(wěn)定性較好。
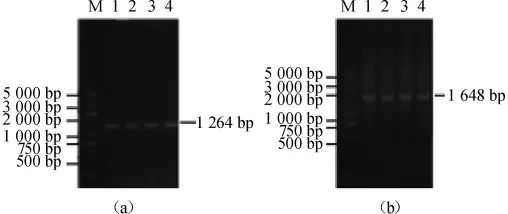
圖3 B.clausii QL-1△spo0A重組菌株PCR擴增產物凝膠電泳圖Fig.3 Electrophoresis of PCR amplified products of B.clausii QL-1△spo0A
2.2 spo0A基因缺失克勞氏芽孢桿菌對芽孢萌發(fā)的影響
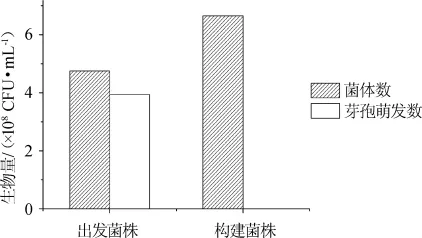
圖4 spo0A基因缺失對克勞氏芽孢桿菌芽孢萌發(fā)的影響Fig.4 Effect of spo0A gene knockout on spore germination of Bacillus clausii
通過平板計數(shù)法對出發(fā)菌株及重組菌株芽孢生成情況進行計算及比較,結果見圖4。由圖4可知,菌株B.clausiiQL-1芽孢萌發(fā)數(shù)為3.93×108CFU/mL,菌體數(shù)為4.67×108CFU/mL,計算得到出發(fā)菌株B.clausiiQL-1芽孢生成率約為84.15%,通過平板計數(shù)法及顯微鏡觀察,B.clausiiQL-1△spo0A均不生成芽孢。結果表明,spo0A基因是克勞氏芽孢桿菌芽孢萌發(fā)過程中的關鍵基因。
2.3 spo0A基因缺失克勞氏芽孢桿菌對菌株生長的影響
對出發(fā)菌株B.clausiiQL-1及重組菌株B.clausiiQL-1△spo0A進行了生長規(guī)律分析及干質量測定,結果見圖5。
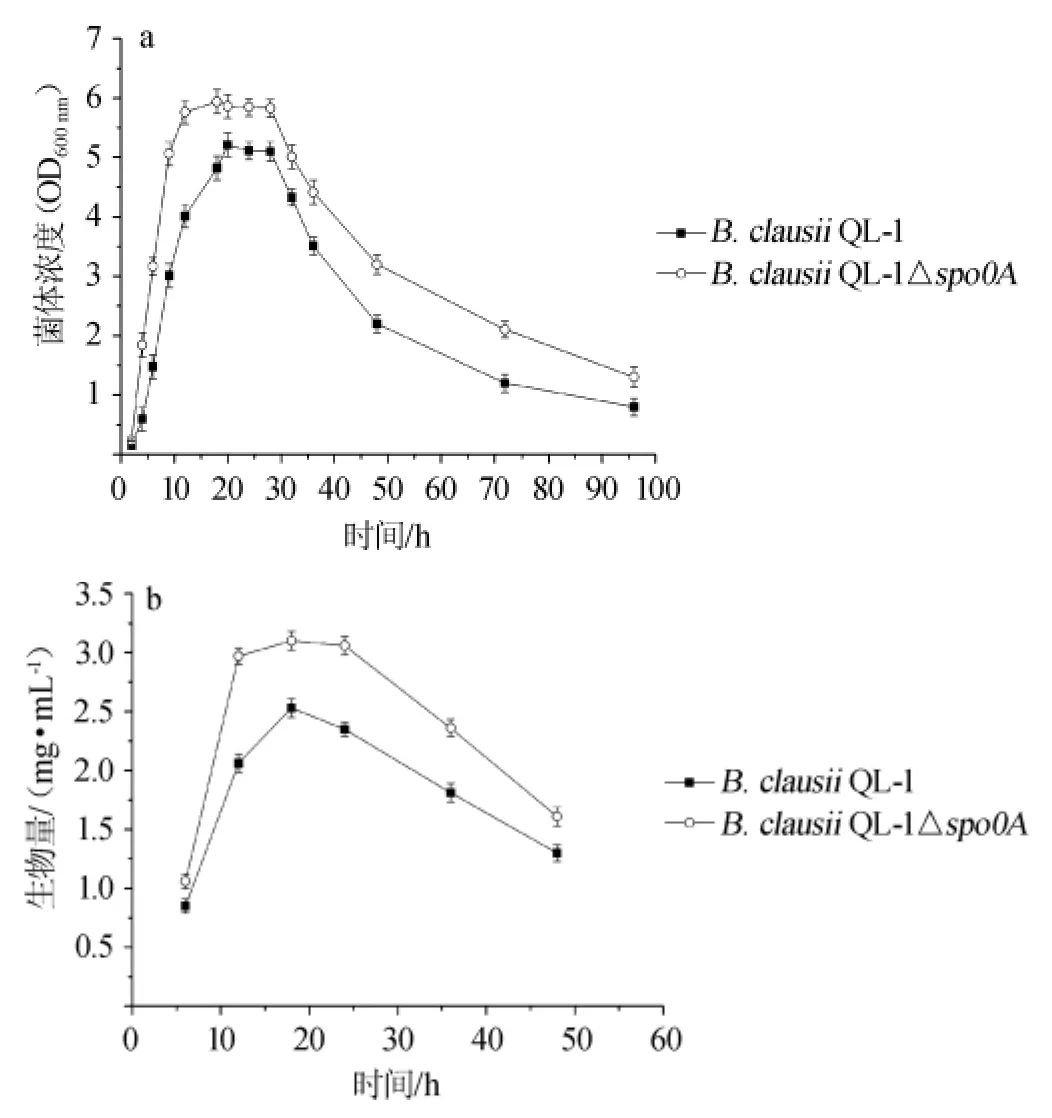
圖5 spo0A基因缺失對菌株在LB培養(yǎng)基中生長周期(a)及干質量(b)的影響Fig.5 Effects of spo0A gene knockout on the growth cycle(a)and dry mass(b)of strain in LB medium
由圖5a可知,B.clausiiQL-1△spo0A菌株與出發(fā)菌株的生長周期基本保持一致,4~18h為對數(shù)生長期,18~28 h為穩(wěn)定期,28 h后進入衰亡期。由圖5b可知,在任何時間段,重組菌株B.clausiiQL-1△spo0A生物量均高于出發(fā)菌株B.clausiiQL-1,且均在20 h出現(xiàn)最大生物量,分別為3.10 mg/mL、2.50 mg/mL,生物量提高了24%。
2.4 spo0A基因缺失克勞氏芽孢桿菌對淀粉酶分泌的影響
可溶性淀粉平板初篩,重組菌株B.clausiiQL-1△spo0A較出發(fā)菌株B.clausiiQL-1透明圈增大,重組菌株透明圈與菌落直徑比為2.75,出發(fā)菌株透明圈與菌落直徑比為1.31,說明構建菌株較出發(fā)菌株水解淀粉能力更強。進一步于LB液體培養(yǎng)基發(fā)酵培養(yǎng)并測定淀粉酶酶活,結果見圖6。由圖6可知,B.clausiiQL-1△spo0A產淀粉能力高于B.clausiiQL-1菌株,且發(fā)酵至72 h時,淀粉酶酶活均達到最高,分別為1.58×105U/g、0.86×105U/g,較出發(fā)菌株B.clausiiQL-1,B.clausiiQL-1△spo0A菌株淀粉酶酶活提高83.72%。
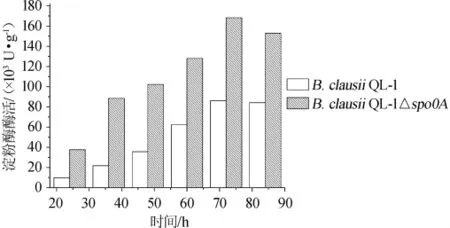
圖6 spo0A基因缺失對淀粉酶酶活的影響Fig.6 Effect of gene knockout on amylase activities
3 結論
本實驗室通過同源單交換技術成功實現(xiàn)了克勞氏芽孢桿菌spo0A基因的敲除,構建了spo0A基因缺失型菌株B.clausiiQL-1△spo0A,并對其發(fā)酵性能進行初步研究,發(fā)現(xiàn)spo0A基因缺失型菌株不生成芽孢,且較出發(fā)菌株B.clausiiQL-1生物量提高24%,表明spo0A基因不僅是克勞氏芽孢桿菌芽孢形成關鍵基因,而且也是菌株生長的重要基因,為構建芽孢缺失型克勞氏芽胞桿菌工業(yè)生產菌株奠定了基礎。通過對B.clausiiQL-1△spo0A菌株的進一步研究,發(fā)現(xiàn)B.clausiiQL-1△spo0A菌株較出發(fā)菌株B.clausiiQL-1淀粉酶酶活提高83.72%,表明spo0A基因也是克勞氏芽孢桿菌淀粉酶生成的重要相關基因,該發(fā)現(xiàn)對高產淀粉酶芽孢桿菌類微生物的構建具有指導意義。
[1]SELLA S R B R,VANDENBERGHE L P S.Life cycle and spore resistance of spore-forming Bacillus atrophaeus[J].Microbiological Research,2014,169(2):931-939.
[2]Al-HINAI M A,JONES S W,PAPOUTSAKIS E T.The clostridium sporulation programs:diversity and preservation of endospore differentiation[J].Microbiology&Molecular Biology Reviews Mmbr,2015,79(1):19-37.
[3]VEENING J W,HAMOEN L W,KUIPERS O P.Phosphatases modulate the bistable sporulation gene expression pattern in Bacillus subtilis[J].Molecular Microbiology,2010,56(6):1481-1494.
[4]HIGGINS D,Dworkin J.Recent progress in Bacillus subtilis sporulation[J].Fems Microbiology Reviews,2015,36(1):131-148.
[5]ERRINGTON J.Regulation of endospore formation in Bacillus subtilis[J].Nature Reviews Microbiology,2003,1(2):117-129.
[6]劉 燕,秦玉昌,潘寶海.枯草芽孢桿菌(Bacillus subtilis)在芽孢形成過程中的幾個關鍵事件[J].生命科學,2005,17(4):360-363.
[7]孫 靜,陳建華.枯草芽孢桿菌形成芽孢時母細胞中基因表達的調控[J].生物技術,2007,17(3):79-83.
[8]CASTILLA V,MUNOZ D,VILLAR L,et al.Spo0A,the key transcriptional regulator for entrance into sporulation,is an inhibitor of DNA replication[J].Embo Journal,2006,25(16):3890-3899.
[9]LEAUX J R,GROSSMAN A D.Isolation and characterization of kinC,a gene that encodes a senor kinase homologous to the sporulation senor kinase KinA and KinB in Bacillus subtilis[J].J Bacteriol,1995,177(6):166-175.
[10]PETTIT L J,BROWNE H P.Functional genomics reveals that Clostridium difficile Spo0A coordinates sporulation,virulence and metabolism[J].BMC Genomics,2014,15(1):160-168.
[11]KATARINA M,ZUZANA C,NIELS B,et al.Morphogenic Protein RodZ Interacts with sporulation Specific SpoIIE in Bacillus subtilis[J].Plos One,2016,11(7):159-167.
[12]GUO M S.Regulation of Bacterial Outer Membrane Homeostasis by the sigma-factor,sigmaE[J].Dissertations&Theses-Gradworks,2014,10(2):147-153.
[13]AHMADPOUR F,YAKHCHALI B.Development of an asporogenic Bacillus cereus strain to improve keratinase production in exponential phase by switching sigmaH on and sigmaF off[J].Fems Microbiology Letters,2017,12(2):259-268.
[14]余志強,楊明明,楊朝霞,等.同源重組法構建枯草桿菌spoA基因缺失突變株[J].武漢大學學報,2004,50(12):229-233.
[15]宋曉蕾,周芬芬,高 瓊,等.spo0A基因在艱難梭菌生長及芽孢形成中的作用[J].中國感染與化療雜志,2017,17(12):33-36.
[16]魏 娟,孫 長.蘇云金芽孢桿菌HD-73菌株基因敲除突變株的構建及特[J].微生物學報,2008,35(10):1581-1586.
[17]SANDOVAL N R,VENKATARAMANAN K P,GROTH T,et al.Whole-genome sequence of an evolved Clostridium pasteurianum strain reveals Spo0A deficiency responsible for increased butanol production and superior growth [J].Biotechnol Biofuels,2015,8(1):227-244.

