應用簡并PCR方法檢測轉cry1A基因作物
李蔥蔥,閆 偉,夏 蔚,董立明,龍麗坤,李飛武*
(吉林省農業科學院農業質量標準與檢測技術研究所,吉林 長春 130033)
盡管轉基因作物在全球已推廣應用超過20 a,且種植面積和產生的經濟社會效益逐年增長,社會公眾對轉基因技術及其產品的安全性仍然存在疑慮[1]。鑒于此,許多國家和地區制定了相應的法律法規,對轉基因作物及其產品實施嚴格的標識監管政策,并設定了相應的標識閾值,歐盟和俄羅斯為0.9%,新西蘭、巴西為1%,日本、印尼、泰國為5%[2]。對某一產品的監管內容主要包括是否含有轉基因成分、特定轉基因成分的含量是多少、是否超過標識閾值等,因此,建立轉基因成分特異、靈敏的檢測方法,是保障安全監管政策法規有效實施的技術支撐。
聚合酶鏈式反應(polymerase chain reaction,PCR)是當前國內外應用最為廣泛的轉基因作物成分檢測技術,許多學者利用常規PCR[3]、多重PCR[4-5]、實時熒光定量PCR[6-7]、數字PCR[8-9]等方法建立了一系列轉基因成分精準檢測方法。檢測靶標包括啟動子、終止子等通用調控元件、外源目的基因、載體構建序列、轉基因作物轉化體特異性序列等[10-14]。轉基因成分檢測的一般流程是先開展針對常見調控元件或外源基因的篩選檢測,若發現含有轉基因成分,再進行轉化體身份確定。
cry1A基因是一類抗蟲基因的總稱,包括cry1Ab、cry1Ac、cry1A.105、mcry1Ac、cry1Ab/Ac等,是抗蟲轉基因作物中使用頻次最高的外源基因,在已商業化的49 個抗蟲轉基因作物轉化體中有32個含有cry1A基因,占65.3%。此外,我國自主研發的抗蟲轉基因玉米、水稻、棉花的靶標基因以cry1A居多,可見,cry1A基因在轉基因檢測工作中可作為一種具有廣泛代表性的靶標。國內外報道了cry1A基因的一系列PCR檢測方法,用于檢測Bt11玉米、Bt176玉米、TT51-1水稻等轉基因作物,其中有些方法已轉化為歐盟標準或我國的行業標準[15-31]。但是,由于轉基因作物研發者往往會對cry1A基因進行序列改造或密碼子修飾,導致不同轉化體中的同一類型的cry1A基因在核苷酸序列上也有較大變異,而上述cry1A基因檢測方法建立時僅在部分轉化體進行了驗證,其是否適用于其他轉cry1A基因作物還不得而知,存在假陰性風險。
本研究擬采用序列比對分析方法,對已報道的cry1A基因PCR檢測方法進行評價,在此基礎上,根據不同轉基因作物中cry1A基因的保守區序列,開發一種可對已知轉cry1A基因作物進行全覆蓋的篩選檢測方法,為此類轉基因作物的監管和檢測提供一種新的技術手段。
1 材料與方法
1.1 材料與試劑
轉cry1Ab基因玉米MON810、Bt11、Bt176、C0030.3.5、雙抗12-5;轉cry1A.105和cry2Ab基因玉米MON89034;轉mcry1Ac基因玉米Bt506;轉cry1Ab/Ac基因水稻TT51-1;轉cry1Ac基因大豆MON87701;轉cry1Ac基因棉花MON531、MON15985;其他轉基因玉米混合樣品(轉cry3Bb基因玉米MON863、轉cry1F基因玉米TC1507、轉cry3A基因玉米MIR604、轉vip3Aa基因玉米MIR162、轉cry34Ab和cry35Ab玉米59122、轉CP4-EPSPS基因玉米NK603);非轉基因玉米樣品;非轉基因大豆水稻混合樣品。以上樣品均由本實驗室收集保存。
植物基因組DNA提取試劑盒(離心柱型) 北京天根生物技術有限公司;HS Taq DNA聚合酶、dNTPs等PCR試劑 大連寶生物工程有限公司;其他生化試劑(均為分析純) 北京鼎國生物科技公司。
1.2 儀器與設備
C1000型梯度PCR儀、GelDoc XR+凝膠成像系統美國Bio-Rad公司;ND1000核酸微量分析儀 美國Thermo Fisher公司。
1.3 方法
1.3.1 基因組DNA提取
將所有樣品研磨成粉末,按照北京天根公司的植物基因組DNA提取試劑盒操作說明書,提取樣品基因組DNA,用核酸微量分析儀測定DNA質量和濃度,用1×TE緩沖液將純化的DNA溶液稀釋至25 mg/L,4 ℃冷藏保存備用。
1.3.2 序列比對
通過查詢網絡數據庫、國內外專利、科技論文等方式,收集不同轉基因作物中的cry1A基因序列及已報道的cry1A基因檢測方法所使用的引物序列,利用Vector NTI Advance 11.5軟件(美國Thermo Fisher公司)進行序列比對分析。
1.3.3 引物設計
以轉基因作物中cry1A基因的相對保守序列為模板,利用Primer premier 5.0軟件進行引物設計。由于cry1A基因的序列差異較大,很難在不同基因序列中找尋到完全一致的引物結合位點,為提高檢測體系的覆蓋度,本研究采用簡并引物策略,篩選出1對cry1A基因PCR檢測引物,cry1A-F:5’-ACCGGYTACACYCCCATCGACATC-3’;cry1A-R:5’-GGCGSWGTTCATGTCGTTGAA-3’,擴增產物大小為330 bp。引物由上海生工有限公司合成,用1×TE緩沖液稀釋至10 μmol/L的工作液。
1.3.4 簡并PCR
優化后的PCR體系包括:10×PCR buffer 2.5 μL、10 mmol/L dNTPs混合溶液2 μL、10 μmol/L上下游引物各0.5 μL、HS Taq DNA聚合酶1 U、DNA模板100 ng,用ddH2O補齊至25 μL。優化后的PCR擴增程序為:94 ℃預變性5 min;94 ℃變性30 s,60 ℃退火30 s,72 ℃延伸30 s,進行35 個循環;72 ℃延伸7 min;4 ℃保存。PCR擴增結束后,取10 μL擴增產物用2%的瓊脂糖凝膠進行電泳分析。
2 結果與分析
2.1 序列比對分析
根據轉基因作物商業化應用數據庫(http://www.isaaa.org/gmapprovaldatabase)的統計,有32 個已商業化種植的轉化體使用cry1A基因作為目的基因,還有一批轉化體處于研發過程中。本研究共收集到轉基因玉米MON810、C0030.3.5、Bt176、SK12-5、Bt11、MON89034、Bt506、轉基因水稻TT51-1、轉基因大豆MON87701、轉基因棉花MON15985、MON531共11 種轉cry1A基因作物材料,并獲得了對應的cry1A基因序列信息,見表1。

表1 轉基因作物中cry1A基因基本信息Table1 Information about cry1A genes used in transgenic crops
對獲得的cry1A基因序列進行比對分析,發現部分基因序列間的同源性非常高,如:Bt176和SK12-5的cry1Ab基因僅有2 個堿基差異,MON810和C0030.3.5的cry1Ab基因僅有1 個堿基差異,MON531和MON15985的cry1Ac基因序列完全一致,且與MON87701僅1 個堿基差異,但11 個cry1A基因整體的同源性較差,僅為59.3%。從5’端向3’端逐段掃描未發現連續15 個以上的一致性堿基,表明采用特定引物序列同時檢測這些cry1A基因是很難實現的,需采取簡并引物策略。
2.2 已報道的cry1A基因檢測方法評價
從轉基因生物檢測方法數據庫(http://gmdd.sjtu.edu.cn)中檢索到cry1A基因的21 種不同檢測方法,標注為M1~M20,其中普通PCR方法15 種、實時熒光PCR方法6 種,這些方法中有5 種已轉化為行業標準或國家標準,每種方法使用的引物及其他信息見表2。將這些方法的引物與上述cry1A基因序列進行比對,結果顯示沒有1 種方法的引物序列能夠同時與11 個cry1A基因序列完全配對,每對引物的錯配堿基數均大于3 個,且許多錯配發生在引物的3’端。此外,部分方法的引物序列在有的基因上沒有結合位點,如:M3、M7和M12方法的上游引物在TT51-1水稻的cry1Ab/Ac基因無結合位點,M15方法的上游引物在MON89034玉米的cry1A.105基因無結合位點,M18和M20方法的上下游引物僅能與cry1Ac基因匹配,而與cry1Ab和cry1A.105不能匹配。上述序列比對分析結果表明,文獻報道的這些cry1A基因檢測方法理論上僅適用于特定靶標基因,不能用于表1中所列11種cry1A基因的高效篩查。

表2 已報道的cry1A基因PCR檢測方法Table2 Reported PCR primers for detecting cry1A genes
2.3 cry1A基因簡并PCR引物設計
盡管11 個cry1A基因整體的同源性不足60%,但5’端序列表現出較好的同源性,選取了5’端1~600 位堿基序列作為模板,應用Primer Premier V5.0軟件篩選到1 對適用于PCR擴增的簡并引物,上、下游引物各有2 個簡并堿基,見圖1的下劃線部分,擴增產物大小為330 bp。

圖1 cry1A基因序列比對結果及引物結合位點Fig.1 Alignment of nucleotide sequences and primer-binding sites for cry1A genes
2.4 簡并PCR體系的特異性分析
以1.1節所列的轉基因作物及非轉基因作物為檢測對象,測試cry1A基因簡并PCR體系的特異性。擴增結果表明,僅從含有cry1A基因的MON810、MON89034、Bt11、Bt176、Bt506、C0030.3.5、SK12-5、TT51-1、MON87701、MON531、MON15985樣品中獲得預期330 bp的特異性PCR產物,而在轉其他基因玉米及非轉基因作物中均未擴增出該產物(圖2),表明建立的cry1A基因簡并PCR方法能夠特異性地用于11 種cry1A基因作物的篩選檢測,可對目前已知的cry1Ab、cry1Ac、cry1Ab/Ac、cry1A.105和mcry1Ac等cry1A類基因進行高效篩查。
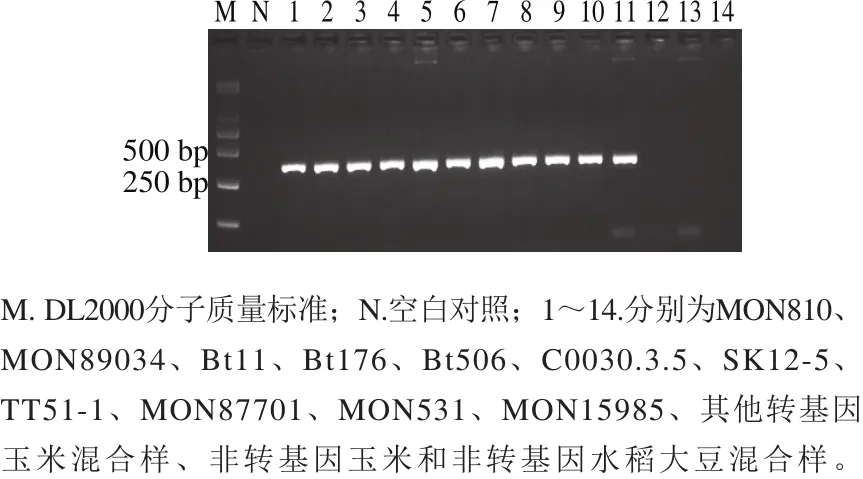
圖2 cry1A基因PCR方法的特異性測試結果Fig.2 Specif i city of the PCR assay
2.5 簡并PCR體系的靈敏度分析
選取轉cry1Ab基因玉米MON810為檢測對象,將MON810玉米粉與非轉基因玉米粉按質量比混合配制成MON810質量分數分別為1%、0.5%、0.1%、0.05%、0.01%、0.005%的梯度樣品,提取基因組DNA,進行cry1A基因的PCR擴增。結果顯示,利用本方法可從MON810玉米質量分數為0.01%以上(含0.01%)的樣品中穩定擴增出預期DNA片段,而在0.005%的樣品中未得到擴增,表明本方法對MON810玉米的檢測靈敏度可達到0.01%(圖3)。

圖3 cry1A基因PCR方法的靈敏度測試結果Fig.3 Sensitivity of the PCR assay
在使用普通PCR進行轉基因成分定性檢測時,一般要求方法的檢測限應穩定達到0.1%。根據國際公認的檢測限判定程序,隨機稱取60 份MON810質量分數為0.1%的玉米樣品,提取基因組DNA,用cry1A基因簡并PCR方法進行擴增。如圖4所示,60 份試樣中均能穩定擴增出與預期大小一致的PCR產物,表明本方法的檢測限可達0.1%,適用于對cry1A基因的高靈敏檢測。
為進一步測試本方法對其他轉cry1A基因作物的檢測靈敏度,以非轉基因玉米、大豆、水稻等量混合物為填充物,制備了9 份測試樣品,用cry1A基因簡并PCR方法進行擴增。如圖5所示,無論是在cry1A基因含量為0.1%的Bt11、MON89034、TT51、MON531、MON87701單一轉基因成分樣品,還是在Bt11/MON87701、TT51/MON531、Bt11/MON531、MON89034/TT51混合樣品中,均能獲得與預期大小一致的特異性擴增產物,表明本方法對不同組分的轉cry1A基因作物均能進行很好地篩查。

圖4 cry1A基因PCR方法的檢測限測試結果Fig.4 Determination of the limit of detection (LOD) of the PCR assay

圖5 cry1A基因PCR方法的檢測限驗證結果Fig.5 Validation of the LOD of the PCR assay
2.6 簡并PCR體系的穩定性測試

圖6 cry1A基因PCR方法退火溫度的穩定性測試Fig.6 Stability of the PCR assay at different annealing temperatures
為評價退火溫度和引物用量波動對方法穩定性的影響,選取MON810、MON89034、Bt11等9 個樣品,對5 種退火溫度和3 種引物終濃度進行測試。結果顯示,當引物終濃度設定為0.2 μmol/L,退火溫度分別為56、58、60、62 ℃和64 ℃時,均能從轉cry1A基因作物中擴增出預期DNA片段,而在陰性對照和空白對照中未獲得該擴增產物(圖6)。當退火溫度設定為60 ℃,引物終濃度分別為0.1、0.2 μmol/L和0.3 μmol/L時,同樣能夠獲得與預期一致的結果(圖7)。表明本方法在退火溫度和引物用量發生一定程度的波動時,也能保持很好的穩定性。

圖7 cry1A基因PCR方法引物用量的穩定性測試Fig.7 Stability of the PCR assay at different dosages of primers
3 結 論
cry1A基因是當前商業化應用的抗蟲轉基因作物中最常見的外源目的基因,而且,從近年來全國轉基因作物監測的情況來看,轉cry1A基因作物的擴散風險較高,是監管的重中之重。因此,建立具有廣泛適用性的cry1A基因定性PCR檢測方法,對于完善我國轉基因作物檢測手段具有十分重要的意義。
由于不同作物的密碼子偏好性差異,科學家們在作物遺傳改良時常常會對目的基因的核苷酸序列進行修改,導致不同轉基因作物中的同一種cry1A基因序列存在較大差異,給檢測方法研究帶來一些挑戰,有時需針對一種靶標基因建立多種方法以確保覆蓋度,增加了檢測工作量。應用簡并PCR策略能在一定程度上解決這一問題,若多個基因序列的某一段區域呈現較高的一致性時,可利用1 對簡并引物實現對多個靶標基因的篩選檢測。
本研究在系統比較分析11 種轉基因作物中的cry1A基因序列基礎上,篩選出具有通用性的cry1A基因簡并引物,并經過特異性、靈敏度、檢測限、穩定性測試,建立cry1A基因簡并PCR方法。該方法可對11 種常見轉cry1A基因作物進行全覆蓋檢測,方法的靈敏度穩定達到0.1%,為轉基因作物的篩選檢測提供了一種高效的技術手段。

