羊水游離RNA中神經發育關鍵基因分析*
楊樹法,劉 妍,秦 朗,閆有圣,陰赪宏
首都醫科大學附屬北京婦產醫院/北京婦幼保健院,北京 100026
Rett綜合征、胼胝體發育異常、孤獨癥以及多動癥等神經發育障礙是常見的先天出生缺陷,嚴重影響患兒及其家庭生活質量,給社會帶來了沉重的經濟負擔[1-4]。產前診斷是預防神經發育障礙出生缺陷的主要手段。隨著分子生物技術的發展,熒光原位雜交技術、熒光定量PCR、芯片檢測以及二代測序等技術陸續應用于產前診斷,更多類型的染色體微缺失/重復、點突變等基因組變異信息被檢出。基因組變異與臨床表型間的復雜聯系,使得高通量檢測報告的臨床解讀面臨巨大挑戰,以美國醫學遺傳學和基因組學學會為代表的多種專業委員會建立了多種遺傳變異的評價系統[5-6],將突變分為致病性、可疑致病性、致病性未明、可疑良性以及良性等。這些評價系統多以兒童和成人的臨床資料為基礎發展而來。與出生后可以廣泛獲取患兒臨床資料相比,在產前診斷中可獲取的胎兒臨床信息極其有限;B超作為最大的獲取源,可獲取的胎兒資料遠遠少于成人;另外,實驗室檢查和物理診斷資料基本缺失。這些都導致了在產前診斷中基因組變異的臨床解讀更具有挑戰性。產前診斷中,存在1%~2%的致病性未明突變[7],因此,獲取更多胎兒發育信息,協助判定胎兒發育情況,成為產前診斷中亟待解決的問題。羊水是產前診斷中最易獲取的胎兒附屬物,先前研究表明,羊水游離RNA(AfcfRNA)包含與胎兒多種組織發育相關的基因表達信息[8-10]。但是,AfcfRNA中基因來源于胎兒多個組織,加之神經系統發育涉及多個過程,這都加大了從AfcfRNA中獲取神經系統發育信息的難度。因此,本研究分析了孕中期正常AfcfRNA轉錄組,結合基因共表達網絡分析和組織特異性基因分析,試圖從AfcfRNA轉錄組中提取神經系統發育相關的關鍵基因,為AfcfRNA轉錄組在產前診斷中應用奠定基礎。
1 資料與方法
1.1一般資料 以孕中期正常胎兒的羊水為研究對象,從Gene Expression Omnibus數據庫中下載正常胎兒AfcfRNA的芯片檢測結果。納入標準:(1)核型分析結果正常;(2)單胎妊娠;(3)羊水采集時間為孕中期(孕13~27周);(4)檢測平臺為Affymetrix Human Genome U133 Plus 2.0芯片。排除標準:(1)染色體核型結果異常;(2)胎兒B超檢查結果異常。按照納入標準和排除標準分別從GSE101141[11]、GSE16176[12]、GSE25634[13]、GSE33168[14]、GSE47394[15]、GSE48521[16]、GSE49893[17]及GSE58435[18]中獲取56例孕中期AfcfRNA轉錄組的檢測結果。
1.2方法
1.2.1神經組織特異性基因的定義 正常組織的基因表達量數據來源于Human Protein Atlas數據庫[19],下載基因表達數據表(https://www.proteinatlas.org/download/rna_tissue_consensus.tsv.zip)到本地。統計該數據庫所包括的組織和基因種類,將基因在神經組織中的表達量高于該基因在所有組織中表達量均值的10倍以上者,定義為神經組織特異性基因。
1.2.2共表達網絡分析 使用Oligo v1.54.1軟件包讀取芯片原始結果,并對結果進行背景校正、歸一化,獲取基因表達量數據[20]。Oligo是用于分析寡核苷酸芯片的軟件包,可用于處理Affymetrix和NimbleGen芯片結果。批次效應[21]的去除借助SVA包,SVA包通過確定和建立代理變量去除高通量數據中的批間差異和其他無關變異。利用加權基因共表達網絡分析(WGCNA) 軟件包[22]進行基因共表達網絡分析,軟閾值設為12,最小類的數量設為25。篩選得到的基因共表達模塊用顏色表示。
1.2.3基因功能富集分析 編寫R腳本從共表達模塊中選取神經組織特異性基因,建立神經組織特異的共表達模塊,利用ClusterProfiler軟件包分別對每個神經組織特異共表達模塊中的基因進行GO(gene ontology)功能富集分析[23],ClusterProfiler軟件包可以對基因和基因集進行多種功能富集分析(GO、KEGG及GSEA等)。用Benjamini-Hochberg方法對P值進行調整,以調整的P值(Padj)<0.05為差異有統計學意義。
1.2.4蛋白共表達網絡分析 共表達模塊中的關鍵基因的篩選,以從STRING數據庫(https://stringdb-static.org/download/protein.links.v11.0/9606.protein.links.v11.0.txt.gz)下載的蛋白間相互作用數據為基礎[24],編寫R語言腳本,建立基因間相互作用關系,將結合力設為900,統計每個基因的連接度。連接度為基因間相互作用的表示形式,連接度越高表示基因與其他基因存在更多的相互作用關系,其所處的位置越關鍵,研究中將連接度大于15(即與15個以上的基因存在蛋白間相互作用)的基因作為關鍵基因。
1.3數據分析軟件環境 數據的分析和處理借助R語言完成。基因不同命名系統間的轉換使用HGU133Plus2.db軟件包,HGU133Plus2.db是對Affymetrix Human Genome U133 Plus 2.0 Array進行注釋的軟件包,包含了探針和不同基因命名系統間的對應關系。蛋白間相互作用數據的制圖使用Igraph軟件包,Igraph軟件具有對網絡進行分析和可視化的功能。其他數據繪圖借用Ggplot2軟件包,Ggplot2是基于圖形繪制語法而設計的用于將數據可視化的軟件。變異系數(CV)=正常AfcfRNA基因表達量的標準差/正常AfcfRNA基因表達量的均值×100%。
2 結 果
2.1正常AfcfRNA中基因共表達模塊建立 研究中共使用56例孕中期AfcfRNA芯片檢測結果,芯片檢測結果經背景校正、歸一化和去除批次效應后,不同樣本間具有相似的均值和標準差,樣本間具有可比性(圖1A)。利用WGCNA建立檢測基因的共表達關系,利用動態樹形剪切算法共建立27個共表達模塊,模塊名稱用顏色表示(藍綠色、藍色、棕色、黃色、綠色、紅色、黑色、洋紅色、粉紅色、紫色、鮭肉色、棕褐色、黃綠色、午夜藍色、淡青色、青綠色、灰色60、淺綠色、淺黃色、寶藍色、深粉藍色、深灰色、暗紅色、深綠色、橘黃色、深橙色、白色),各共表達模塊中基因的數量如圖1B所示。不具有共表達關系的基因分類到灰色模塊中。
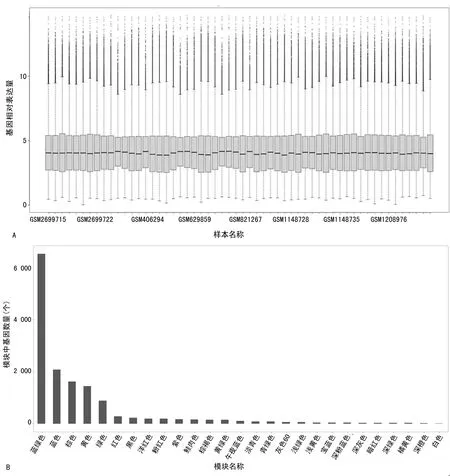
注:A表示56例AfcfRNA芯片檢測結果背景校正、歸一化和批次校正結果;B表示各共表達模塊中基因數量。圖1 正常AfcfRNA基因共表達模塊的建立
2.2神經組織特異性基因共表達模塊的功能富集分析 Human Protein Atlas數據庫中神經系統組織包括大腦皮質、胼胝體、海馬結構、下丘腦、杏仁核、中腦、嗅覺區、垂體、腦橋和延髓、脊髓、基底神經節、黑質、丘腦以及小腦等組織。利用表達量高于整體表達量10倍的標準,共篩選得到832個神經組織特異性基因。分別將27個共表達模塊中的基因與832個神經組織特異性基因取交集,獲取神經組織特異基因的共表達模塊。在獲取神經組織特異性共表達模塊的基礎上,分別對共表達模塊中的基因進行GO分析。在藍色、棕色、藍綠色以及黃色模塊中集中富集到神經功能相關的GO術語(GO term),見圖2。這些術語涉及前腦發育、神經突觸組裝和功能、神經遞質釋放過程、軸突發生以及學習和記憶過程等神經系統功能的多個方面。

注:A、B、C、D分別為藍色、棕色、藍綠色以及黃色模塊中神經組織特異性共表達模塊內基因的GO分析結果;橫軸為富集到術語的模塊內基因的數量,條圖顏色深淺表示Padj大小(Padj<0.05為差異有統計學意義),縱軸為富集到的GO術語。圖2 神經組織特異性共表達模塊功能富集分析
2.3神經組織特異性共表達模塊的關鍵基因 將藍色、棕色、藍綠色以及黃色共表達模塊內的神經組織特異性基因利用STRING數據庫,分析他們間的相互作用關系,尋找各個模塊中具有高連接度的關鍵基因。共篩選到27個關鍵基因,分析芯片中各基因表達量均值,計算得到27個基因表達量的P25為3.09,CV的P50為14.09%。將基因表達量均值低于27個基因表達量P25的關鍵基因刪除,共發現17個關鍵基因,藍色模塊中3個(SLC18A3、TACR3、SYT2),棕色模塊中6個(SSTR5、STX1A、SNAP25、GHSR、SSTR4、GABBR2),藍綠色模塊中5個(DRD2、SLC32A1、GNG3、OPN4、PENK),黃色模塊中3個(RAB3A、HCRT、GRM5)。關鍵基因的模塊來源、連接度、基因平均表達量及CV見表1。

表1 關鍵基因信息統計表

續表1 關鍵基因信息統計表
3 討 論
羊水是產前診斷中最安全且最易獲取的胎兒附屬物,先前研究表明羊水中含有來源于胎兒多種組織的AfcfRNA,這些基因的變化與胎兒發育密切相關[8-9],通過分析AfcfRNA中基因變化為監測胎兒發育提供了可能。但是,AfcfRNA中基因來源和各系統發育過程的復雜性,極大地增加了從AfcfRNA中獲取各系統發育信息的難度。與單個基因的變化相比,基因集的變化更具穩定性,能夠降低單基因變化的噪聲污染;同時組織和器官的發育涉及多種基因的相互和共同作用。WGCNA基于多個樣品表達數據,通過計算基因間的相關性,利用剪切算法將具有相同表達模式的基因歸為同一表達模塊。本課題組利用WGCNA,將AfcfRNA中的基因分為27個具有共表達關系的模塊(圖1B),不具有共表達關系的基因被過濾到灰色模塊中。
Human Protein Atlas數據庫中包含了已知基因在正常人體大部分組織的表達量數據。在建立基因間共表達關系模塊的基礎上,利用Human Protein Atlas數據庫篩選并建立了神經組織特異的基因共表達網絡[19]。這些神經組織特異的共表達模塊內的基因參與神經系統主要的生物學過程(神經遞質傳遞、神經組織發生、學習和認知等)。這些結果表明,研究中建立的基因共表達網絡與神經系統功能密切相關(圖2)。
STRING數據庫中存儲了蛋白-蛋白間相互作用的數據,利用蛋白-蛋白間相互作用數據可以構建篩查到的基因的相互作用網絡。與其他基因具有更多聯系的基因是共表達網絡的關鍵基因,是該網絡功能的集中體現。研究中利用STRING[24]數據庫對WGCNA建立的網絡進行了篩選,選取了每個網絡中的關鍵基因。在藍色模塊中得到3個關鍵基因(SLC18A3、TACR3、SYT2),通過基因富集分析發現,該模塊中基因主要與突觸后膜電位、神經遞質傳導以及突觸組裝的突觸功能有關(圖2A)。前人研究表明,SLC18A3囊泡乙酰膽堿通道,其缺陷可以導致先天性肌無力綜合征[25];TACR3編碼速激肽受體3,廣泛表達于神經系統,并參與情緒障礙、疼痛、學習和記憶缺陷、神經系統發育等神經生理和病理過程[26];SYT2編碼突觸結合蛋白,SYT2的缺陷與突觸前先天性肌無力綜合征有關[27],并且在髓母細胞瘤的發生發展中起關鍵作用[28]。
棕色模塊有6個關鍵基因(SSTR5、STX1A、SNAP25、GHSR、SSTR4、GABBR2)。其中,SSTR5和SSTR4是生長激素抑素受體,廣泛分布在大腦、下丘腦、外周神經系統、胰腺等多種組織[29],是實體瘤潛在的藥物作用靶點[30];STX1A編碼突觸結合蛋白1A,與兒童多動癥[31]有關,JNK2與STX1A間相互作用參與N-甲基-D-天門冬氨酸誘發的谷氨酸釋放[32];SNAP25是漢族人孤獨癥的候選基因[33],參與神經信息傳遞[34];GHSR是生長激素促分泌素受體,其甲基化狀態與腫瘤的發生密切相關[35];GABBR2是γ-氨基丁酸受體2,參與多種神經遞質傳遞過程。這些基因廣泛參與了神經信號傳導的多個過程。
在藍綠色模塊中得到5個關鍵基因:DRD2、SLC32A1、GNG3、OPN4、PENK。DRD2是多巴胺受體,其多態性與精神疾病和藥物依賴密切相關[36-37];SLC32A1是囊泡γ-氨基丁酸轉運體成員,其可能參與了γ-氨基丁酸和乙酰膽堿囊泡釋放[38];GNG3是多形性成膠質細胞瘤發生和發展過程中的關鍵基因[39-40];OPN4是黑素蛋白基因,其功能與睡眠和清醒有關[41];PENK是腦啡肽原,其水平降低與亨廷頓氏舞蹈病的癥狀的嚴重程度密切相關[42]。
在黃色模塊中得到3個關鍵基因:RAB3A、HCRT、GRM5。RAB3A是Ras樣GTP酶[43],參與激素釋放、神經遞質釋放以及細胞膜循環等多個過程[44];HCRT為下丘腦神經肽前體;GRM5為谷氨酸受體。這些基因與機體的學習和記憶功能密切相關。
胎兒發育是多個基因協同表達的結果,AfcfRNA中基因的變化是胎兒發育情況的綜合表現。胎兒發育異常會導致AfcfRNA中的基因表達量變化,AfcfRNA中存在能夠檢測胎兒發育情況的標志物。本課題組認為潛在標志物應具備如下3個特點:(1)組織特異性表達基因。通過選擇特異性基因可以在一定程度上排除其他組織發育對基因表達變化的影響。(2)穩定表達基因。對于芯片檢測,該穩定性表現為較高的表達量和較低的CV;本研究中, 基因平均表達量的P25為3.09,CV的P50為14.09%,筆者使用了3.09和14.09%分別作為二者的臨界值,但這是存在爭議的,需要更多的實驗證實。(3)起關鍵作用的基因。這些基因能夠同多個基因協同作用,在基因調控和組織發育中起到關鍵作用,組織的異常發育也經常與關鍵基因的變化密切相關。基于上面的考慮筆者設計了關鍵基因的挖掘方法:基因組織特異性分析、基因表達量和變異分析以及共表達網絡和關鍵基因分析。同時要說明的是,研究中使用的56例正常羊水標本是指核型分析和B超檢查正常的標本,絕大部分胎兒為發育正常的胎兒,基于這些標本篩選得到的基因通過文獻檢索證實與神經系統功能和神經發育異常密切相關,但能否作為檢測神經發育異常的標志物尚需要進一步實驗驗證,這些基因表達的變化可能與這些基因突變有關,也可能由于其他基因的表達異常導致。
綜上所述,本研究通過對孕中期AfcfRNA轉錄組進行共表達網絡分析和組織特異性分析,獲得了神經系統發育密切相關的并且具有共表達關系的關鍵基因。這些關鍵基因來源的共表達網絡模塊與神經系統功能密切相關,其異常與神經系統相關疾病密切相關,可作為潛在的產前診斷中監測神經系統發育異常的標志物。

