基于InDel標記的谷子株高QTL定位
杜曉芬 錢枰勵 唐楚楚 杜德杰 韓康妮 李禹欣 王智蘭 王 軍,*
(1山西農業大學谷子研究所/山西省后稷實驗室,山西 長治 046011;2山西農業大學農學院,山西 太谷 030801;3中國農業大學農學院,北京 100193)
谷子(Setariaitalica)具有耗水少、抗旱耐瘠、營養豐富均衡、糧飼兼用等特點,是旱作生態農業綠色發展的主栽作物[1]。隨著谷子參考基因組的公布[2-3],谷子基礎研究在近十年飛速發展,并逐漸發展為禾本科作物功能基因組研究的模式作物[4]。
分子標記是作物遺傳研究的基礎,其中,插入/缺失(insertion/deletion,InDel)多態性標記是指等位基因的核苷酸序列在個體間因插入或缺失引起的長度變異,具有分布廣、密度高、變異穩定、重復性好、性價比高等優點[5],廣泛應用于種質資源分析、遺傳多樣性分析、親緣關系鑒定、種質鑒定、遺傳圖譜構建、重要性狀QTL 定位與圖位克隆等方面。趙慶英等[6]對名優谷子品種晉谷21 號進行重測序,與參考基因組相比發現了169 037 個InDel 位點,進一步驗證了68 個InDel 位點,并開發了一個晉谷21 號的特異InDel 標記,為分子標記輔助育種提供了分子標記資源。Zhang 等[7]基于谷子品種SSR41 的重測序結果,開發了156 個適合瓊脂糖凝膠電泳的InDel 標記,并利用多態性InDel 標記對谷子白葉鞘基因進行了圖位克隆。王智蘭等[8]根據SiGRAS23開發了一個與株高性狀緊密連鎖的InDel 標記D8-1,可用于谷子種質資源株高變異的篩選。張碩等[9]利用7 個InDel 標記將谷子白條紋葉基因定位于20.13 Mb 內,為該基因的精細定位和克隆奠定了基礎。由此可見,InDel標記在谷子基礎研究方面發揮了重要的作用。
株高是谷子的重要農藝性狀之一,主要通過調節株型和抗倒伏性影響谷子產量。前人遺傳分析結果表明,谷子株高是典型的數量性狀,由多基因控制[10]。近十年來,研究人員定位了許多株高相關的數量性狀位點(quantitative trait loci,QTL)[11-16],這些QTL 在谷子的9條染色體上均有分布。同時,前人精細定位和克隆了一些候選基因,例如編碼DELLA蛋白的SiD1和編碼細胞色素P450 蛋白的SiD2[17-18]。盡管如此,相比水稻、小麥等主要農作物,谷子株高QTL 或基因的鑒定仍相對較少,這嚴重制約了谷子株高遺傳機理研究的進展。
鑒于此,本研究對2 個株高存在極顯著差異的谷子品種(衡谷12號和長農35號)進行深度重測序,比較兩者之間的InDel差異,開發InDel標記,并利用多態性InDel標記掃描“衡谷12號×長農35號” F2分離群體,構建遺傳連鎖圖譜、定位株高QTL,進一步在重組自交系群體中驗證株高QTL,并對候選基因進行預測。本研究旨在為谷子基礎研究提供一套好用的InDel標記,同時為株高基因的克隆奠定基礎。
1 材料與方法
1.1 試驗材料及性狀調查
本研究中,用于株高QTL 定位的雜交組合的母本為衡谷12 號,由河北農林科學院旱作農業研究所選育,具有矮稈、極早熟、分蘗性強等特點[19],父本長農35 號由山西農業大學谷子研究所選育,屬于高稈、晚熟、無分蘗品種[20]。2016年,將衡谷12號和長農35號進行雜交;2017年,鑒定F1植株;2018年,將F1自交獲得F2分離群體,在谷子成熟期調查株高,其中,雙親調查5株,取平均值作為性狀值,F2子代逐株掛牌調查株高。
采用單粒傳法,將F2分離群體繁殖到F6代。2022年,將雙親和重組自交系群體(recombinant inbred line,RIL,F6代共283個株系)種植2行,行長2 m,株距0.33 m。在谷子成熟期調查株高,每個株系調查5株,以平均值作為性狀值。所有材料均種植于山西農業大學谷子研究所試驗田,田間管理、水肥等條件一致。
1.2 DNA提取
在谷子拔節期,分別采集新出葉片,液氮速凍后置于-80 ℃備用。采用改良的十二烷基硫酸鈉(sodium dodecyl sulfate,SDS)法提取總DNA。SDS提取液配制:1 mol·L-1Tris-HCl(pH 值8.5)100 mL,0.5 mol·L-1乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA)(pH 值8.0)100 mL,5 mol·L-1NaCl 20 mL,20% SDS 100 mL,加去離子水定容至1 L。提取步驟為:將葉片放入研缽中加入液氮研磨成粉狀,轉移至2.0 mL 離心管中;加入SDS 提取液750 μL,混勻后置于65 ℃水浴鍋中1 h;加入750 μL苯酚/氯仿(1∶1),混勻15 min后,4 ℃,12 000 r·min-1離心15 min;吸取上清液600 μL,加等體積氯仿,混勻15 min 后,4 ℃,12 000 r·min-1離心15 min;吸取上清液450 μL,加0.6 倍體積的異丙醇混勻,靜置30 min;4 ℃,12 000 r·min-1離心10 min 后棄上清,用70%乙醇清洗2 次;室溫干燥后加入100 μL 的Tris-EDTA(TE)緩沖液備用。
1.3 RNA提取和cDNA合成
分別在谷子孕穗期和開花期,對衡谷12 號和長農35 號的倒二莖進行取樣,液氮速凍研磨,參照RNAiso Plus 試劑(9108,北京寶日醫生物技術有限公司)說明書提取總RNA,參照PrimeScript Ⅱ 1st strand cDNA synthesis kit 試劑盒(6210A,北京寶日醫生物技術有限公司)說明書合成cDNA。
1.4 重測序
在苗期,分別隨機采集衡谷12號和長農35號的葉片,采用高效植物基因組DNA 提取試劑盒(DP350,北京天根生化科技有限公司)提取DNA,送至北京百邁客生物科技有限公司進行重測序,重測序按照Illumina公司(美國)提供的標準步驟執行,簡要流程為:對提取的基因組DNA 進行質檢,DNA 檢測合格后用超聲波將DNA片段化;對片段化的DNA進行末端修復、3'端加A和連接測序接頭;用瓊脂糖凝膠電泳對片段大小進行選擇,并進行PCR 擴增形成測序文庫;對測序文庫進行文庫質檢,質檢合格的文庫用Hiseq 2500 測序儀(Illumina公司,美國)進行測序。
1.5 基因組變異檢測
測序得到的原始序列(raw reads)進行過濾,獲得高質量的序列(clean reads),過濾的標準為:去除帶接頭的reads;過濾N 含量超過10%的reads;去除質量值低于10、堿基超過50%的reads。利用Burrows-Wheeler Transform 軟件[21]將獲得的clean reads 比對到谷子參考基因組豫谷1號上(Setariaitalicav2.2,https://phytozome.jgi.doe.gov/pz/portal.html#%21info?alias=Org_Sitalica_er)。使用Picard(https://sourceforge.net/projects/picard/)去掉重復,Genome Analysis ToolKit 軟件[22]用于檢測單核苷酸多態性(single nucleotide polymorphisms,SNP)和InDel,并用SnpEff 軟件[23]對檢測到的SNP 和InDel 進行注釋,Breakdancer(http://breakdancer.sourceforge.net/)用于檢測結構變異(structral variation,SV),對于檢測到的SV,通過比對變異位點在參考基因組上的位置對其進行注釋。
1.6 InDel標記開發和多態性鑒定
比較衡谷12號和長農35號差異InDel位點,選擇衡谷12號和長農35號插入或缺失堿基數量差異大于等于3 bp且間隔大于5 kb的位點作為引物設計的目標位點。分別提取目標位點上下游側翼序列150 bp,整理成Fasta格式,利用BatchPrimer3 v2.0(https://probes.pw.usda.gov/batchprimer3/)批量設計引物,產物大小設置為100~300 bp,引物由南京金斯瑞生物科技股份有限公司合成。
PCR 反應體系為10 μL:10×PCR buffer 1.0 μL,2 μmol·L-1上下游引物各1 μL,rTap DNA聚合酶0.1 μL,2.5 mmol·L-1dNTP 0.8 μL,模板DNA 1.0 μL,滅菌水5.1 μL。PCR 擴增程序為:94 ℃預變性5 min,94 ℃變性30 s,58 ℃退火30 s,72 ℃延伸30 s,35個循環;72 ℃終延伸10 min。PCR 擴增產物利用8%聚丙烯酰胺凝膠電泳進行分離,經硝酸銀染色后,拍照記錄。
1.7 遺傳圖譜構建和QTL定位
選擇能夠良好分型的多態性InDel標記,分別掃描F2群體(120 個單株)和RIL 群體(283 個株系),對擴增結果進行統計,其中與衡谷12號帶型一致的記為“A”,與長農35 號帶型一致的記為“B”,雜合帶型記為“H”,條帶缺失時記為“-”。采用JoinMap 4[24]構建遺傳連鎖圖譜,選擇回歸算法(regression mapping)和Kosamb,s函數,其他參數為默認參數。采用WinQTLCart 2.5[25]對株高進行QTL 定位,使用復合區間作圖(composite interval mapping,CIM)法,在P<0.05 條件下,進行1 000 次排列測驗(permutation test)確定QTL 的似然比的自然對數(likelihood of odd,LOD)閾值。QTL命名規則按照q+性狀簡稱+染色體進行命名,當同一條染色體出現多個QTL,按照-1、-2、-3的順序命名。
1.8 候選基因預測和分析
利用Phytozome數據庫(https://phytozome-next.jgi.doe.gov/),以豫谷1 號為參考基因組,對定位區間的基因及其功能注釋進行分析。同時根據衡谷12 號和長農35 號重測序數據,分析定位區間內編碼區發生突變的基因,預測候選基因。對預測的候選基因進行克隆測序,采用DNAMAN 7 軟件對測序結果進行比對分析。參考王智蘭等[8]的方法進行候選基因的熒光實時定量表達分析。
1.9 數據統計分析
利用SPSS 17 分析群體性狀的最大值、最小值、平均數、標準差、峰度、偏度以及進行獨立樣本t檢驗。利用GraphPad Prism 5進行頻率分布分析和作圖。
2 結果與分析
2.1 衡谷12號和長農35號重測序及變異分析
衡谷12 號和長農35 號的clean data 為31.82 Gbp,Q30 達到88.38%,兩個樣品與參考基因組平均比對率為98.63%,平均覆蓋深度為34×,其中衡谷12 號的覆蓋深度為30×,長農35 號的覆蓋深度為38×。變異檢測結果表明,衡谷12 號中單核苷酸多態性(SNP)、插入/缺失(InDel)和結構變異(SV)數量分別為524 486、103 442 和22 295 個,長農35 號中SNP、InDel 和SV 數量分別為1 131 571、196 477 和24 509 個(圖1-A),比較而言,檢測到的SNP 數量最多,SV 數量最少,且長農35 號中這三種變異類型都高于衡谷12 號,同時發現,長農35 號和衡谷12 號在相同位點共同檢測到SNP 變異位點和InDel 變異位點分別有259 259 和55 224 個(圖1-E、F)。
SNP 變異類型分為堿基轉換(T/A 和C/G)和堿基顛換(A/C、G/T、C/T 和G/A),衡谷12 號中堿基顛換的數量占總變異類型的87.7%,長農35 號中堿基顛換的數量占總變異類型的88%,同時發現,長農35號中的堿基轉換和堿基顛換的數量均高于衡谷12號(圖1-B)。
SV 變異類型分為大片段插入(insertion,INS)、大片段缺失(deletion,DEL)、反轉(inversion,INV)、染色體內易位(intra-chromosomal translocation,ITX)和染色體間易位(inter-chromosomal translocation,CTX)等,衡谷12 號和長農35 號的變異類型主要是INS 和DEL,分別占總變異類型的84.76%和79.17%(圖1-C)。
InDel變異類型分為小片段插入(INS)和小片段缺失(DEL),分析表明,在衡谷12號和長農35號中,主要發生均為1 bp的INS或DEL,同時發現大于等于39 bp且小于等于50 bp的INS數量高于DEL數量(圖1-D)。
2.2 InDel標記的開發和多態性分析
比對衡谷12 號和長農35 號中InDel 變異位點,選擇兩者差異大于等于3 bp 且間隔大于5 kb 的位點,最后篩選了3 961 個位點,并設計引物。首先,篩選在雙親間具有多態性的標記,初步獲得1 392 對多態性標記,多態性率為35.14%,其中第3 染色體的多態性標記最多,第4 染色體的多態性標記最少(圖2-A)。然后,用F2群體的部分單株進一步篩選,獲得591 個穩定性好,且在后代群體中能夠良好分型的多態性標記(圖2-B),為下一步遺傳圖譜構建奠定了基礎。
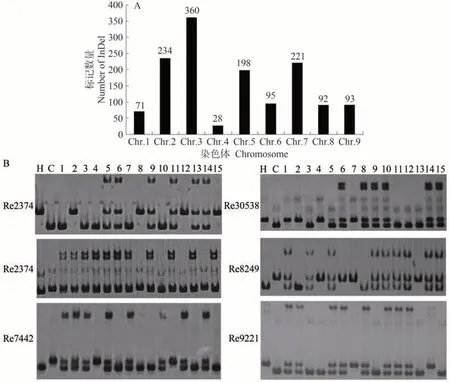
圖2 InDel標記的多態性分析Fig.2 Development and polymorphism analysis of InDel markers
2.3 遺傳圖譜構建及F2群體株高QTL定位
利用591個InDel標記對包含120個單株的F2群體進行基因分型,采用JoinMap 4 進行連鎖分析,最后獲得一張包括467 個InDel 標記的谷子遺傳連鎖圖譜(電子附圖1),該圖譜總圖距448.45 cM,平均圖距0.96 cM,其中第4 和第8 染色體標記數量最少(16 個),第3 染色體標記數量最多(160 個),第1 染色體圖距最短(9.67 cM),第9染色體圖距最長(105.22 cM)(表1)。
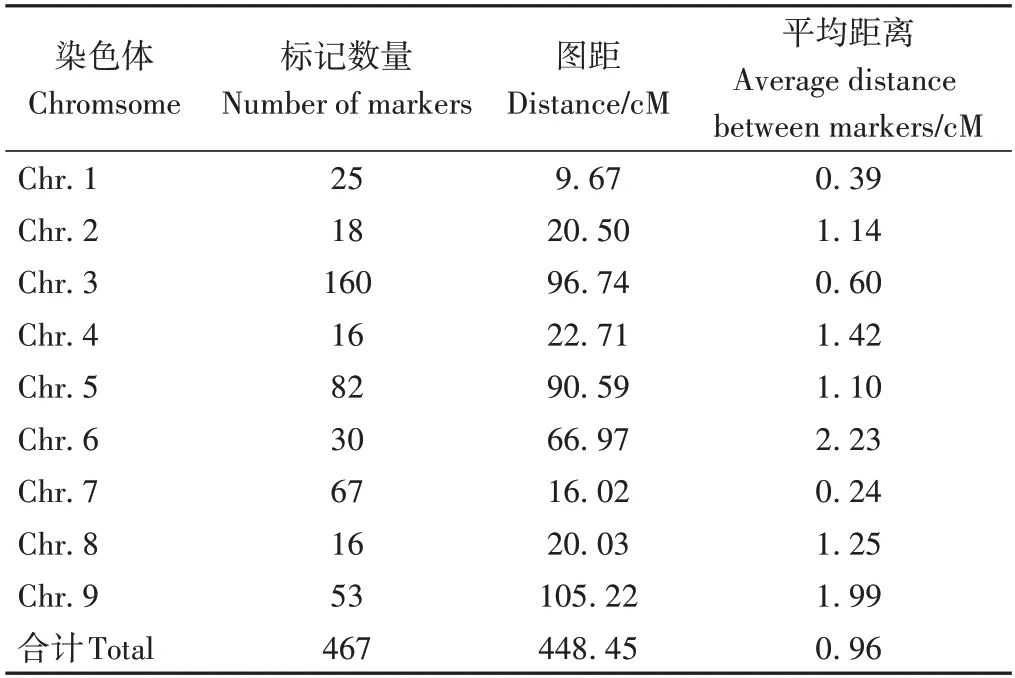
表1 “衡谷12號×長農35號” F2群體遺傳連鎖圖譜信息Table 1 Characteristics of the genetic map in F2 population of ‘Henggu12 × Changnong35’
對雙親及120 個F2子代株高進行分析,發現雙親之間差異極顯著(P<0.01,圖3-A),株高在F2群體中呈雙向超親分離(表2),頻率分布結果顯示,株高呈雙峰分布(圖3-A)。
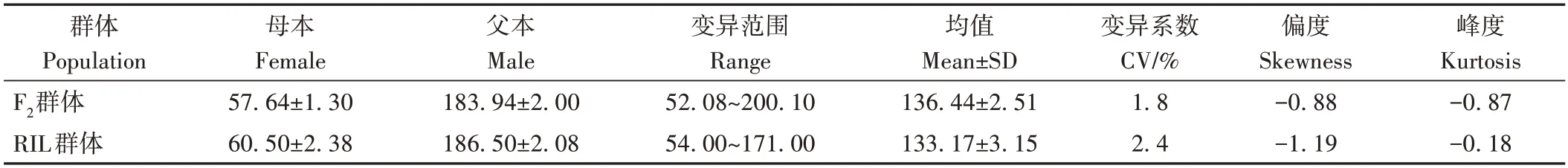
表2 雙親及群體株高統計分析Table 2 Phenotypic analysis of plant height in the parents,F2 population and RIL population /cm
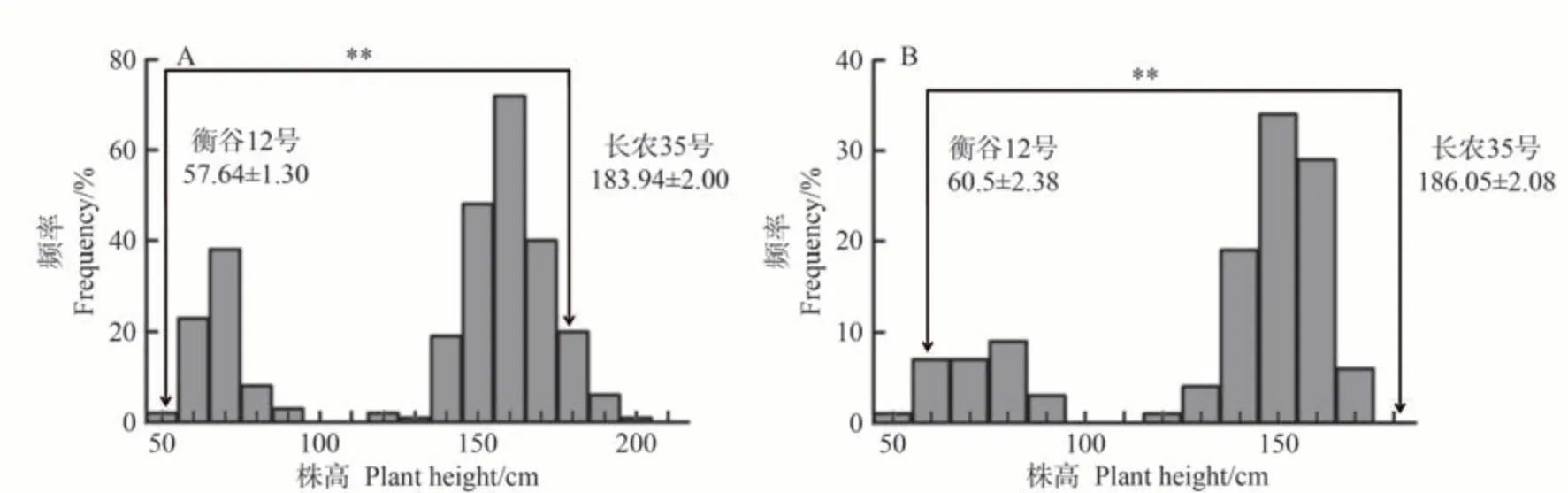
圖3 “衡谷12號×長農35號” F2群體和RIL群體株高頻率分布直方圖Fig.3 Histogram of frequency distribution of plant height in F2 population and RIL population of ‘Henggu 12×Changnong 35’
利用WinQTLCart 2.5 對F2群體的株高進行QTL定位,共檢測到4個QTL 位點,除qPH5-1外,加性效應均為負值,說明來自父本長農35 號的等位位點對株高貢獻率大。在4 個QTL 中,第5 染色體檢測到2 個QTL,分別為qPH5-1和qPH5-2,其中qPH5-1可解釋表型變異率最大,為8.41%;第9 染色體檢測到2 個QTL,分別為qPH9-1和qPH9-2,其中qPH9-2可解釋表型變異率為16.47%,為主效QTL(表3)。
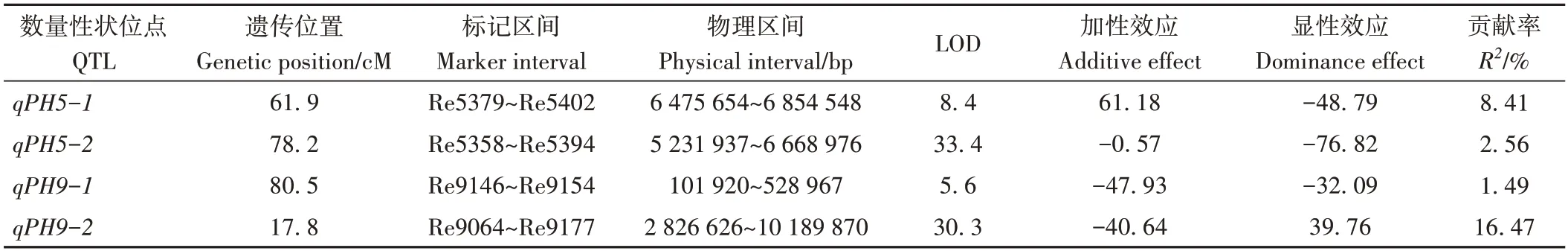
表3 “衡谷12號×長農35號” F2群體株高QTL定位Table 3 Putative QTL detected for plant height in F2 population of ‘Henggu12×Changnong35’
2.4 RIL群體株高QTL的驗證
首先,對雙親和283 個株系的株高進行分析,發現株高在RIL 群體中呈單向超親分離,變異范圍比F2群體小,但是變異系數比F2群體大(表2)。頻率分布結果顯示,與F2群體的結果相似,株高呈雙峰分布(圖3-B)。然后在RIL 群體中對F2群體中鑒定的效應較大的QTL(qPH5-1和qPH9-2)進行了驗證。結果表明,在RIL 群體中,僅檢測到qPH9-2,LOD 值為93.6,加性效應為-46.15,解釋表型貢獻率達91.06%(表4)。

表4 “衡谷12號×長農35號”重組自交系群體株高QTL定位Table 4 Putative QTL detected for plant height in RIL population of ‘Henggu12 × Changnong35’
2.5 qPH9-2候選基因分析
利用Phytozome 數據庫分析qPH9-2定位區間(2 776 961~7 257 031 bp),該區間包含706 個基因,分析衡谷12號和長農35號在該區間的重測序結果,發現70個基因在編碼區、5'UTR和3'UTR發生突變。其中,27 個基因由于SNP 變異發生非同義突變,4 個基因由于InDel 變異發生移碼突變(電子附表1)。分析發生非同義突變的基因功能注釋,發現編碼bHLH 轉錄因子的基因Seita.9G080400,在其5'UTR 處和編碼區分別存在2 處SNP。由于轉錄因子在植物生長發育中發揮重要作用,因此對其進行了重點分析。
根據參考基因組Seita.9G080400的基因組序列,設計特異引物(電子附表2),分別對衡谷12 號和長農35 號進行克隆測序,測序結果驗證了其5'UTR 的2 處SNP(SNP1和SNP2)和第一外顯子的2處SNP(SNP3和SNP4),同時還發現在其第一內含子、第二內含子和第三內含子分別存在1 處SNP(SNP5、SNP6 和SNP7)(電子附圖2)。外顯子中的SNP3在衡谷12中編碼纈氨酸(V),在長農35號中編碼丙氨酸(A);外顯子中的SNP4在衡谷12 中編碼組氨酸(H),在長農35 號中編碼天冬氨酸(D)(圖4-A)。表達分析結果表明,孕穗期莖中,Seita.9G080400相對表達量在衡谷12號中是長農35號的10倍;開花期莖中,Seita.9G080400相對表達量在衡谷12號中是長農35號的42倍(圖4-B)。
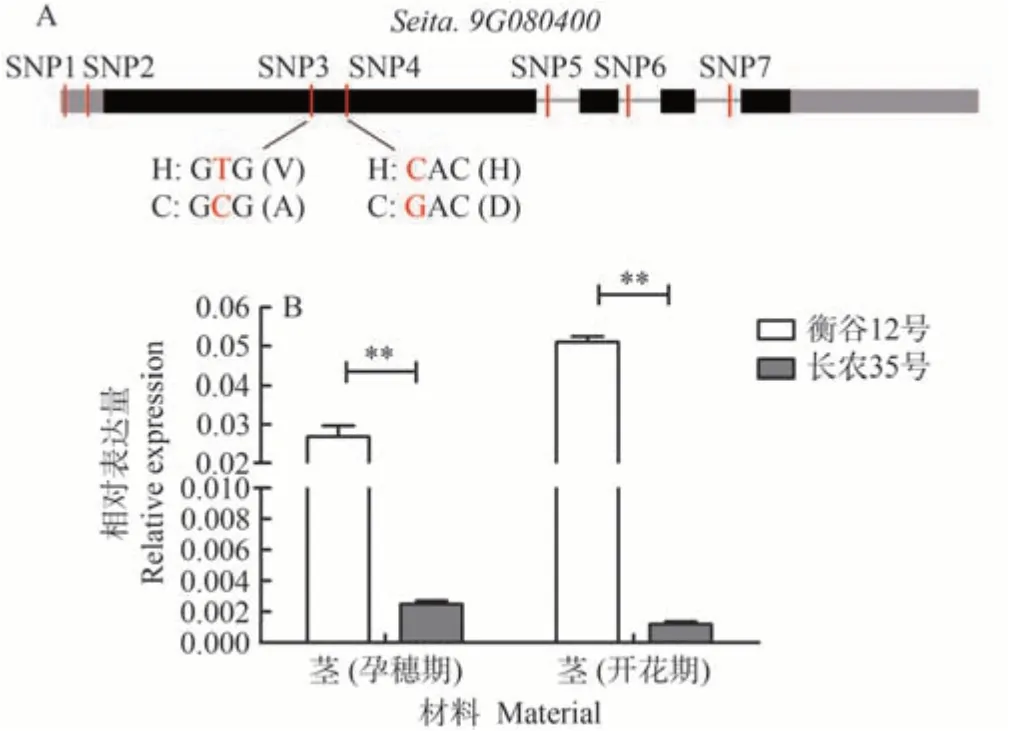
圖4 Seita.9G080400序列分析和表達分析Fig.4 Sequences and expression level analysis of Seita.9G080400 between Henggu12 and Changnong35
3 討論
隨著高通量測序技術的快速發展,基于重測序技術,在全基因組水平發掘的分子標記在作物中得到廣泛應用。大量研究表明,InDel標記是植物中的第二大類多態性高的標記,具有準確性高、共顯性、易于基因分型等諸多優點[5],已在遺傳圖譜構建、重要性狀QTL定位、種質資源分析與鑒定等方面廣泛應用。谷子中相關文獻證實,InDel標記數量僅次于SNP標記[6-7,26-28]。例如,Li等[27]對312份谷子種質進行重測序,共鑒定了3 027 706 個SNP 和256 826 個InDel,Bai 等[28]在農家種十里香中檢測到915 434 個SNP 和28 546 個InDel。本研究中,衡谷12 號中的SNP 和InDel 分別為524 486和103 442個,長農35號中的SNP和InDel分別為1 131 571和196 477 個。由于測序深度和材料的不同,導致檢測到的SNP 和InDel 數量有所不同。本研究進一步驗證了InDel 標記是一類具有豐富多態性的標記,此外,本研究鑒定的大量InDel位點為開發標記提供了分子基礎。
目前已有研究中,主要利用簡單重復序列(simple sequence repeat,SSR)標記[29-31]進行種質資源群體結構分析[32]、圖位克隆[33]、優異等位變異檢測[34],而InDel標記應用的相關報道較少。盡管谷子中InDel變異豐富,但對其進行開發并驗證的標記數量同樣相對較少,谷子中可用的InDel 標記資源有限,因此在全基因組水平開發InDel 標記非常必要。趙慶英等[6]在晉谷21 中開發驗證了68 個InDel 標記,Zhang 等[7]在SSR41中開發驗證了156個InDel標記。本研究選擇了3 961 個在衡谷12 號和長農35 號之間有差異的InDel位點,經過初步篩選,獲得了1 392 個多態性InDel 標記,多態性率為35.14%。與前人研究結果相比,本研究驗證的InDel標記數量較多,并能夠有效應用于株高QTL定位。
數量性狀QTL 定位結果易受定位群體和環境條件影響,盡管初級定位群體(F2、F2:3和DH)表型數據不能重復,但是一些大效應的QTL 還是會被準確定位。例如,水稻“綠色”基因DEP1的克隆,最初利用DH 群體檢測到控制水稻直立穗型的主效QTL(qPE9-1,貢獻率為41.72%)[35],隨后利用2 個F2群體及1 521 個F2子代將其精細定位并確定候選基因DEP1[36]。本研究利用開發的多態性InDel 標記,構建了“衡谷12 號×長農35 號” F2群體的遺傳連鎖圖譜,初步定位了4 個株高QTL,并對其中2 個效應值較大的QTL 在RIL 群體中進行了驗證,相比F2群體的定位結果,qPH9-2在RIL 群體中能重復檢測到,貢獻率達91.06%,且物理區間(2 776 961~7 257 031 bp)更小。前人在谷子第9染色體上相同區間也檢測到株高QTL(H9a:3 452 395~9 964 754 bp),貢獻率為7.6%~15.5%[12],暗示該區間存在控制株高的主效QTL。
bHLH 是植物中重要的一類轉錄因子,參與植物生長發育與形態建成。研究表明,小麥TabHLH123調控小麥根系發育,TabHLH123-6B與根重、千粒重和株高相關[37],TabHLH123-6A與根系構型相關[38]。本研究中,Seita.9G080400編碼bHLH 轉錄因子,測序結果表明,在衡谷12 號和長農35 號中,其第一外顯子有2處SNP,導致氨基酸發生非同義突變。其中,SNP3在衡谷12 號中編碼纈氨酸,長農35 號中編碼丙氨酸,這兩種氨基酸都為疏水性氨基酸;SNP4 在衡谷12 號中編碼組氨酸,長農35 號中編碼天冬氨酸,由堿性氨基酸變為酸性氨基酸,這些氨基酸的改變是否影響蛋白的功能,需要進一步驗證。另外,在不同發育時期的莖中,Seita.9G080400相對表達量在衡谷12 號和長農35號之間存在極顯著差異。綜合以上結果,推測Seita.9G080400可能是控制株高的關鍵候選基因,但是仍然需要通過進一步精細定位和功能鑒定進行驗證。
4 結論
本研究利用2 個谷子品種的重測序數據,在全基因組水平開發了1 392 個分布于谷子9 條染色體的InDel 標記。利用多態性InDel 標記,構建了一張包含467 個InDel 標記的谷子遺傳連鎖圖譜,定位了1 個控制株高的主效QTL,預測了1 個關鍵候選基因,驗證了InDel標記的有效性和應用性,為今后谷子種質資源鑒定、群體結構分析和親緣關系分析等提供了良好的分子標記資源。
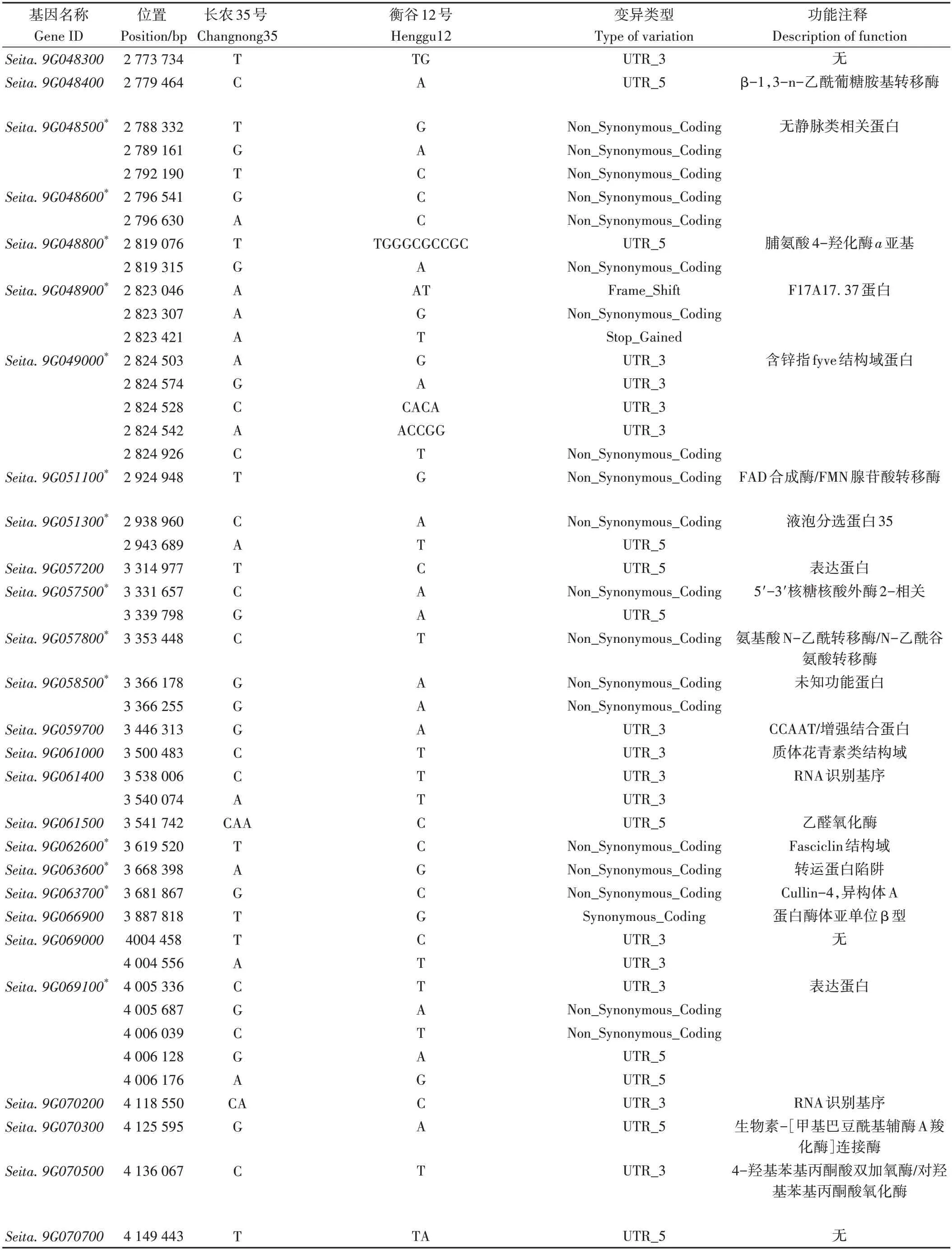
電子附表1 突變基因的信息Electronic Table S1 Information of mutant genes
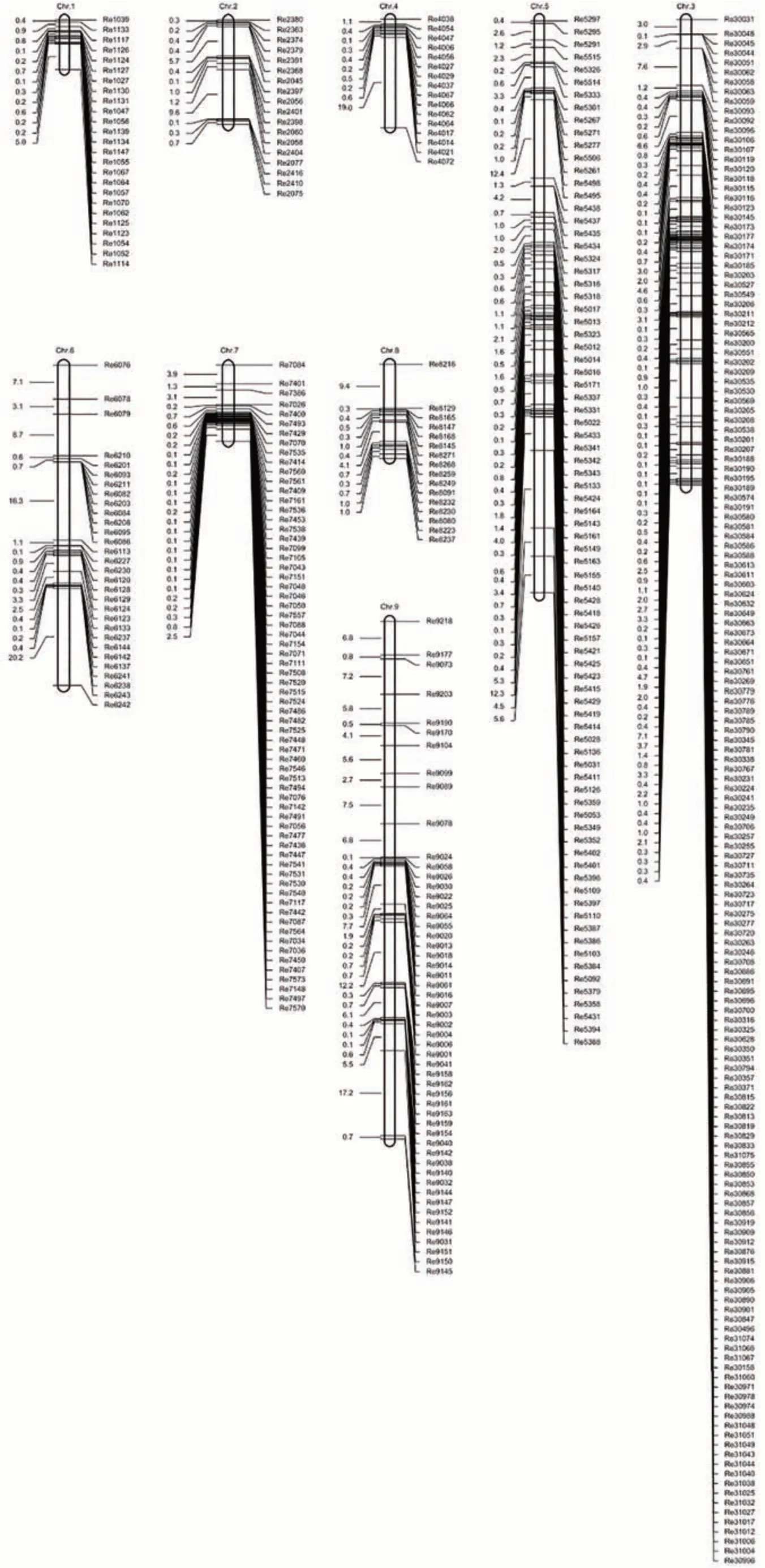
電子附圖1 “衡谷12號×長農35號”F2群體InDel標記連鎖遺傳圖Electronic Fig.S1 The genetic linkage map constructed using 467 InDel markers in F2 population derived from ‘Henggu12 ×Changnong35’

