哈爾濱產乙醇桿菌屬AFLP反應體系的建立與優化
鄭國香,陳忠林,鄭文玲,關正軍,吳憶寧,任南琪
(1.哈爾濱工業大學市政環境工程學院,150090哈爾濱,rnq@hit.edu.cn;2.東北農業大學工程學院,150030哈爾濱)
擴增片段長度多態性(Amplified Fragment Length Polymorphism,AFLP)是結合 RFLP和RAPD優點的基礎上發展起來的一項技術.基于其可靠性和PCR技術的高效性,且具有快速、靈敏、穩定、所需DNA量少、多態性檢出率高、重復性好、可以在不知道基因組序列特點的情況下進行研究等特點,被認為是一種十分理想的、有效的、先進的分子標記方法,現已廣泛用于遺傳圖譜構建[1-2]、遺傳多樣性研究[3-5]、基因定位及品質鑒定[6-7]等方面.利用AFLP分子標記的方法探討產氫突變菌株和野生菌株的基因組DNA之間的遺傳多態性差異,將為更深入地研究產氫突變菌株的突變機理提供重要的信息基礎.針對哈爾濱產乙醇菌桿菌屬的分子標記研究還未見有報道,本研究以發酵產氫細菌作為研究對象,建立和優化適合于哈爾濱產乙醇桿菌屬基因組AFLP分析的技術體系,為進行發酵產氫細菌的遺傳代謝機理的深入研究提供重要的技術支撐和信息基礎.
1 實驗
1.1 實驗菌株和培養條件
本實驗所用的出發菌株分離于CSTR生物制氫反應器的活性污泥,為乙醇型厭氧發酵產氫細菌,哈爾濱產乙醇桿菌Ethanoligenens harbinense ZGX4,以其為出發菌株經過誘變篩選獲得突變株Ethanoligenen harbinenseYR-3,以上菌株由哈爾濱工業大學市政環境生物技術中心提供.
1.2 微生物的保存、激活及厭氧培養條件
[8]方法進行.
1.3 DNA的提取
將1 mL已經預先活化的菌液接入10 mL的RZL-2培養基中,37°C進行厭氧過夜培養,然后將2 mL的接種液轉接入裝有50 mL RZL-2培養基的培養瓶中,在37°C培養24~28 h.菌液的DNA提取利用華舜細菌DNA提取試劑盒,提取的DNA用0.8%瓊脂糖電泳進行檢測,并用蛋白質核酸分析儀(Beckman Coultor DU800)檢測DNA的質量和純度.
1.4 AFLP指紋分析體系的優化
1.4.1 酶切與連接
1)酶切:37℃下,采用EcoRI和MseI兩種酶對細菌DNA的全基因組進行酶切.
2)純化酶切樣品:將1/10體積的醋酸鈉和2.5體積的冰醋酸加入酶切樣品,-20℃條件下保存2 h;然后在4℃,20 000 r/min離心15 min,棄上清液,加70%乙醇溶解沉淀,20 000 r/min離心15 min,棄上清液,干燥,30 μL TE液溶解,置于-20°C下保存.
3)接頭的連接:利用T4 DNA酶連接DNA酶切片段和接頭.(酶切+接頭)混合樣品分別稀釋10、20和30倍,做預擴增的模板.
1.4.2 預擴增反應
94℃變性30 s,56℃退火30 s,72℃延伸80 s.取4 μL樣在0.8%的瓊脂糖凝膠電泳檢測,樣品稀釋10、20和30倍,做選擇性擴增的模板.
1.4.3 選擇性擴增反應
采用選擇性引物對進行特異性選擇擴增.程序:30個循環,94℃30 s,65℃→55℃30 s,72℃80 s退火溫度每個循環降0.3℃.取PCR樣品3 μL進行瓊脂糖電泳檢測.
2 結果與討論
擴增片段長度多態性分子標記(AFLP)具有在一次實驗中可同時觀測大量限制性片段的優點,是目前最有效的分子標記技術.AFLP反應程序中模板DNA質量及酶切片段擴增反應的條件是否恰當都將影響最終結果的分析.因此,利用產氫野生菌株和突變菌株作為研究對象,探討和優化AFLP程序中的試驗條件將為增加產氫發酵細菌的遺傳多態性和高效性提供重要的實驗基礎.
2.1 模板DNA的質量和純度
AFLP對樣本的DNA純度要求較苛刻,基因組DNA的純度直接影響AFLP后續的實驗結果.使用細菌DNA抽提試劑盒對產氫野生菌株ZGX4和突變菌株YR-3進行了DNA提取.利用紫外分光法測定DNA的質量和純度,結果表明,DNA質量在50~100 ng時,當 OD260/OD280小于1.8時,DNA樣品中存在蛋白質污染現象,見圖1的泳道3和4,同時出現明顯的彌散現象,即DNA降解跡象.而OD260/OD280大于1.9的DNA樣品,存在明顯的RNA污染和DNA降解脫尾現象,如圖1中的泳道5和6所示;選用OD260/OD280在1.8~1.9之間的DNA樣本,經瓊脂糖電泳檢測結果(圖1的泳道1和2)顯示,產氫菌株的DNA帶型整齊,無RNA污染和DNA降解,完全符合AFLP試驗要求,進行PCR擴增可以獲得穩定、清晰的帶譜,表明DNA質量符合AFLP后續的試驗操作要求.
2.2 基因組DNA的酶切和連接
通常情況下,選用單一限制性內切酶對DNA進行酶切,產生的DNA片段較少,體現出來的DNA片段多態性效果較差[4].AFLP所使用的限制性內切酶一般采用兩種,一種是內切酶的識別位點是6個堿基,例如EcoRI、PstI和SacI;另一種是4堿基內切酶,如MseI、TaqI和SseI.選用兩種酶同時酶切可以產生比較小的酶切片段,經過PCR反應擴增出的產物范圍可在100~2 000 bp之間.本實驗選用EcoRI/MseI作為產氫細菌YR-3和ZGX4基因組DNA的限制性內切酶.
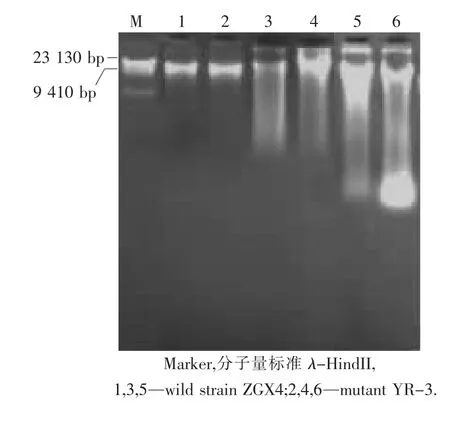
圖1 產氫細菌基因組DNA瓊脂糖電泳圖
AFLP片段的多態性及豐富性是由酶切的多態性和豐富性決定的,因此,酶切質量是決定AFLP成功與否的關鍵前提條件之一.酶用量過大,首先是造成浪費,MseI酶是稀有酶,價格昂貴,用量過多還會在相同時間出現酶切過頭的現象.用量過少,所需要的時間過長,而且常常會導致酶切不完全.因此,準確把握酶的用量及酶切時間十分重要.
2.2.1 酶切用量
大量實驗已經表明,AFLP的兩種限制性內切酶的總量不能超過反應體積的1/10[9],本實驗針對發酵產氫細菌基因組DNA材料,采用EcoRI (20 U/μL)/MseI(10 U/μL)做雙酶切,設定其用量組合分別為 1,EcoRI(0.5μL)/MseI (0.2 μL);2,EcoRI(1.0 μL)/MseI(0.4 μL); 3,EcoRI(1.5 μL)/MseI(0.6 μL);4,EcoRI (2.0 μL)/MseI(0.7 μL);5,EcoRI(2.5 μL)/MseI (1 μL);6,EcoRI(3.0 μL)/MseI(1.5 μL),DNA質量為60 ng,時間為3 h.利用瓊脂糖凝膠電泳進行檢測,具體的酶切用量對產氫細菌基因組DNA產生的酶切效果見圖2所示.
試驗結果表明,利用EcoRI(0.5 μL)/MseI (0.2 μL)對哈爾濱產乙醇發酵細菌的DNA進行酶切之后效果很差,如圖2的泳道1,DNA酶切片段多分布在800~2 000 bp,基本沒有切開;利用EcoRI (1.0 μL)/MseI(0.4 μL)和EcoRI(1.5 μL)/MseI (0.6 μL)組合的酶切效果表明,DNA酶切片段分布的范圍在500~2 000 bp,如圖2的泳道2和3所示;而通過對比發現,利用 EcoRI(2.0 μL)/MseI (0.7 μL)和EcoRI(2.5 μL)/MseI(1 μL)兩個酶切組合的試驗結果顯示,在100~2 000 bp都有DNA酶切片段出現,見圖2的泳道4和5,酶切用量都比較理想,鑒于經濟性考慮,在相同酶切效果的前提下,選擇EcoRI(2.0 μL)/MseI(0.7 μL)為哈爾濱產乙醇桿菌屬的最佳酶切用量;當繼續增加酶切用量,即EcoRI(3.0 μL)/MseI(1.5 μL)時,如圖2的泳道6所示,出現了小于100 bp的DNA酶切片段.可見,限制性內切酶用量太少,導致酶切不完全,基因組多態性不豐富.酶用量過多,既造成了浪費,又容易出現酶切過頭現象,產生低于100 bp的DNA片段,導致接頭接不上,PCR反應失敗[10].

圖2 產氫細菌基因組DNA不同酶切用量的酶切效果
2.2.2 酶切時間
在確定最佳酶切組合和酶切用量后,選擇最恰當的酶切時間也是影響酶切成敗的必要條件.如果酶切時間過短,酶切不完全,酶切片段不能覆蓋整個基因組,影響PCR擴增的多態性[11].從圖3可以看出,酶切3 h的效果比較理想(泳道3),酶切片段在100~2 000 bp之間都有分布.而泳道1和2的酶切時間相對較短,分別為1 h和2 h,DNA酶切不充分.泳道4(4 h)和5(5 h)顯示酶切時間過長,出現100 bp以下的片段,酶切過頭將使接頭連接不上,導致PCR反應無結果.因此,通過酶切時間的試驗確定利用EcoRI(2.0 μL)/MseI(0.7 μL)酶切哈爾濱產乙醇桿菌屬的最佳時間為3 h.將MseI和EcoRI接頭配制成終質量濃度為100 μmol/L,T4 DNA酶連接DNA酶切片段和接頭,連接時間過夜效果最好.
2.3 預擴增反應條件的選擇
AFLP反應程序中另一重要的環節就是酶切連接產物的預擴增反應,該反應起著呈上啟下的作用,既反映酶切連接效果的好壞,又決定后續選擇性擴增反應成功與否.預擴增的目的是為選擇性擴增提供大量的模板,同時對模板起到選擇性純化的作用,以使選擇性擴增能產生清晰、穩定、易重復的條帶[12-13].本試驗酶切和接頭的混合樣本分別稀釋10倍(帶1和4)、20倍(帶2和5)和30(帶3和6)倍進行預擴增.圖4顯示,樣品稀釋的倍數對預擴增影響的效果不是很明顯,3個梯度的稀釋效果都較好,擴增信號強,相對分子質量大小在200~1 000 bp之間出現連續、均勻的彌散帶,表明預擴增效果較好,稀釋后可為選擇性擴增反應提供理想的模板.

圖3 產氫細菌基因組DNA不同酶切時間的酶切效果

圖4 預擴增反應圖譜
2.4 選擇性引物組合的選擇
選擇性引物的篩選很重要,不同的引物組合擴增效果明顯不同.而且不同的微生物基因組DNA,同一引物組的擴增效果也大不相同.選擇性引物中3'末端選擇性核苷酸數目的多少確定了AFLP擴增產物的多少,一般選擇性堿基數目越少,擴增的條帶數越多;選擇性堿基數目越多,擴增的條帶數越少.條帶太多,不利于分辨;而條帶太少,則不利于多態性的檢出[14].因此,根據具體的實驗材料即基因組DNA的大小確定引物末端所需的選擇性核苷酸數目.
哈爾濱產乙醇厭氧發酵產氫細菌的基因組較小,所以,采用的擴增策略是在兩種限制性內切酶的核心序列及識別序列基礎上分別在3'增加一個選擇性堿基(A,C,G,T)進行隨機組合,表1顯示的是擴增產氫野生菌株ZGX4和突變菌株YR-3的選擇性引物的序列.選擇稀釋10倍的預擴增產物為模板,利用0.8%瓊脂糖凝膠電泳對16個限制性酶切引物組合擴增出的DNA片段多態性進行檢測.每一個引物組合的試驗對象為野生產氫菌株ZGX4(左側)和突變菌株YR-3(右側),結果表明,不同引物組合的選擇性擴增DNA多態性分布差異較為明顯,具體見圖5所示.其中以1 (EcoRI-A/MseI-A)、2(EcoRI-A/MseI-C)、5(EcoRI-C/MseI-A)及6(EcoRI-C/MseIC)為選擇性擴增引物組合擴增酶切片段的PCR效果表現出的差異性不明顯,產氫菌株ZGX4和YR-3的DNA酶切片段的PCR產物集中分布在100~500 bp之間,DNA擴增片段太小,表明基因組DNA片段的多態性豐度不夠,不能很好地表現出基因組酶切片段的多態性差異,因此,本試驗可以確定(EcoRI-A/MseI-A)、(EcoRI-A/MseIC)、(EcoRI-C/MseI-A)及(EcoRI-C/MseIC)引物組合不適合作為哈爾濱產乙醇桿菌屬細菌ZGX4和YR-3的選擇性擴增引物.相對而言,利用引物組合4(EcoRI-A/MseI-G)、9(EcoRIG/MseI-A)、10(EcoRI-G/MseI-C)、13 (EcoRI-T/MseI-A)、14(EcoRI-T/MseI-C)、15(EcoRI-T/MseI-G)、16(EcoRI-T/MseIT)對產氫野生菌株ZGX4和突變菌株YR-3的酶切片段進行PCR的擴增結果顯示,擴增片段在100~1 800 bp之間呈現出明顯的特異性及多態性分布.而引物3(EcoRI-A/MseI-G)、7(EcoRIC/MseI-G)、8(EcoRI-C/MseI-T)和 12 (EcoRI-G/MseI-T)組合的DNA擴增片段在瓊脂糖凝膠中表現相似的的分布狀態,野生菌株和突變菌株的DNA酶切片段的擴增產物所呈現出的特異性不強,差異微小,因此,不適合作為哈爾濱產乙醇桿菌屬AFLP檢測技術的選擇性引物組合.
①參見北京市第一中級人民法院[2001]一中知初字第185號“白秀娥與國家郵政局、國家郵政局郵票印制局”一案。
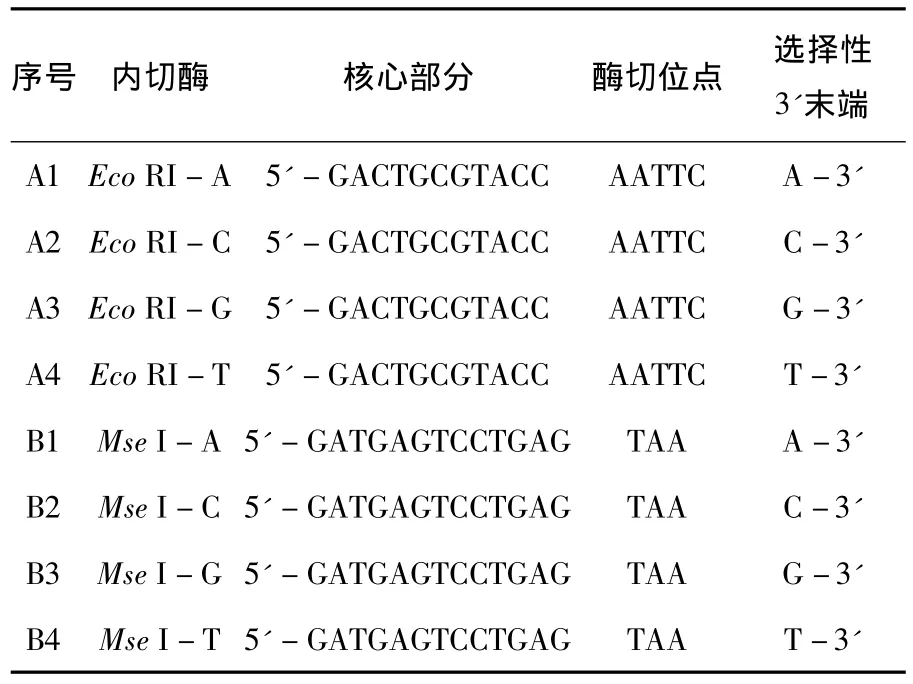
表1 選擇性引物的序列
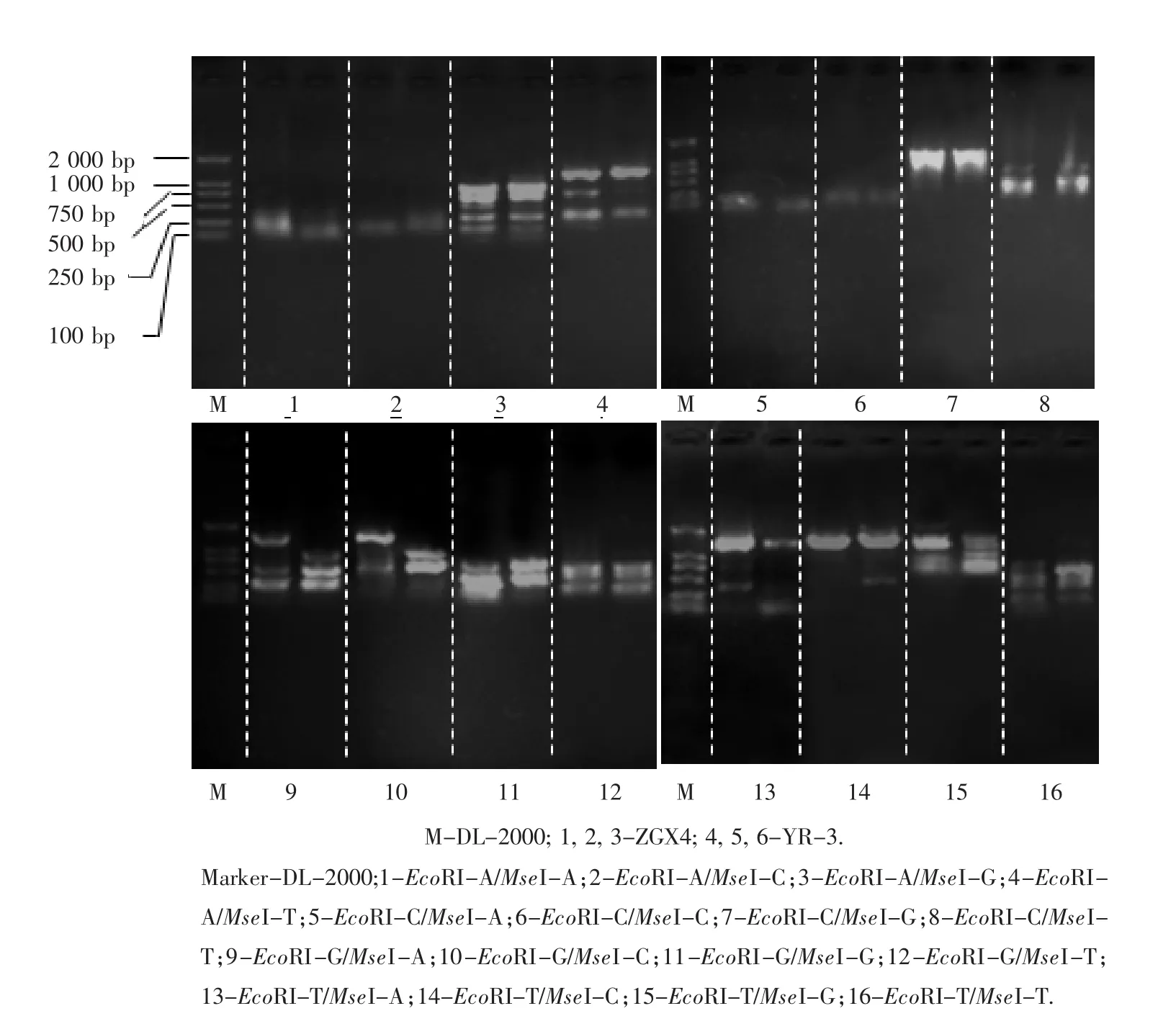
圖5 不同引物組合擴增產氫細菌ZGX4和YR-3的PCR產物在瓊脂糖凝膠電泳中的多態性
3 結論
1)AFLP的反應體系為 20 μL,利用EcoRI/MseI作為產氫細菌YR-3和ZGX4基因組DNA的限制性內切酶,最適酶切基因組DNA質量和純度(OD260/OD280比值)范圍分別為50~100 ng和1.8~1.9.
2)最佳酶切用量和酶切時間分別為EcoRI (2.0 μL)/MseI(0.7 μL)和3 h;預擴增產物稀釋10倍為模板,以引物組合EcoRI-A/MseIG、EcoRI-G/MseI-A、EcoRI-G/MseI-C、EcoRI-T/MseI-A、EcoRI-T/MseI-C、EcoRI-T/MseI-G和EcoRI-T/MseI-T進行選擇性擴增,效果較為理想,可以獲得較為明顯的DNA多態性分布差異.
參考文獻:
[1] GANTER P F,BARROS D E,LOPES M.The use of anonymous DNA markers in assessing worldwide relatedness in the yeast species pichia kluyveri bedford and kudrjavzev[J].Can J Microbiol,2000,46(11):967-980.
[2] ZIMNOCH-GUZOWSKA E,MARCZEWSKI W,LEBECKA R,et al.QTL analysis of new sources of resistance to Erwinia carotovo-rassp.atrosepticain potato done by AFLP,RFLP and resistance gene-like markers[J].Crop Sci,2000,40:1156-1167.
[3] MCULIFFE L,KOKOTOVIC B,AYLING R D,et al. Molecular epidemiological analysis of mycoplasma bovis isolates from the United Kingdom shows two genetically distinct clusters[J].J Clin Microbiol,2004,42 (10):4556-4565.
[5] KUMAR V,SHARMA S,KERO S,et al.Assessment of genetic diversity in common bean(Phaseolus vulgaris L.)germ plasm using amplified fragment length polymorphism(AFLP)[J].Scientia Horticu Lturae,2008,116:138-143.
[6] THOMAS C,VOS C,ZABEAN M,et al.Identification of amplified restriction fragment length polymorphism(AFLP)markers lightly linked to the tomatoCf-9 gene for resistence to Cladosporiumfluvum[J].The Plant Jour,1995,8:785-794.
[7] SIMONS G,VANDER L T,DIERGRADE P,et al. AFLP-based fine mapping of the Mlo gene to a 30- kb DNA segment of the barley genome[J].Genetics,1997,44:61-70.
[8] 鄭國香,任南琪,李永峰,等.一株高效產氫突變體RF-9的篩選與產氫特性[J].中國環境科學,2007,27(2):184-188.
[9] VOS P R,HOGERS M,BLEEKER M,et al.AFLP: a new concept for DNA fingerprinting[J].Nucleic Acids Res,1995,23:4407-4414.
[10] WANG Z,GAO H W,WU Y Q,et al.Genetic diversity and population structure of Caragana korshinskii revealed by AFLP[J].Crop Science,2007,47:1737-1743.
[11] LARSON S R,JONES T A,JEBSEN K B.Population structure in Pseudoroegneria spicata(Poaceae: Triticeae)modeled by Bayesian clustering of AFLP genotypes[J].American Journal of Botany,2004,91(11):1789-1801.
[12] 宋曉飛,李曉麗,馮志紅,等.黃瓜基因組DNA提取及AFLP體系優化研究[J].安徽農業科學,2009,37(29):14035-14037.
[13] 陳旭輝,高玉葆,朱敏杰,等.小葉錦雞兒基因組DNA的提取及AFLP反應體系的建立[J].植物研究,2009,29(5):529-533.
[14] SHI J,XIN L,YANG Y,et al.Discussion on AFLP molecular markers in Pipermethysticum and pepper[J].Agricultural Science&Technology,2007,8(3/ 4):5-10.

