LC 基因在細胞自噬過程中的表達研究
倪志華+張玉明+鄧傳懷+等

摘要:自噬是細胞為維持自身代謝發生的胞質組分降解的動態過程。自噬不僅是細胞在饑餓環境下的一種適應性反應,而且對于組織和細胞維持動態平衡十分重要。細胞自噬出現問題往往會導致多種疾病。自噬相關基因被命名為Atg(Autophagy-related gene),LC3(Microtubule-associated protein 1 light chain 3)是哺乳動物中了解的最透徹的一個Atg同源蛋白。LC3蛋白在細胞中存在兩種剪切形式,即LC3-Ⅰ和LC3-Ⅱ, 定位于自噬體膜的LC3-Ⅱ是自噬的標志性分子。詳細論述自噬過程和LC3在自噬過程中的表達修飾過程,并進一步討論自噬在神經退行性疾病和微生物感染中的生物學功能。
關鍵詞:自噬;LC3;神經退行性疾病;免疫應答
中圖分類號:Q786 文獻標識碼:A 文章編號:0439-8114(2015)20-4932-05
DOI:10.14088/j.cnki.issn0439-8114.2015.20.002
Expression of LC3 Gene in Cell Autophagy
NI Zhi-hua1,2,ZHANG Yu-ming1,DENG Chuan-huai1,ZHAO Ze1,ZHANG Rui-ying1,ZHOU Yan-fen1,2
(1.College of Life Sciences,Hebei University,Baoding 071002,Hebei,China;
2.Research Center of Bioengineering of Hebei Province,Baoding 071002,Hebei,China)
Abstract:Autophagy is a dynamic process for degradation of cytosolic components in cell, aiming to maintain cells metabolic balance. Autophagy is an adaptive reaction of nutrient deficiency. Moreover, autophagy is very important for tissue and cell to keep dynamic metabolic balance. Disturbance of autophagy could trigger various diseases. Usually, autophagy-related gene is named as Atg. Microtuble associated protein lightchain 3 (LC3), a mammalian homolog of yeast Atg8, has been extensively reported. In cell,LC3 proteins are presented in two forms (LC3-Ⅰand LC3-Ⅱ). LC3-Ⅱ, locating on autophosome membrane, is usually used as a specific marker to monitor autophagy. In this review, the process of autophagy and the function of LC3 were involved. Moreover, the biological function of autophagy in neurodegenerative disease and immune response were also discussed intensively.
Key words:autophagy;LC3;neurodegenerative disease;immune response
自噬(Autophagy)一詞源于希臘詞語“Self”和“Eating”,指細胞為了維持自身的代謝平衡而自我犧牲的一種重要的細胞功能[1]。1993年,Tsukada等[2]發現了釀酒酵母(Saccharomyces cerevisiae)的細胞自噬現象,并對自噬相關的基因突變體以Atg(Autophagy-related gene)進行了命名和性質研究。從酵母到哺乳動物,細胞自噬過程是高度保守的。目前,哺乳動物的多種組織和器官中都觀測到了自噬的發生。研究發現,自噬途徑對細胞清除功能缺陷的蛋白質和細胞器,維持細胞的生存、分化,組織的發育十分重要[3]。如果自噬途徑受損或自噬相關基因的改變會導致一系列的疾病,例如神經退行性疾病及各種癌癥[4]。本文將就自噬過程以及哺乳動物細胞中參與自噬的標志性分子LC3 (Microtubule-associated protein 1 light chain 3)在自噬過程中的表達修飾概述,并闡述自噬在神經退行性疾病和免疫應答反應中的生物學功能。
1 自噬過程
細胞自噬包括三種主要類型:大自噬(Macroautophagy)、小自噬(Microautophagy)和分子伴侶介導的自噬(Chaperone mediated autophagy,CMA),此外還有一些器官特異性的自噬途徑[5]。其中大自噬(以后簡稱為自噬,Autophagy)是細胞內自身發生的大規模的降解過程,包括細胞質組分的降解和細胞器成分的分解。待降解的細胞組分會被稱為自噬體的雙層膜結構所包裹,然后被運送至溶酶體中進行降解。小自噬是不需任何載體的協助,溶酶體直接攝取細胞組分并進行降解的過程[6]。分子伴侶介導的自噬有別于大自噬和小自噬的是,待降解的底物蛋白必須存在一段五肽序列,熱休克蛋白HSP70與底物蛋白結合形成底物-分子伴侶復合物,溶酶體CMA受體識別該復合物,并將底物去折疊后轉運至溶酶體中進行消化[7]。
自噬是從酵母到哺乳動物細胞都普遍存在的保守的代謝途徑。一般認為自噬的動態過程分成5個緊密相連的步驟:歐米伽體(Omegasome)的形成,自噬體膜的形成與延伸,自噬體的形成,自噬體—溶酶體的融合,自噬體內物質被溶酶體水解酶降解等過程[8]。
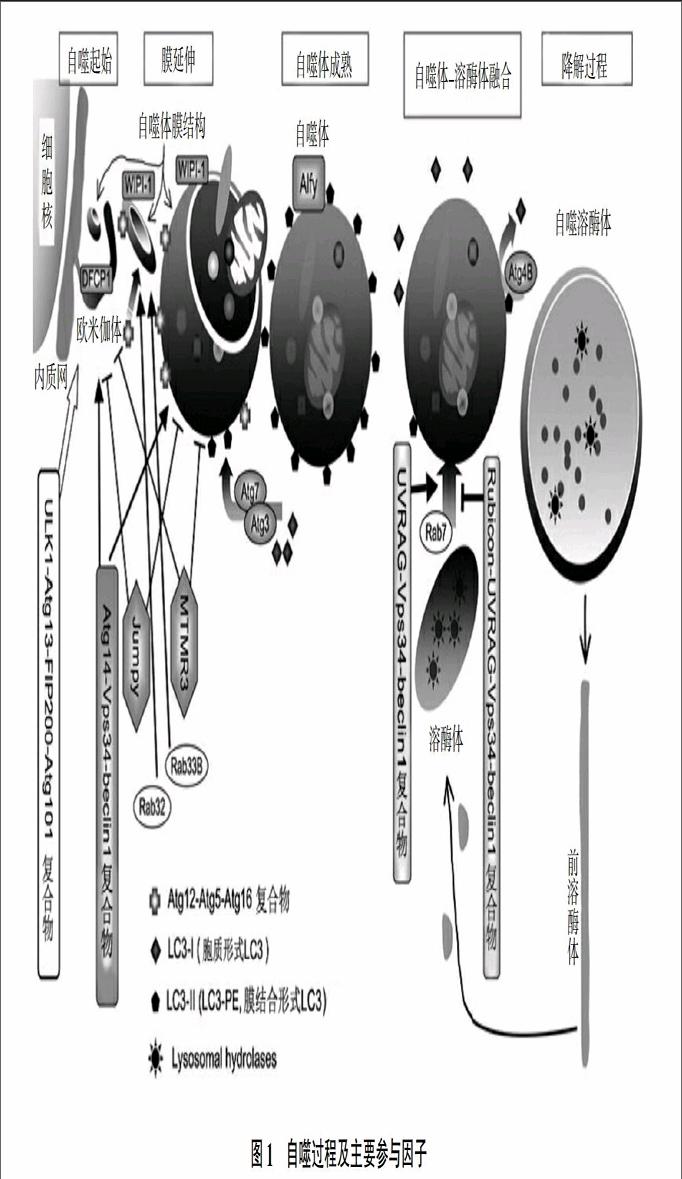
細胞自噬過程大多可在10~15 min內完成,如圖1所示。首先是mTOR(mammalian target of rapamycin)激酶信號途徑激活細胞內的泛素樣激酶1(Ubiquitin like kinase 1,ULK)-Atg13-FIP200(Focal adhesion kinase family interacting protein of 200 kD)-Atg101復合物,誘導自噬的起始。隨著自噬的開始,內質網通過與DFCP1(Double FYVE-containing protein 1)結合形成歐米伽體,同時,Atg13招募Atg14-Vps34(PI3KC3的激酶亞基)-beclin1蛋白,促進自噬體膜的形成并為自噬體的形成貯備原料。歐米伽體與Atg5-Atg12-Atg16L復合物結合形成孤立的自噬膜結構,該膜結構的進一步延伸過程中細胞質組分包括線粒體和內質網被包裹起來。隨著自噬體膜結構的進一步延伸,Atg5-Atg12-Atg16L從膜上解離,膜結構發生閉合,形成自噬體,LC3-II在自噬體的形成過程中定位于封閉的自噬體膜結構上。最后溶酶體與自噬體發生融合,形成自噬體-溶酶體復合物。在自噬體-溶酶體復合物內,被包裹的細胞質組分被溶酶體中的水解酶所降解,其中LC3-II也隨之降解。降解結束后,從自噬體-溶酶體復合物變成長形結構的原溶酶體,原溶酶體在細胞中會再次形成溶酶體。
由此可見,在哺乳動物細胞中,核心的Atg蛋白分成5個亞組:ULK1蛋白激酶復合物[9]、Vps34-beclin1 III型PI3-激酶復合物、Atg9-WIPI1(WD-repeat domain protein interacting with phosphoinositides)復合物[10]、Atg12結合系統[11]以及LC3結合系統[12]。對于自噬的發生,順序進入的蛋白激酶復合物、磷酸化酶、脂類以及ATP依賴的結合系統復合物是必需的,缺少任何一個組分均會導致自噬受損。自噬啟動之初,PI3激酶和mTOR激酶是必需的[13],Vps34-beclin1 III型PI3激酶復合物在自噬中也是必不可少的[14],此外,自噬過程中還有兩個泛素化系統Atg12結合系統和LC3結合系統對自噬體的形成十分重要。
自噬過程主要是由mTOR信號通路調節。在生理條件下,mTOR途徑通過抑制泛素樣激酶ULK的活性來抑制自噬的發生[6]。在饑餓狀態下,mTOR的活性被抑制,ULK被激活從而誘導細胞啟動細胞自噬。因此,mTOR可激活饑餓誘導的細胞自噬細胞。
2 LC3在自噬過程中的表達
自噬相關基因(Autophagy-related genes)對于細胞及生物個體十分重要,自噬相關基因的缺失不僅會導致酵母細胞和黏細菌細胞的快速死亡,而且對于剛脫離母體的新生小鼠也是致命的。缺失自噬相關基因的小鼠表現為神經細胞和肝細胞蛋白質的積累[15]。
大多數自噬相關基因在酵母、真菌、多細胞動物和綠色植物以及幾種原生動物中都相對保守。自噬相關基因Atg8(Autophagy-related 8 genes,Atg8s)的研究起源于酵母。到目前為止在哺乳動物中至少發現了4個Atg8的同源物:LC3、GABARAP、GATE-16、Atg8L[16]。其中,LC3是了解的最透徹的一個Atg8同源蛋白,目前已知人類有4個編碼LC3的基因(MAP1LC3A、MAP1LC3B、MAP1LC3B2、MAP1LC3C)[17],分別定位于染色體20q11.22、16q24.2、12q24.22和1q43。蛋白質三級結構研究發現,LC3和其他Atg8家族的蛋白結構類似,如圖2所示[18],因其有與泛素十分相似的結構,被稱為泛素樣蛋白。作為自噬形成中重要的一個組分,LC3結合系統的目的是進行磷脂化(PE),因此LC3連接系統也被稱為LC3-脂化系統。LC3蛋白在細胞中存在兩種剪切形式LC3-Ⅰ和LC3-Ⅱ,LC3-Ⅰ定位于細胞質,LC3-Ⅱ定位于自噬體膜。
細胞營養缺乏時,LC3基因的轉錄被激活,從而翻譯出足夠的LC3前體。在自噬體膜形成過程中,LC3前體被Atg4B剪切形成具有C-末端甘氨酸的胞質形式——LC3-Ⅰ。LC3-Ⅰ的C末端甘氨酸幫助催化其與Atg7和Atg3活性位點處的半胱氨酸形成硫酯鍵,并且促進LC3-Ⅰ與磷脂以酰胺鍵進行結合形成LC3-PE,即膜結合形式的LC3-Ⅱ[19]。整個反應過程由Atg7和Atg3介導并且有泛素化折疊。隨著自噬體的形成,LC3-Ⅱ逐漸聚集并定位于自噬體膜上。在此過程中LC3-Ⅱ的表達量逐漸增加[20]。之后,自噬體與溶酶體融合,自噬溶酶體降解,自噬體膜表面的LC3-Ⅱ被Atg4B脂化重新變成LC3-Ⅰ,以完成LC3-Ⅰ在自噬體形成過程中的再次利用[21];自噬溶酶體內的LC3-Ⅱ被溶酶體的水解酶降解。LC3-Ⅰ修飾成LC3-II 的過程不是孤立的,此過程會受到很多因素的影響。在缺乏Atg12-Atg5的結合時,修飾過程無法進行[22]。此外,有學者發現了兩個新的自噬調節因子Nix(BCL2-related protein) 和 DOR (Diabetes- and obesity-regulated gene),能夠與LC3蛋白結合參與自噬過程中靶蛋白的選擇或者自噬體的形成與聚集[23,24]。
LC3-Ⅱ對于自噬體的形成十分必要,有研究表明去除LC3-Ⅱ將形成一系列不閉合的自噬體結構[25]。因LC3-Ⅱ的數量和自噬體的數量存在一一對應的關系,LC3-Ⅱ被普遍認為是哺乳動物自噬體的標記物。試驗中常常通過Western blotting、免疫熒光和GFP-LC3法檢測LC3用以衡量細胞或組織的自噬行為[26]。
3 自噬的生物學功能
自噬的功能首先是細胞在饑餓環境下的一種適應性反應[27]。細胞處于營養缺乏的環境時,會啟動自噬來重復利用自身的材料產生游離的氨基酸以應對饑餓脅迫[28]。自噬在細胞的發育和分化過程中起著十分重要的作用[29]。在細胞發育、變態和細胞分化過程中細胞重塑的動態過程需要細胞成分的大規模降解。研究證明在米曲霉[30]乃至人的角質細胞[31]、皮膚和毛發的分化[32]、心肌細胞分化和心臟發育[33]、T細胞的發育分化過程中[34]都需要Atg基因的參與。
此外,研究發現自噬過程對于組織和細胞維持動態平衡十分重要。細胞自噬與多種疾病相關,例如神經退行性疾病、病原微生物所引發的細胞免疫應答、心肌病、腫瘤、糖尿病、脂肪肝及克羅恩病等[35-37]。
神經退行性疾病包括阿爾茲海默癥(Alzheimers diseases,AD)、帕金森病(Parkinsons disease,PD)、亨廷頓舞蹈癥(Huntingtons disease,HD)等[38]。自噬異常在這些疾病的病理學和發病過程中起著十分重要的作用。神經元細胞是高度分化的極性細胞,其活力和功能的維持除了需要有賴于神經營養因子的存在,還必須活躍的膜運輸在細胞體與樹突和軸突之間進行連接。神經元細胞的高度極化特性,也使其對細胞質組分或細胞膜的聚集和積累更加敏感,而要清除錯誤折疊的蛋白質其中最重要的途徑之一就是自噬-溶酶體途徑。該途徑可以在細胞內對胞漿的長壽蛋白和細胞器進行降解[39]。因此,在神經系統中,自噬的主要作用是維持神經元細胞內蛋白的合成與降解水平的平衡,對于諸如神經細胞這樣的長壽細胞,自噬功能和自噬水平的穩定尤其重要。當自噬功能異常時往往會導致神經退行性疾病[40,41]。
此外,自噬的管家功能是與先天性和獲得性免疫相關[42],使細胞遠離不必要的或者可能有毒的環境及作為監控體系控制病原體入侵自噬具有胞內抗病毒作用。病原微生物感染可以通過不同的途徑影響自噬。一方面自噬作為防御機制參與先天性免疫或者獲得性免疫。當病原微生物侵入細胞后,即被自噬識別,然后被自噬體吞噬,最終被自噬溶酶體降解。另一方面,有些病原菌也進化出了不同的策略來破壞自噬途徑,從而逃脫了溶酶體的降解并利用該途徑所產生的氨基酸進行菌體代謝。這時自噬的功能就變成了清除感染的細胞[43]。自噬清除病原體的能力與病原體自身的性質有關。目前研究發現,當細胞感染結核分枝桿菌(Mycobacterium tuberculosis)或者A群鏈球菌(Group A streptococcus)時,自噬不僅承擔清除入侵者的任務還在有2型MHC存在時起著遞呈抗原的作用。在病毒侵染時,自噬體以病毒組分作為靶標,啟動溶酶體降解系統降解病毒粒子和病毒的主要組分,在病毒感染的免疫應答系統啟動過程中發揮重要作用[44]。總之,精確測定自噬活性對于研究感染后的免疫應答十分重要,而且調節自噬活性也將成為抗感染治療的一種途徑。
4 展望
自噬作為維持自身平衡的重要細胞代謝過程,擔負著重復利用細胞質中游離物質和清除受損細胞器和有毒分子的作用。LC3的結構和在細胞自噬中的作用已經研究清楚,LC3檢測已經成為自噬體檢測的一種重要手段。
研究表明,除了應對營養缺乏所致的細胞饑餓,自噬還可以清除細胞代謝過程中產生的錯誤折疊蛋白,以維持細胞正常的生理功能,并且在細胞感染微生物后積極地參與細胞免疫。自噬功能的缺乏或異常可能是神經退行性疾病的主要原因。總之,研究自噬會對相關疾病的發病機理和治療起到重要的推動作用。
參考文獻:
[1] EISENBERG-LERNER A, BIALIK S, SIMON H-U, et al. Life and death partners: Apoptosis, autophagy and the cross-talk between them[J]. Cell Death and Differentiation, 2009, 16(7):966-975.
[2] TSUKADA M,OHSUMI Y. Isolation and characterization of autophagy-defective mutants of Saccharomyces cerevisiae[J]. FEBS Letters, 1993, 333(1-2):169-174.
[3] RAVIKUMAR B, SARKAR S, DAVIES J E, et al. Regulation of mammalian autophagy in physiology and pathophysiology[J]. Physiological Reviews, 2010, 90(4):1383-1435.
[4] MIZUSHIMA N,LEVINE B, CUERVO A M, et al. Autophagy fights disease through cellular self-digestion[J]. Nature, 2008, 451(7182):1069-1075.
[5] MANJITHAYA R,NAZARKO T Y,FARRE J-C, et al. Molecular mechanism and physiological role of pexophagy[J]. FEBS Letters, 2010, 584(7):1367-1373.
[6] GLICK D, BARTH S, MACLEOD K F. Autophagy: Cellular and molecular mechanisms[J]. The Journal of Pathology, 2010, 221(1):3-12.
[7] KAUSHIK S,BANDYOPADHYAY U,SRIDHAR S,et al. Chaperone-mediated autophagy at a glance[J]. Journal of Cell Science, 2011, 124(Pt 4):4995-4999.
[8] ALAVIAN S M, ANDE S R, COOMBS K M, et al. Virus-triggered autophagy in viral hepatitis-possible novel strategies for drug development[J]. Journal of Viral Hepatitis, 2011, 18(12):821-830.
[9] MIZUSHIMA N. The role of the Atg1/ULK1 complex in autophagy regulation[J]. Current Opinion in Cell Biology, 2010, 22(2):132-139.
[10] HE C C,KLIONSKY D J.Atg9 trafficking in autophagy-related pathways[J]. Autophagy, 2007, 3(3):271-274.
[11] MIZUSHIMA N, NODA T, YOSHIMORI T, et al. A protein conjugation system essential for autophagy[J]. Nature,1998,395(6700):395-398.
[12] KUMA A, MATSUI M, MIZUSHIMA N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: Caution in the interpretation of LC3 localization[J]. Autophagy, 2007, 3(4):323-328.
[13] PETIOT A, OGIER-DENIS E, BLOMMAART E F, et al. Distinct classes of phosphatidylinositol 3'-kinases are involved in signaling pathways that control macroautophagy in HT-29 cells[J]. The Journal of Biological Chemistry, 2000, 275(2):992-998.
[14] MATSUNAGA K, SAITOH T, TABATA K, et al. Two Beclin 1-binding proteins, Atg14L and Rubicon, reciprocally regulate autophagy at different stages[J]. Nature Cell Biology, 2009, 11(4):385-396.
[15] KOMATSU M, WAGURI S, CHIBA T, et al. Loss of autophagy in the central nervous system causes neurodegeneration in mice[J]. Nature, 2006, 441(7095):880-884.
[16] TANIDA I, SOU Y, MINEMATSU-IKEGUCHI N, et al. Atg8L/APg8L is the fourth mammalian modifier of mammalian Atg8 conjugation mediated by human Atg4B, Atg7 and Atg3[J]. Febs Journal, 2006, 273(11):2553-2562.
[17] SHPILKA T, WEIDBERG H, PIETROKOVSKI S, et al. Atg8: An autophagy-related ubiquitin-like protein family[J]. Genome Biology, 2011, 12(7):226.
[18] SUGAWARA K, SUZUKI N N, FUJIOKA Y, et al. The crystal structure of microtubule-associated protein light chain 3, a mammalian homologue of Saccharomyces cerevisiae Atg8[J]. Genes to Cells, 2004, 9(7):611-618.
[19] SOU Y S, TANIDA I, KOMATSU M, et al. Phosphatidylserine in addition to phosphatidylethanolamine is an in vitro target of the mammalian Atg8 modifiers, LC3, GABARAP, and GATE-16[J]. The Journal of Biological Chemistry, 2006, 281(6):3017-3024.
[20] TAGUCHI-ATARASHI N, HAMASAKI M, MATSUNAGA K, et al. Modulation of local PtdIns3P levels by the PI phosphatase MTMR3 regulates constitutive autophagy[J]. Traffic, 2010, 11(4):468-478.
[21] TANIDA I. Autophagy basics[J]. Microbiology and Immunology, 2011, 55(1):1-11.
[22] OHSUMI Y, MIZUSHIMA N. Two ubiquitin-like conjugation systems essential for autophagy[J]. Seminars in Cell & Developmental Biology, 2004, 15(2):231-236.
[23] SCHWEERS R L, ZHANG J, RANDALL M S, et al. NIX is required for programmed mitochondrial clearance during reticulocyte maturation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(49):19500-19505.
[24] MAUVEZIN C, ORPINELL M, FRANCIS V A, et al. The nuclear cofactor DOR regulates autophagy in mammalian and drosophila cells[J]. EMBO Reports, 2010, 11(1):37-44.
[25] SOU Y, WAGURI S, IWATA J, et al. The Atg8 Conjugation system is indispensable for proper development of autophagic isolation membranes in mice[J]. Molecular Biology of the Cell, 2008, 19(11):4762-4775.
[26] GONG C, BAUVY C, TONELLI G, et al. Beclin 1 and autophagy are required for the tumorigenicity of breast cancer stem-like/progenitor cells[J]. Oncogene, 2013, 32(18):2261-2272.
[27] MIZUSHIMA N. The pleiotropic role of autophagy: From protein metabolism to bactericide[J]. Cell Death and Differentiation, 2005, 12 (Suppl 2):1535-1541.
[28] XIE Z P, KLIONSKY D J. Autophagosome formation: Core machinery and adaptations[J]. Nature Cell Biology, 2007, 9(10):1102-1109.
[29] NEZIS I P, VACCARO M I, DEVENISH R J, et al. Autophagy in development, cell differentiation, and homeodynamics: From molecular mechanisms to diseases and pathophysiology[J]. BioMed Research International, 2014, 2014: Article ID 349623.
[30] KIKUMA T, OHNEDA M, ARIOKA M, et al. Functional analysis of the ATG8 homologue Aoatg8 and role of autophagy in differentiation and germination in Aspergillus oryzae[J]. Eukaryotic Cell, 2006, 5(8):1328-1336.
[31] AYMARD E, BARRUCHE V, NAVES T, et al. Autophagy in human keratinocytes: An early step of the differentiation?[J]. Experimental Dermatology, 2011, 20(3):263-268.
[32] YOSHIHARA N, UENO T, TAKAGI A, et al. The significant role of autophagy in the granular layer in normal skin differentiation and hair growth[J]. Archives of Dermatological Research, 2015, 307(2):159-169.
[33] SIMON H U. Autophagy in myocardial differentiation and cardiac development[J]. Circulation Research, 2012, 110(4):524-525.
[34] BRONIETZKI A W, SCHUSTER M, SCHMITZ I. Autophagy in T-cell development, activation and differentiation[J]. Immunology and Cell Biology, 2015, 93(1):25-34.
[35] LEVINE B, KROEMER G. Autophagy in the pathogenesis of disease[J]. Cell, 2008, 132(1):27-42.
[36] ESKELINEN E-L, SAFTIG P. Autophagy: A lysosomal degradation pathway with a central role in health and disease[J]. Biochimica et Biophysica Acta(BBA)-Molecular Cell Research, 2009, 1793(4):664-673.
[37] VIRGIN H W, LEVINE B. Autophagy genes in immunity[J]. Nature Immunology, 2009, 10(5):461-470.
[38] XILOURI M, STEFANIS L. Autophagy in the central nervous system: Implications for neurodegenerative disorders[J]. Cns & Neurological Disorders-Drug Targets, 2010, 9(6):701-719.
[39] NIJHOLT D A, DE KIMPE L, ELFRINK H L, et al. Removing protein aggregates: The role of proteolysis in neurodegeneration[J]. Current Medicinal Chemistry, 2011, 18(16):2459-2476.
[40] CIECHANOVER A. Proteolysis: From the lysosome to ubiquitin and the proteasome[J]. Nature Reviews Molecular Cell Biology, 2005, 6(1):79-87.
[41] RUBINSZTEIN D C. The roles of intracellular protein-degradation pathways in neurodegeneration[J]. Nature, 2006, 443(7113):780-786.
[42] LEVINE B, DERETIC V. Unveiling the roles of autophagy in innate and adaptive immunity[J]. Nature Reviews Immunology, 2007, 7(10):767-777.
[43] CAMPOY E, COLOMBO M I. Autophagy in intracellular bacterial infection[J]. Biochimica et Biophysica Acta-Molecular Cell Research, 2009, 1793(9):1465-1477.
[44] KERNBAUER E, CADWELL K. Autophagy, viruses, and intestinal immunity[J]. Current Opinion in Gastroenterology, 2014, 30(6):539-546.

