重慶市豌豆枯萎病病原菌的分離與鑒定
張慧 馬連杰 馮牧野


摘要:采用組織分離法,對重慶市豌豆枯萎病病原菌進行分離和純化,結合形態學和基因序列分析對分離物進行鑒定,采用無傷接種法測定分離物對豌豆離體葉片、莖稈的致病性。結果表明,從枯萎病樣品中分離獲得5株菌株,菌落為圓形,初期為白色絮狀,后期呈黃棕色至褐色;供試菌株的ITS序列和G3PDH基因序列與Ascochyta pinodes的同源性達到99%;致病性測定結果表明,供試菌株對豌豆葉片及莖稈均有致病性。表明引起重慶市豌豆枯萎病的病原菌為Ascochyta pinodes。
關鍵詞:豌豆;豌豆枯萎病;病原鑒定;Ascochyta pinodes
中圖分類號:S436.43 ? ? ? ? 文獻標識碼:A
文章編號:0439-8114(2020)06-0084-04
DOI:10.14088/j.cnki.issn0439-8114.2020.06.016 ? ? ? ? ? 開放科學(資源服務)標識碼(OSID):
Isolation and identification of pathogen of pea fusarium wilt in Chongqing city
ZHANG Hui,MA Lian-jie,FENG Mu-ye,LU Wen-cai,LIAO Dun-xiu
(Institute of Agricultural Resources and Environment,Chongqing Academy of Agricultural Sciences,Chongqing 401329,China)
Abstract: The tissue isolation method was used to isolate and purify the pathogen of spot of fusarium wilt of peas in Chongqing city, and the isolates were identified by combining with the analysis of morphology and gene sequence. The pathogenicity of the isolates to the leaves and stems of isolated peas was determined by the method of inoculation without injury. The results showed that five strains were isolated from infected leaves. The colony was round, white flocculent in the early stage and yellowish brown to brown in the later stage; The homology of ITS sequence and G3PDH gene sequences of the tested strain reached 99% with that of Ascochyta pinodes; The results of pathogenicity determination showed that the strain had pathogenicity to both leaves and stems of pea. It indicated that Ascochyta pinodes was the cause of pea blight in Chongqing city.
Key words: pea; pea fusarium wilt; pathogens identified; Ascochyta pinodes
豌豆是中國食用豆的一個主要品種,中國豌豆生產量占全球豌豆總產量的60%以上。中國是第二大干豌豆生產國家,2013年干豌豆產量達1 566億t。在生產上,豌豆病害是制約產量提高的重要因素。已報道的豌豆病害有25余種[1],而豌豆殼二孢枯萎病(常被稱為黑斑病)是豌豆最嚴重的病害之一,分布于世界各地,包括幾乎所有種植豌豆的區域[2]。該病菌可為害植株的根、莖、葉和豆莢,造成豌豆產量減少10%~60%[1,3]。殼二孢枯萎病是由Ascochyta pinodes(有性態Mycosphaerella pinodes)、Phoma medicaginis、Ascochyta pisi(有性態Didymella pisi)和Phoma koolunga復合侵染引起的真菌病害[4,5]。這種枯萎病復合物引起一系列不同的癥狀,包括黑斑、腳腐、莖腐、葉片和莢果斑點。Ascochyta pinodes可以侵染豌豆植株的幼苗和所有地上部分,引起壞死性葉斑,莖基部變黑,幼苗蛀爛。由Phoma pinodella引起的癥狀與Ascochyta pinodes癥狀相似。然而,Phoma pinodella侵染可導致更嚴重的基部腐爛癥狀,可以延伸到地面以下,同時對葉、莖和莢果造成的損害較小。Ascochyta pisi侵染葉片、莢果和莖,這種病原菌通常不會侵染豌豆植物莖基部,引起腳腐[6]。這3種病原菌均可以引起豌豆枯萎病,且又極其相似,鑒定過程中容易將其混淆。通過調查發現豌豆枯萎病在重慶市普遍發生,且嚴重影響豌豆的產量和品質,但目前國內對該病害的報道較少[7],并且不同地區引起該病害的病原菌種類也各不相同。鑒于此,本研究基于形態學特征、ITS序列和功能基因G3PDH序列分析鑒定病原菌種類,并經柯赫氏法則對病菌的致病性進行驗證,以明確重慶市豌豆枯萎病的致病菌,為進一步研究該病害的發病規律、防治措施和豌豆抗病品種的培育提供科學依據。
1 ?材料與方法
1.1 ?材料
1.1.1 ?供試植物 ?于2016年11—12月從永川區和墊江縣沙坪鎮發現豌豆病株,采集具有典型癥狀的豌豆葉片及莖稈,將病葉及莖稈保存于標本袋內帶回實驗室進行病原菌分離。
1.1.2 ?培養基 ?①馬鈴薯葡萄糖瓊脂(PDA)培養基:馬鈴薯200 g,葡萄糖20 g,瓊脂12 g,去離子水1 000 mL。②燕麥瓊脂培養基(OA):燕麥片30 g,瓊脂15 g,去離子水1 000 mL。
1.1.3 ?試劑及儀器 ?PCR擴增試劑盒2×Taq PCR Mix,生工生物工程(上海)股份有限公司;DL 2 000 DNA Marker,寶生物工程大連有限公司;其余試劑均為國產分析純。MJ Mini PCR 擴增儀,美國伯樂公司;DYY-7B電泳儀,北京六一儀器廠;UVP凝膠成像系統,美國UVP公司;BPX-272恒溫培養箱,上海博迅實業有限公司醫療設備廠。
1.2 ?方法
1.2.1 ?病原菌的分離與純化 ?每株豌豆選取莖、葉2個部位,先用無菌水沖洗,從病葉病健交界處剪切2~3 mm小塊組織,在75%乙醇中浸泡2~3 s,再用1%的氯化汞溶液浸泡3 min,再用滅菌水沖洗4次,然后用無菌濾紙吸去多余的水分,用無菌鑷子將組織塊放在PDA培養基上培養。25 ℃恒溫、黑暗培養5 d后,將長出的菌落進行純化,純化后的菌株于4 ℃冰箱中保存備用。
1.2.2 ?菌株的致病性測定 ?采用無傷接種法進行致病性測定。隨機選取1株純化菌株WHYC2平板培養活化。取健康新鮮無傷的豌豆葉10片,用自來水沖洗干凈,經75%乙醇表面消毒,晾干,將測試菌株菌餅(5 mm)貼接于葉片和莖基部,對照用無菌培養基塊,放入鋪有濕潤濾紙的發芽盒內,每個測試菌株分別接種豌豆葉片及莖稈,每個葉片、莖稈接種1個位點,于25 ℃恒溫培養7 d,觀察發病情況。
1.2.3 ?病原菌的鑒定 ?①病原菌的形態鑒定。將菌株接種于PDA培養基、OA培養基中,25 ℃光暗交替培養,6 d后觀察菌落顏色、形狀。②病原菌的分子鑒定。用CTAB法提取基因組DNA,采用通用引物ITS4/ITS5和AscGAPDH-F/AscGAPDH-R[7]進行PCR擴增,引物合成及測序由北京六合華大基因科技有限公司完成。PCR反應條件:95 ℃預變性5 min;95 ℃變性30 s,55 ℃退火30 s,72 ℃延伸1 min,共35個循環;72 ℃延伸10 min。采用1.0%瓊脂糖凝膠電泳檢測擴增結果,測序所得序列在GenBank中進行同源性比對,用MEGA 6.0軟件以鄰接法構建系統發育樹。
2 ?結果與分析
2.1 ?病原菌的分離
從豌豆病葉病莖上共分離得到5株菌株,從永川區分離到2株,墊江縣分離到3株,該菌為害豌豆植物的幼苗和所有地上部分,在葉上形成病斑,并不斷擴展,形成棕色和褐色的圓環(圖1a)。莖上的病斑一般是藍黑色或黑紫色,造成莖基部變黑壞死和葉斑癥狀(圖1b),壞死斑很少擴展至土表以下,引起種子皺縮和變色,病斑上生小黑點。發生嚴重時病斑密集成片,葉片枯死,全田發病植株莖葉灰色干枯死亡(圖1c)。
2.2 ?病原菌的致病性
菌株WHYC1接種3 d后接種部位出現明顯的病害癥狀,接種點發暗,周圍出現水漬狀腐爛。繼續保濕條件下,葉部病斑繼續擴大,水漬狀部位連成一片呈軟腐狀(圖2a)。莖稈的接種部位變黑呈長條狀,棕褐色病斑(圖2b)。采集發病葉片進行病原菌再次分離,所得的分離物與WHYC1菌落形態一致,證實分離菌為豌豆枯萎病病原菌。
2.3 ?病原菌的形態特征
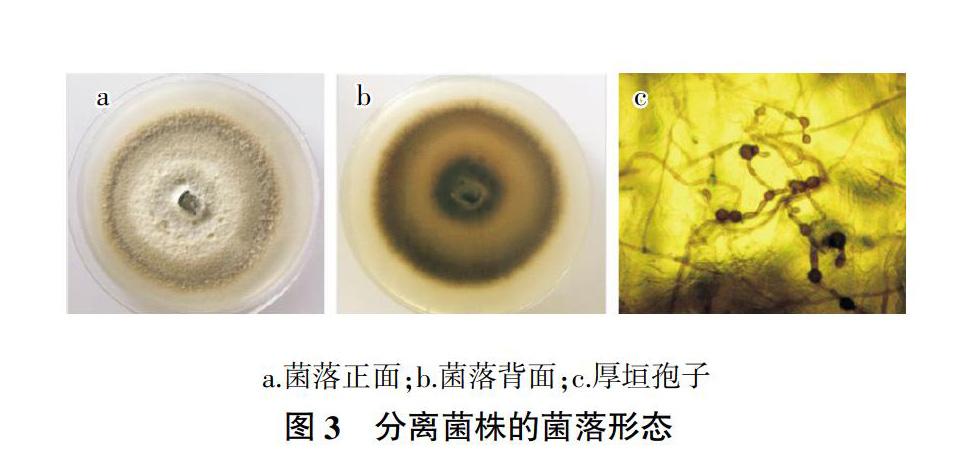
菌株WHYC1在PDA上進行純培養,前6 d內,菌落為白色,圓形,菌絲絮狀,致密,不易產生子座、分子孢子器等子實體(圖3a、圖3b);15 d后,菌落變成黃棕色至褐色;在PDA長時間培養后,菌絲老熟變褐色并產生厚垣孢子(圖3c),厚垣孢子單個或成串產生在菌絲頂端或中間,黑褐色,球形至不規則形,多數光滑。培養條件下未發現分生孢子器及分生孢子。
2.4 ?病原菌rDNA-ITS
利用真菌通用引物ITS1/ITS4對菌株WHYC1的基因組DNA進行PCR擴增,獲得約520 bp的序列。BLAST分析結果表明,菌株WHYC1的ITS序列與GenBank中Ascochyta pinodes(FJ165554.1)、Ascochyta pinodes(FJ200219.1)、Phoma pinodella PC2(DQ826427.1)和Phoma pinodella CBS(AY831562.1)的同源性高達99%。從GenBank數據庫中選取代表菌株ITS序列,以Phoma macrostoma ATCC 24524 (DQ474069.1)、Pleosporales sp. M1C(KP122266.1)、Peyronellaea prosopidis CPC(KF777180.1)作為外群建立系統發育樹,結果顯示,病原菌菌株WHYC1與親緣關系較近的Ascochyta pinodes和Phoma pinodella菌株在系統發育樹中聚為一類,但ITS不能區分Ascochyta pinodes和Phoma pinodella(圖4)。
2.5 ?病原菌G3PDH基因序列分析
利用引物AscGAPDH-F/AscGAPDH-R對代表菌株WHYC1進行G3PDH基因序列擴增,均獲得約492 bp的序列。BLAST分析結果表明,菌株WHYC1的G3PDH序列與GenBank中Ascochyta pinodes MP2(DQ383976.1)同源性高達99%。從GenBank數據庫中選取代表菌株G3PDH序列,以Ascochyta pisi AP5(DQ383965.1)作為外群建立系統發育樹,結果顯示,G3PDH基因序列可將殼二孢屬(Ascochyta)的不同種顯著區分,菌株WHYC1與Ascochyta pinodes位于同一分支,聚為一類,殼二孢屬的其他種位于不同分支,進一步確定重慶豌豆枯萎病的病原菌為Ascochyta pinodes(圖5)。
3 ?小結與討論
殼二孢屬引起的豌豆枯萎病是由多種真菌引起的病害,包括Ascochyta pinodes、Phoma pinodella、Ascochyta pisi、Phoma koolunga,Phoma herbarum和Phoma glomerata。這些病原菌可以同時發生在一塊豌豆田,甚至同時發生在一株豌豆上。然而,殼二孢真菌的種群結構和分布在不同地區均有所不同,Ascochyta pinodes是加拿大和法國豌豆的主要致病菌[8-13]。Ascochyta pinodes和Phoma pinodella是澳大利亞豌豆主要致病菌,而Phoma koolunga主要分布在澳大利亞南部[14]。在立陶宛Ascochyta pisi是豌豆枯萎病主要的致病菌,占復合物分離的一半,而在某些地方,Ascochyta pinodes和Phoma pinodella則是主要致病菌[15]。在本研究中,從永川區以及墊江縣分離得到5株病原菌,通過ITS和G3PDH序列鑒定,5株菌均為Ascochyta pinodes。前人研究表明,ITS序列可以將Ascochyta pinodes和Phoma pinodella與Ascochyta pisi和Phoma koolunga區分開,但是不能區分Ascochyta pinodes和Phoma pinodella[4,15,16],與本研究結果一致。通過G3PDH序列構建系統發育樹進行分析能有效地區分Ascochyta pinodes和Phoma pinodella[15]。本研究中,根據形態學特征ITS和G3PDH序列分析確定,Ascochyta pinodes是引起重慶市蠶豆枯萎病的主要病原菌。有關該病害的發生流行規律、侵染循環及防治措施等還需進一步研究。
參考文獻:
[1] KRAFT J M,PFLEGER F L. Compendium of pea diseases and pests[M].St Paul,Minnesota:American phytopathological society press,2001.
[2] BRETAG T W,KEANE P J,PRICE T V. The epidemiology and control of ascochyta blight in field peas: A review[J].Australian journal of agricultural research,2006,57(8):883-902.
[3] TIVOLI B,B?魪ASSE C,LEMARCHAND E,et al. Effect of ascochyta blight (Mycosphaerella pinodes) on yield components of single pea (Pisum sativum) plants under field conditions[J].Annals of applied biology,1996,129(2):207-216.
[4] DAVIDSON J A,HARTLEY D,PRIEST M,et al. A new species of Phoma causes ascochyta blight symptoms on field peas (Pisum sativum) in South Australia[J].Mycologia,2009,101(1):120-128.
[5] LIU J F,CAO T S,FENG J,et al. Characterization of the fungi associated with ascochyta blight of field pea in Alberta,Canada[J].Crop protection,2013,54:55-64.
[6] CHILVERS M I,ROGERS J D,DUGAN F M,et al. Didymella pisi sp. nov.,the teleomorph of Ascochyta pisi[J].Mycological research,2009,113(3):391-400.
[7] LIU N,XU S C,YAO X F,et al. Studies on the control of ascochyta blight in field peas (Pisum sativum L.) caused by Ascochyta pinodes in Zhejiang province,China[J].Frontiers microbiology,2016,7:481.
[8] MOUSSART A,TIVOLI B,LEMARCHAND E,et al. Role of seed infection by the ascochyta blight pathogen of dried pea (Mycosphaerella pinodes) in seedling emergence, early disease development and transmission of the disease to aerial plant parts[J].European journal of plant pathology,1998,104(1):93-102.
[9] TIVOLI B,BANNIZA S. Comparison of the epidemiology of ascochyta blights on grain legumes[J].European journal of plant pathology,2007,119:59-76.
[10] GOSSEN B D,HWANG S F,CONNER R L,et al. Managing the ascochyta blight complex on field pea in western Canada[J].Prairie soils and crops journal,2011,4:135-141.
[11] LE MAY C,GUIBERT M,LECLERC A,et al. A single,plastic population of Mycosphaerella pinodes causes ascochyta blight on winter and spring peas (Pisum sativum) in France[J].Applied and environmental microbiology,2012,78(23):8431-8440.
[12] AHMED H,CHANG K F,HWANG S F,et al. Morphological characterization of fungi associated with the ascochyta blight complex and pathogenic variability of Mycosphaerella pinodes on field pea crops in central Alberta[J].The crop journal,2015,3(1):10-18.
[13]ESNULEVIIEN R,GAURILIKIEN I,RAMANAUSKIEN?J. Control of ascochyta blight (Ascochyta complex) in pea under Lithuanian conditions[J].Zemdirbyste-Agriculture,2014,101(1):101-108.
[14] FATEHI J,BRIDGE P D,PUNITHALINGAM E. Molecular relatedness within the “Ascochyta pinodes-complex”[J].Mycopathologia,2003,156(4):317-327.
[15] PEEVER T L,BARVE M P,STONE L J,et al. Evolutionary relationships among Ascochyta species infecting wild and cultivated hosts in the legume tribes Cicereae and Vicieae[J].Mycologia,2007,99(1):59-77.
[16] TADJA A,BENKADA M Y,RICKAUER M,et al. Characterization of ascochyta as pathological species of pea (Pisum sativum L.) at the north-west of Algeria[J].Journal of agronomy,2009,8(3):100-106.

