基于Weka平臺和代價敏感特征選擇的基因表達數據分類研究
韓磊,黃瑞龍,范文靜,葉明全
皖南醫學院 醫學信息學院,安徽 蕪湖 241002
0 引言
腫瘤是目前人類在疾病面前面臨的主要威脅之一。據2014年的《世界癌癥》報告[1]顯示,僅2012年一年就有超過1000萬的癌癥新發病例。腫瘤不是瞬間產生的[2],腫瘤細胞的分類增殖存在一個相對較長的演變時期。因此,這種在基因層面對腫瘤進行早期識別的研究[3],對患者的治療具有重大意義[4-5]。
由于腫瘤基因表達數據是一種典型的不平衡數據[6-7],使之很難直接應用于腫瘤的分類診斷[8-9]。因此本文提出了一種基于Weka平臺和代價敏感特征選擇的基因表達數據分類方法[10]用于解決這類基因表達數據分布不平衡的分類問題[11-12]。該方法彌補了分類器只注重分類精度的片面性,并且它的合理性在于通過引入代價敏感而尋求總體的最小代價,而不是僅僅擁有精度最高這個特性。通常在代價敏感學習中,對于一個N分類問題,用
1 資料與方法
1.1 資料來源
本文實驗從Kent Ridge Biomedical Data Set數據庫中選取兩個類別,共計六個小組的腫瘤樣本數據,分別為神經系統疾病NervSys(central nervous system embryonal tumor)、結腸癌(colon cancer)、彌漫性大B細胞瘤(DLBCL)、卵巢癌(ovarian cancer)、前列腺癌(prostate cancer)和肺癌(lung cancer)。數據集的詳細描述見表1。
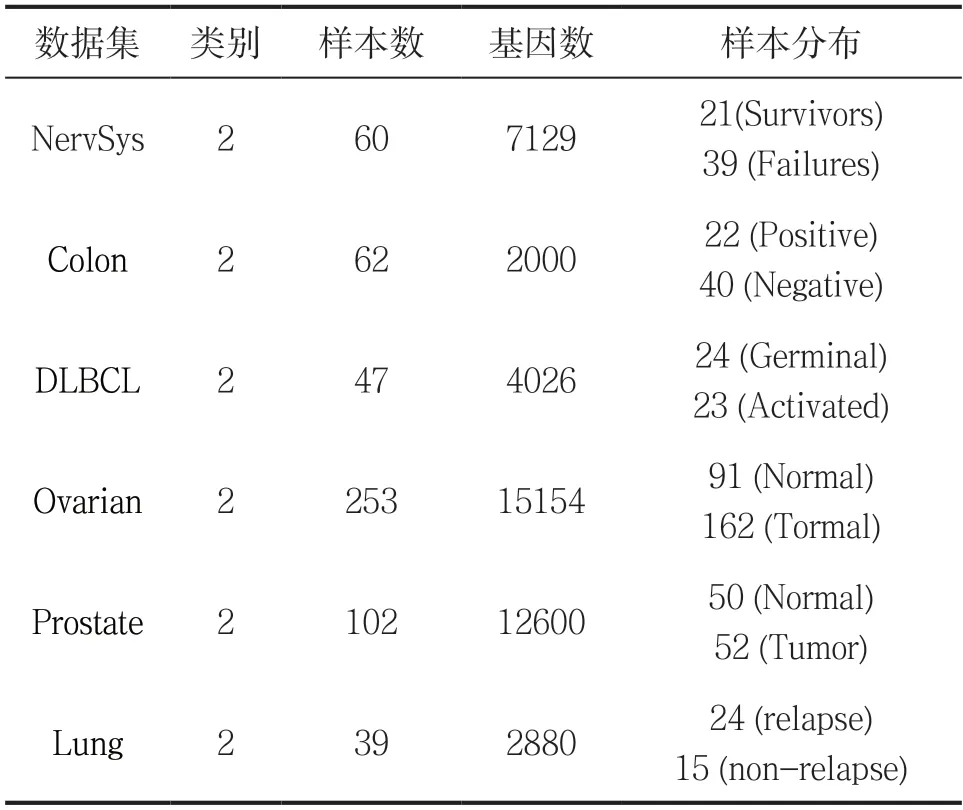
表1 實驗數據集描述
1.2 實驗方法
本文實驗基于Windows 平臺完成和實現。為了消除不同量綱對實驗結果的影響,實驗過程中,我們首先通過Weka平臺[13-14]對實驗數據集進行標準化預處理,使數據分析更加準確,然后選擇本文提出的代價敏感特征選擇方法(cost sensitive attribute eval),使用特征選擇的搜索函數Ranker來調整信息基因個數,并且通過支持向量機(SVM)、K近鄰(IBK)、樸素貝葉斯(NB)和隨機森林(RF)這4種分類器對數據進行分類得到的分類準確率來評估該方法的有效性。在實驗過程中均使用Weka平臺中分類器的默認參數。具體實驗流程見圖1。
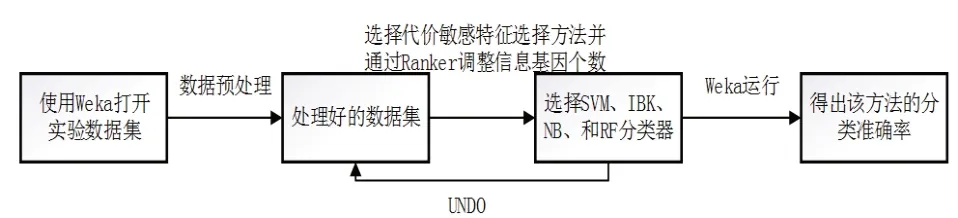
圖1 實驗流程圖
2 實驗結果及分析
表2為4種分類器在6組兩類別的腫瘤樣本數據的分類準確率,表中Std表示在原始實驗數據[15]上只執行標準化處理后就進行4種分類器的分類建模,本文方法即通過代價敏感特征選擇[16]處理后再進行4種分類器的分類建模。
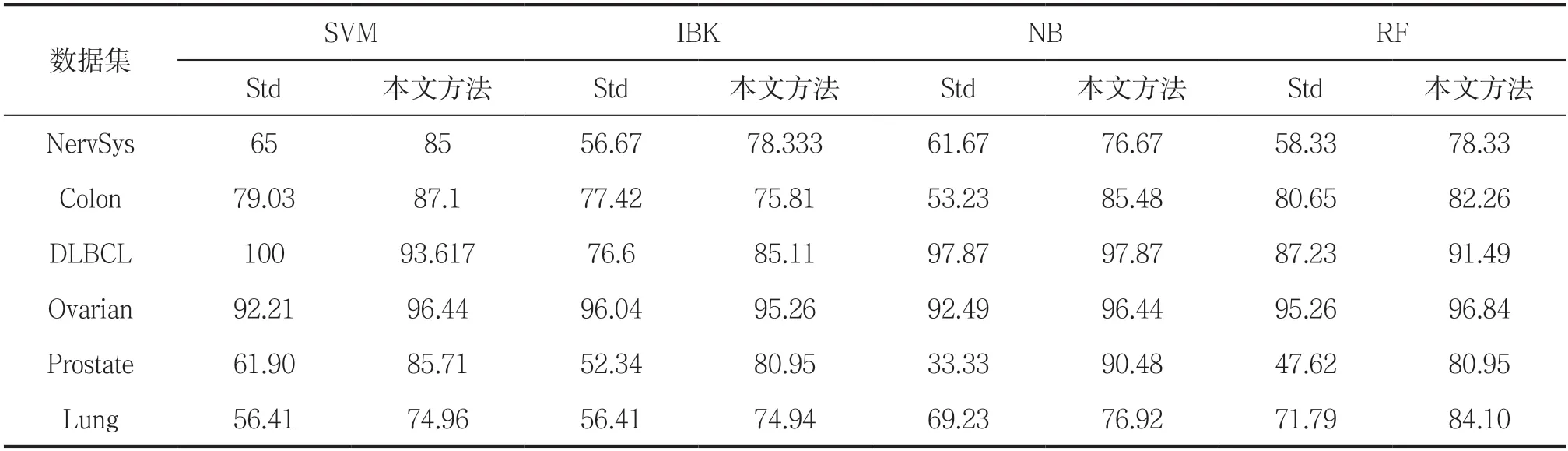
表2 4 種分類器在6 個數據集上的分類準確率對比
為了方便對比,本文實驗選擇的信息基因數分別為3、4、5、6,選擇4種分類器中最高分類準確率作為最終評價值。具體實驗結果見表2。
從圖2可以看出,六組數據直接在只進行標準化預處理后,在SVM、IBK、NB和RF分類器評估分類性能時,大部分分類準確率較低。但是通過本文實驗方法得到的分類準確率大部分高于只進行標準化預處理的分類準確率,這在一定程度上說明了本文提出的代價敏感特征選擇方法的有效性。

圖2 4 種分類器在6 個數據集上的分類準確率對比
為了更直觀地表明本文方法在提高分類準確率上的優良性能,實驗還對比分析了其他兩種流行特征選擇方法的分類準確率。包括SUAE(symmetrical uncert atrribute eval)根據屬性的對稱不確定性評估屬性和CA(correlation attribute)通過測量特征與類別之間的皮爾遜(Pearson's)相關性評估基因的價值。具體實驗結果見表3。

表3 3 種方法在6 個數據集上的最優分類準確率
從圖3可以看出,對比SUAE和CA的特征選擇方法在六組數據的最優分類準確率,本方法也獲得了相對更好的分類準確率,進一步有力地證明了該方法具有良好的特征選擇效果,能夠針對腫瘤基因表達數據獲取較高的分類性能。
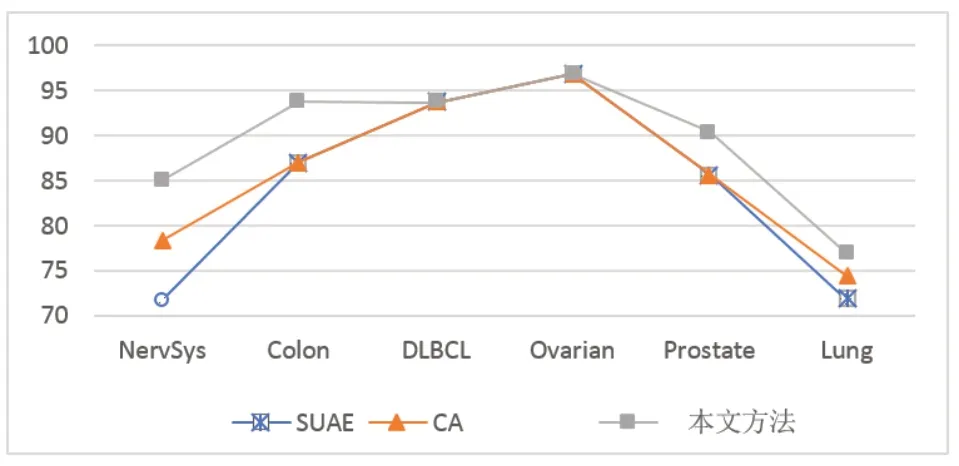
圖3 3 種方法在6 個數據集上的最優分類準確率
3 結論
本文提出的基于Weka平臺和代價敏感特征選擇的基因表達數據分類方法可以有效地解決腫瘤基因表達數據不平衡數據的分類問題,大幅度提高分類準確率,但仍存在一些不足和缺陷[17],如分類過程中真實的誤分類代價很難通過人為經驗進行準確估計。由于此方法本身的性能指標與代價參數設置等方面存在一定空缺,可能會導致其最終的分類結果存在相對較強的主觀性而不夠客觀,因此代價敏感算法[18]仍有繼續完善優化的空間。通過改變一些相關代價參數從而進一步改進本文方法等方式,都是今后的研究方向。

