沉默減數分裂內切酶1(EME1)抑制肝癌細胞增殖的機制
陳椿 王可欣 賀夢雯 李樂 王春艷 劉妍 紀冬


摘要: 目的 探討減數分裂內切酶1 (EME1) 在肝癌組織中的表達及其對肝癌細胞生物學行為的影響。方法 篩選TCGA數據庫肝癌樣本中的差異表達基因。采用免疫組化和Western Blot分析EME1在肝癌組織中的表達豐度。通過短發夾RNA(shRNA) 構建慢病毒并感染BEL-7404細胞干擾EME1基因表達, 分為沉默組 (shEME1) 和對照組 (shCtrl)。通過實時熒光定量PCR法和Western Blot檢測兩組EME1 mRNA和蛋白表達水平, Celigo計數法及MTT活性檢測細胞增殖率, 流式細胞術檢測細胞周期, Caspase3/7活性檢測細胞凋亡。兩組間比較采用成組t檢驗。結果 TCGA結果顯示EME1的mRNA表達水平在肝癌組織中是癌旁組織的18. 9倍 (114. 5±153. 0 vs 8. 0±7. 2, t=5. 00, P<0. 001); EME1的蛋白表達水平在肝癌組織中是癌旁組織的7. 0倍(免疫組化檢測, 8. 4±2. 6 vs 1. 2±0. 4, t=7. 55, P<0. 001)和2. 5倍(Western Blot檢測, 249. 0%±35. 5% vs 100. 0%±77. 8%, t=3. 02, P<0. 05)。慢病毒感染后, 相對于對照組, 沉默組EME1的mRNA表達水平下降了29. 9%(29. 9%± 0. 9% vs 100. 0%±3. 6%, t=32. 82, P<0. 001), 蛋白表達水平顯著下降了35. 7% (35. 7%±14. 9% vs 100. 0%±28. 9%, t=3. 42, P<0. 05); 細胞計數下降了45. 1% (4 053±167 vs 8 988±477, t=16. 91, P<0. 001)、 細胞活性下降至66. 9% (0. 518±0. 046 vs 0. 774±0. 022, t=8. 74, P<0. 001) 及細胞克隆形成能力下降至29. 0% (75±6 vs 260±9, t=28. 92, P<0. 001)。與對照組比較, 沉默組G1期細胞 (49. 9% vs 44. 0%, t=8. 96, P<0. 001) 比例增多, G2/M期 (15. 9% vs 17. 9%, t=9. 13, P<0. 001) 與S期 (34. 2% vs 38. 1%,t=6. 91, P<0. 001)的細胞比例減少; Caspase3/7活性增強了1. 5倍(145. 8%±5. 9% vs 100. 0%±2. 3%, t=12. 50, P<0. 001)。結論 EME1在肝癌組織中高表達, 沉默EME1基因可抑制肝癌細胞增殖, 促進細胞凋亡。
關鍵詞: ?癌, ?肝細胞; ?減數分裂內切酶1; ?RNA, ?小分子干擾; ?細胞增殖
基金項目: ?北京市自然科學基金面上項目 (7222173)
Silencing essential meiotic endonuclease ?1 inhibits the proliferation of liver cancer cells: A study of?related mechanisms
CHEN Chun1, WANG Kexin2, 3, HE Mengwen2, 4, LI Le5, WANG Chunyan2, LIU Yan5, JI Dong1, 2, 3, 4. (1. The Second Clinical Medical School of Southern Medical University, Guangzhou 510515, China; 2. Senior Department of Hepatology, The Fifth Medical Center of Chinese PLA General Hospital, Beijing 100039, China; 3. PLA 307 Medical College of Anhui Medical University, Hefei 230032, China;4. Peking University 302 Clinical Medical School, Beijing 100191, China;5. Senior Department of Infectious Diseases, The Fifth Medical Center of Chinese PLA General Hospital, Beijing 100039, China)
Corresponding authors: ?JI Dong, ?jidg302@126.com ?(ORCID: ?0000-0001-8214-462X); ?LIU Yan, ?liuyan5360@163.com ?(ORCID: ?0000-0003-1498-4314)
Abstract: ?Objective To investigate the expression of essential meiotic endonuclease ?1 (EME1) ?in liver cancer tissue and its?effect on the biological behavior of hepatoma cells. Methods The TCGA database was used to identify the differentially expressed genes between liver cancer tissue and paracancerous tissue. Immunohistochemistry and Western Blot were used to measure the expression abundance of EME1 in liver cancer tissue. A lentivirus was constructed by short hairpin RNA, and BEL-7404 cells were transfected with the lentivirus to interfere with the expression of the EME1 gene; the cells were divided into silencing group (shEME1 group) and control group (shCtrl group) . Quantitative real-time PCR and Western Blot were used to measure the mRNA and protein expression levels of EME1; Celigo Image Cytometer and MTT assay were used to measure cell proliferation rate; flow cytometry was used to observe cell cycle; Caspase 3/7 activity was used to measure cell apoptosis. The independent-samples t-test was used for comparison between two groups. Results TCGA results showed that the mRNA expression level of EME1 in liver cancer tissue was 18.9 times that in paracancerous tissue (t=5.00, P<0.001), and the protein expression level of EME1 in liver cancer tissue was 7.0 times (based on immunohistochemistry: 8.4±2.6 vs 1.2±0.4, t=7.55, P<0.001) or 2.5 times (based on Western Blot: 249.0%±35.5% vs 100.0%±77.8%, t=3.02, P<0.05) that in paracancerous tissue. After lentivirus infection, compared with the shCtrl group, the shEME1 group had an mRNA expression level of EME1 reduced by 29.9% (29.9%±0.9% vs 100.0%±3.6%,
t=32.82, P<0.001), a protein expression level of EME1 reduced by 35.7% (35.7%±14.9% vs 100.0%±28.9%, t=3.42, P<0.05), and a level of cell counting reduced by 45.1% (4 053±167 vs 8 988±477, t=16.91, P<0.001), as well as a level of cell activity reduced to 66.9% (0.518±0.046 vs 0.774±0.022, t=8.74, P<0.001) and a level of colony forming ability reduced to 29.0% (75±6 vs 260±9, t=28.92, P<0.001) . Compared with the shCtrl group, the shEME1 group had a significant increase in the proportion of cells in G1 phase (49.9% vs 44.0%, t=8.96, P<0.001) and significant reductions in the proportion of cells in G2/M phase (15.9% vs 17.9%, t=9.13, P<0.001) and S phase (34.2% vs 38.1%, t=6.91, P<0.001), while Caspase 3/7 activity was enhanced by 1.5 times (145.8%±5.9% vs 100.0%±2.3%,t=12.50, P<0.001) . Conclusion EME1 is highly expressed in liver cancer tissue, and silencing the EME1 gene can inhibit the proliferation of hepatoma cells and promote cell apoptosis.
Key words: Carcinoma, Hepatocellular; Essential Meiotic Endonuclease 1; RNA, Small Interfering; Cell Proliferation
Research funding: Beijing Municipal National Science Foundation (7222173)
肝細胞癌是全球癌癥相關死亡的第三大常見原因,對于晚期肝癌患者, 靶向治療的效果仍不能滿足臨床需求[1-5] , 尋找新的基因靶點可進一步提升療效及安全性,成為目前肝癌治療領域的研究熱點。減數分裂內切酶1(essential meiotic endonuclease 1, EME1) 編碼的蛋白質可與MUS81形成核酸內切酶復合物, 作用于特定的DNA底物, 包括Holiday連接、 3′ 末端翹翼結構以及復制叉結構, 在DNA鏈間交聯、 DNA雙鏈斷裂后修復過程中對重組中間產物的分解發揮著關鍵作用[6] 。既往研究[7-9] 發現, EME1高表達是膀胱癌、 乳腺癌及腎透明細胞癌不良預后的高危因素, 但其在肝癌中的表達和作用尚未見報道。本研究從TCGA數據庫入手, 通過生物信息學尋找肝癌組織中的高表達基因EME1, 并在肝癌組織/癌旁組織中進行驗證, 通過基因沉默探討EME1對肝癌細胞生物學行為的影響, 以期鑒定新的治療靶點。
1 材料與方法
1. 1 材料 人肝癌細胞BEL-7404/7402、 Hep3B及人胚腎細胞293T均為本單位感染病研究所保存。慢病毒載體購自上海吉凱基因公司。鼠源EME1單克隆抗體購自美國Santa Cruz公司, HRP標記山羊抗小鼠的二抗檢測試劑盒購自北京中杉金橋公司。HiScript Ⅲ All-in one RT、 SuperMix Perfect for qPCR 逆轉錄試劑和 Taq Pro Universal SYBR qPCR Master Mix購自南京諾唯贊公司。RPMI 1640和MEM培養基購自美國Corning公司, 胎牛血清 (FBS) 購自美國Geniala公司, 四甲基偶氮唑鹽 (MTT)購自北京鼎國昌盛公司, Caspase-Glo? 3/7 Assay試劑盒購自美國Promega公司, 碘化丙啶 (PI) 購自吉至生化公司。
1. 2 TCGA數據庫分析 下載TCGA數據庫中肝細胞癌樣本的mRNA seq表達譜, 利用R軟件中的 “edge” 包篩選癌及癌旁組織中高表達基因。
1. 3 細胞培養 BEL-7404/7402細胞在含10%FBS的RPMI 1640細胞培養液、 Hep3B細胞在含10%FBS的MEM細胞培養液中37 ℃、 5% CO2條件下培養。細胞傳代使用0. 25%胰蛋白酶消化, 按照1∶3比例傳代。
1. 4 免疫組化染色 石蠟切片經脫蠟、 水化、 抗原修復后, FBS封閉后加入EME1一抗, 4 ℃孵育過夜, 加入二抗37 ℃孵育20 min, DAB顯色, 蘇木精復染, 中性樹膠封片, 鏡檢評分。染色結果進行半定量分析, 以染色強度評分 [陰性為0分, 淡黃色 (弱陽性) 為1分, 棕黃色 (陽性) 為2分, 棕褐色 (強陽性) 為3分] 和陽性細胞范圍評分[≤25% 為 1 分, 26%~50% 計為 2 分, 51%~75% 為3分, >75%為4分] 乘積作為最終結果。
1. 5 EME1沉默慢病毒載體的構建 EME1 短發夾RN(shRNA) 干擾序列為CTGAGAAGACAGGAAAGAA, 兩端分別添加AgeI (A^CCGGT) 和EcoRI (G^AATTC) 酶切位點黏性末端, 并在正鏈3′ 端添加終止信號 (TTTTT), 反鏈5端添加終止信號互補序列 (AAAAA), 合成DNA Oligo。雙酶切hU6-MCS-CMV-EGFP慢病毒載體使其線性化, 將DNA Oligo與載體質粒相連后轉化感受態細胞, 之后PCR擴增及測序鑒定 (表1)。提取鑒定正確的質粒, 利用第二代慢病毒包裝系統, 穿梭質粒GV115、 包裝質粒psPAX2、 包膜質粒pMD2. G共轉染293T細胞, 轉染后48 進行病毒收獲, 濃縮與純化慢病毒。
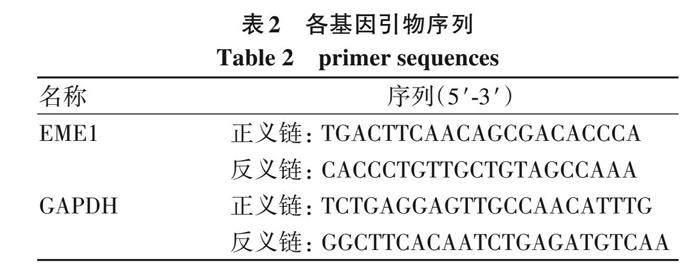
1. 6 EME1沉默慢病毒感染BEL-7404細胞 對數生長期的BEL-7404細胞接種于6孔板 (2×105個/孔), 培養至鋪板量達30%后分別加入shRNA干擾表達慢病毒和陰性對照慢病毒, 感染12 h后更換完全培養基繼續培養。感染72 h后, 熒光顯微鏡觀察細胞感染效率。以GADPH為內參照, 進行實時熒光定量PCR (RT-qPCR) 檢測 (表2) , 2?△△Ct法計算EME1相對表達量。
1. 7 Western Blot檢測 制備樣品, 每孔上樣量為30 ?g,10%的SDS-PAGE電泳分離, 并轉移到PVDF膜上, 室溫封閉1 h, 一抗 (1∶800) 4 ℃孵育過夜, TBST洗膜。二抗(1∶5 000) 室溫孵育1 h, TBST洗膜。化學發光法 (ECL法) 檢測條帶, 以GAPDH為內參照, 用Image J軟件進行灰度值分析。
1. 8 Celigo與MTT檢測 對數生長期細胞接種于96孔板 (2×103個/孔), 每天用Celigo檢測讀板。另設置一組進行MTT檢測, 培養終止前4 h加入5 mg/mL的MTT溶液20 ?L, 繼續培養4 h后吸取培養液, 每孔加入100 ?L二甲基亞砜, 振蕩器振蕩2 min, 檢測490 nm處的吸光值。
1. 9 細胞克隆形成能力檢測 對數生長期細胞接種于6孔板 (800個/孔), 持續培養10 d后使用4%多聚甲醛固定細胞30 min, PBS洗滌后加入500 ?L/孔吉姆薩染液(10 min), ddH2O洗滌細胞, 晾干后拍照并克隆計數。
1. 10 Caspase3/7檢測 對數生長期細胞接種于96孔板(1×104個/孔), Caspase-Glo3/7緩沖液10 mL溶解Caspase-Glo3/7 凍干粉, 每孔加入 Caspase-Glo 反應液 100 ?L,500 r/min離心30 min, 室溫孵育2 h后使用酶標儀測定信號強度。
1. 11 PI-FACS-周期檢測 收集處于對數生長期的細胞, 用4 ℃預冷的D-Hanks洗滌細胞并沉淀, 用4 ℃預冷的75%乙醇固定細胞1 h。1 300 r/min離心5 min, 去固定液, 用4 ℃預冷的D-Hanks洗滌細胞沉淀1次。加入1 mL的細胞染色液重懸, 上機檢測。
1. 12 統計學方法 采用R語言軟件4. 2. 1進行統計分析, 每個實驗均設置3個復孔, 數據以x ˉ±s表示, 兩組間比較采用成組t檢驗。P<0. 05為差異有統計學意義。
2 結果
2. 1 人肝癌細胞中EME1 mRNA表達水平 TCGA數據庫篩選出50對肝癌樣本, EME1 mRNA在肝癌組織(114. 5±153. 0) 的表達量是癌旁組織 (8. 0±7. 2) 的18. 9倍 (t=5. 00, P<0. 001)(圖1a)。RT-qPCR結果顯示, 人肝癌細胞BEL-7404/7402和Hep3B中EME1 mRNA相對表達量 (ΔCT值) 分別為9. 25、 10. 32和10. 89, 均<12, 為高豐度表達 (圖1b)。
2. 2 人肝癌細胞中EME1的蛋白表達水平 選取病理確診的3例肝癌患者的手術切除癌/癌旁組織進行Western Blot分析, 結果顯示肝癌組織EME1蛋白水平 (249. 0%± 35. 5%) 是癌旁組織 (100. 0%±77. 8%) 的2. 5倍 (t=3. 02,P<0. 05)(圖2a、 b)。選取病理確診的5例肝癌患者的手術切除肝癌組織/癌旁組織進行免疫組化分析, 結果顯示肝癌組織EME1蛋白水平 (8. 4±2. 6) 是癌旁組織 (1. 2±0. 4) 的7. 0倍 (t=7. 55, P<0. 001)(圖2c、 d)。
2. 3 EME1穩定沉默BEL-7404細胞系的構建 熒光倒置相差顯微鏡觀察慢病毒感染72 h后的BEL-7404細胞,結果顯示感染效率達到80%以上, 細胞狀態正常 (圖3)。RT-qPCR結果顯示, 沉默組 (shEME1) EME1 mRNA表達水平 (29. 9%±0. 9%) 相較于陰性對照組 (shCtrl)(100. 0%± 3. 6%) 顯著降低 (t=32. 82, P<0. 001)(圖4a); Western Blot結果顯示, shEME1組EME1蛋白表達水平 (35. 7%±14. 9%)相較于shCtrl組 (100. 0%±28. 9%) 顯著降低 (t=3. 42, P<0. 05)(圖4b), 表明EME1穩定沉默的肝癌細胞株BEL-7404構建成功。
2. 4 EME1沉默后BEL-7404細胞增殖能力的變化 Celigo觀察培養1~5天的BEL-7404細胞, 結果顯示隨著培養時間的延長, 相對于對照組, 沉默組細胞數量顯著下降, 培養5天時沉默組為4 053±167, 對照組為8 988±477, 細胞數量下降了45. 1% (t=16. 91, P<0. 001)(圖5a、 b)。MTT檢測培養1~5天的BEL-7404細胞在490 nm處的吸光值,相對于對照組, 沉默組細胞吸光值顯著下降, 培養5天時沉默組為0. 518±0. 046, 對照組為0. 774±0. 022, 細胞活性下降了66. 9% (t=8. 74, P<0. 001)(5c)。培養10天后,沉默組克隆數 (75±6) 較對照組 (260±9) 顯著減少, 細胞克隆形成能力下降了29. 0% (t=28. 92, P<0. 001)(圖5d、e)。表明沉默EME1降低BEL-7404細胞的增殖能力。
2. 5 EME1沉默后BEL-7404細胞周期變化 流式細胞術檢測慢病毒感染5天后的BEL-7404細胞周期, 沉默組的G1期細胞比例顯著高于對照組 (49. 9%±0. 8% vs 44. 0%±0. 9%, t=8. 96, P<0. 001), G2/M期細胞比例顯著低于對照組(15. 9%±0. 2% vs 17. 9%±0. 7%, t=9. 13, P<0. 001), S期細胞比例顯著低于對照組 (34. 2%±0. 6% vs 38. 1%±0. 5%, t=6. 91, P<0. 001)(圖6)。
2. 6 EME1沉默后的BEL-7404細胞凋亡的變化 Caspase3/7活性分析顯示感染shRNA慢病毒5天后, 沉默組細胞的Caspase3/7 活性(145. 8%±5. 9%)較對照組組(100. 0%± 2. 3%) 顯著升高1. 5倍 (t=12. 50, P<0. 001)(圖7) 。
3 討論
EME1對于哺乳動物細胞中 DNA 代謝的各個方面都很重要, Guo等[10]研究發現EME1與胃癌細胞AGS和MGC-803的增殖及侵襲能力密切相關, 沉默EME1基因表達可通過抑制蛋白激酶B活化, 降低糖原合酶激酶-3β、 細胞周期蛋白D1, 從而抑制癌細胞增殖和侵襲, 促進細胞凋亡。基于中國廣西人群的研究[11] 發現EME1的Glu69Asp錯義多態性與肝癌風險增加顯著相關。EME1的另一外顯子Ile350Thr變異會提高乳腺癌的易感性[12] 。
本研究發現沉默EME1的表達可降低肝癌的增殖能力。正常細胞中同源重組 (homologous recombination, HR)相關蛋白受到高度的調控, 對DNA序列具有相當高的特異性, 一旦失調就會導致基因組的不穩定性。MUS81/EME1在異常的時間節點被激活, 可能會導致細胞周期的阻滯、 DNA向子細胞的不對稱分布以及非整倍體。相反,過早的磷酸化會刺激Holiday連接交叉。HR途徑的過表達可能引起DNA重排, 導致癌基因活化或者抑癌基因的雜合性的缺失[13] 。HR介導基因表型的持續變化為腫瘤細胞提供生長優勢, 惡性表型的不斷發展以及耐藥性的產生。有研究[14] 發現, 多發性骨髓瘤細胞和患者樣本中的HR酶活性升高密切相關, 抑制HR活性可顯著減少新基因表型的產生, 上調HR活性會增加基因突變的數量。持續獲得HR介導的新基因, 可能會導致癌基因的激活, 促進腫瘤或耐藥表型的獲得。筆者推測高表達水平的EME1蛋白可能通過催化DNA-RNA雜交的形成導致基因組不穩定, 或通過促成同源性驅動的易出錯過程 (如單鏈退火、 復制模板切換和非等位HR) 來促進突變。
結果表明, 沉默EME1表達后Caspase3/7活性顯著增高, 有效促進細胞凋亡。細胞凋亡主要由Caspase家族蛋白酶執行, Caspase3/7負責對特定的細胞底物進行關鍵的切割, 導致最終的凋亡細胞死亡。癌前病變的DNA損傷可通過誘導細胞凋亡來清除潛在的有害細胞從而阻斷腫瘤生長。這一死亡過程的失控與異常的細胞增殖、 腫瘤的發生和進展有關, 細胞凋亡的失調被認為是癌癥的標志之一[15] 。本研究同時發現沉默EME1表達后G1期細胞比例高于shCtrl組, 表明抑制EME1表達能有效阻滯細胞周期, 抑制腫瘤細胞生長。EME1雖然在G2/M期活性受到嚴格的調控, 但其在細胞全周期中都能檢測到。有研究[16] 證明有絲分裂中Cdk1活性的失調, 會過早激活MUS81/EME1的有絲分裂同源重組,可能會誘導DNA損傷應答缺陷相關的細胞周期改變。
蛋白激酶2 (CK2) 通過磷酸化調控MUS81/EME1復合物的活性, 從而在有絲分裂和DNA復制應激后發揮不同的功能。在細胞有絲分裂過程中, CK2通過磷酸化MUS81/EME1增強其活性, 有助于維持基因組的穩定性。但在細胞受到DNA復制應激后, CK2過度活化MUS81/EME1復合物會導致DNA損傷和染色體不穩定性[17]。事實上, 已經在許多腫瘤中發現CK2過表達[18-19] , 因此可以推測CK2可通過提高MUS81/EME1的生物學功能, 促進人類基因組的不穩定性, 還需要進一步研究確定肝癌中CK2的表達水平與MUS81/EME1的關系。在檢查點激酶缺陷1的細胞中, 由于核苷酸的缺乏不足以維持有絲分裂期DNA合成, MUS81/EME1復合物切割有絲分裂期間合成的新生DNA[20] 。這種機制并不會導致細胞的死亡, 而是造成子細胞遺傳了受損的DNA, MUS81/EME1復合物對晚期復制產物的異常處理會導致基因組不穩定。在FIBP敲低的肺腺癌中, EME1的過表達逆轉了其對肺腺癌細胞增殖的抑制作用[21] 。該研究結果顯示,EME1的過表達顯著減少了 γ-H2AX 病灶形成, 表明EME1 參與肺腺癌細胞增殖和放射耐藥性。FIBP 與STAT3互作通過增強其磷酸化從而提高轉錄活性, 并誘導靶基因EME1的表達。外源性STAT3的過表達也提高了FIBP缺陷肺腺癌細胞中的EME1表達水平, 這一結果在另一項研究[22] 中也得到證實。此外, 腫瘤細胞中的某些特異性的細胞因子常通過結合EME1的啟動子上調其表達。
綜上, EME1在肝癌組織中高表達, 并在細胞功能學層面證明了沉默EME1基因可抑制肝癌細胞的增殖, 阻滯細胞周期, 誘導肝癌細胞的凋亡, 為臨床治療提供了新思路。此外本研究對EME1在肝癌中發生發展限于細胞的功能性實驗, 后續還需對動物模型和分子機制進行下一步的探索。
倫理學聲明: 本研究于2020年7月14日通過解放軍總醫院第五醫學中心倫理委員會審批, 批號: 2020055D。
利益沖突聲明: 本文不存在任何利益沖突。
作者貢獻聲明: 紀冬負責課題設計及擬定寫作思路; 陳椿、 王可欣、 賀夢雯進行實驗研究及撰寫論文; 王春艷、劉妍、 紀冬指導撰寫文章并最后定稿。
參考文獻:
[1] SUNG H, FERLAY J, SIEGEL RL, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. DOI: 10.3322/caac.21660.
[2] LLOVET JM, KELLEY RK, VILLANUEVA A, et al. Hepatocellular car?cinoma[J]. Nat Rev Dis Primers, 2021, 7: 6. DOI: 10.1038/s41572-020-00240-3.
[3] CHEN SH, WANG CY, GUO C, et al. Establishment of a nomogram model for predicting the survival of hepatitis B virus-related hepato?cellular carcinoma[J]. J Clin Hepatol, 2022, 38(7): 1566-1571. DOI: 10.3969/j.issn.1001-5256.2022.07.020.
陳松海, 王春艷, 郭暢, 等. 預測HBV相關肝細胞癌生存的列線圖模型的建立[J]. 臨床肝膽病雜志, 2022, 38(7): 1566-1571. DOI: 10.3969/j.issn.1001-5256.2022.07.020.
[4] WANG JJ, WANG KX, CHEN C, et al. Survival analysis and development of a prognostic nomogram for patients with hepatitis B virus-associated hepatocellular carcinoma[J]. Heliyon, 2023, 9(10): e20850. DOI: 10.1016/j.heliyon.2023.e20850.
[5] JI D, CHEN Y, BI JF, et al. Entecavir plus Biejia-Ruangan compound reduces the risk of hepatocellular carcinoma in Chinese patients with chronic hepatitis B[J]. J Hepatol, 2022, 77(6): 1515-1524. DOI: 10.1016/j.jhep.2022.07.018.
[6] GWON GH, JO A, BAEK K, et al. Crystal structures of the structure-selective nuclease Mus81-Eme1 bound to flap DNA substrates[J]. EMBO J, 2014, 33(9): 1061-1072. DOI: 10.1002/embj.201487820.
[7] HAN YY, ZHENG QY, TIAN Y, et al. Identification of a nine-gene panel as a prognostic indicator for recurrence with muscle-invasive bladder cancer[J]. J Surg Oncol, 2019, 119(8): 1145-1154. DOI: 10.1002/jso.25446.
[8] V?QUAUD E, DESPLANQUES G, J?Z?QUEL P, et al. Survivin con?tributes to DNA repair by homologous recombination in breast can?cer cells[J]. Breast Cancer Res Treat, 2016, 155(1): 53-63. DOI: 10.1007/s10549-015-3657-z.
[9] PENG L, LIANG J, WANG Q, et al. A DNA damage repair gene sig?nature associated with immunotherapy response and clinical prog?nosis in clear cell renal cell carcinoma[J]. Front Genet, 2022, 13: 798846. DOI: 10.3389/fgene.2022.798846.
[10] GUO ZG, LIANG EB, LI W, et al. Essential meiotic structure-specific endonuclease1 (EME1) promotes malignant features in gastric can?cer cells via the Akt/GSK3B/CCND1 pathway[J]. Bioengineered, 2021, 12(2): 9869-9884. DOI: 10.1080/21655979.2021.1999371.
[11] WANG YX, HUANG XL, SU ZH, et al. The Glu69Asp polymorphism of EME1 gene is associated with an increased risk of hepatocellular carcinoma in Guangxi population, China[J]. Int J Gen Med, 2022, 15: 7855-7866. DOI: 10.2147/IJGM.S383261.
[12] ZHAO JW, LIU L, ZHANG AQ, et al. Effect of EME1 exon variant Ile350Thr on risk and early onset of breast cancer in southern Chinese women[J]. J Biomed Res, 2013, 27(3): 193-201. DOI: 10.7555/JBR.27.20130013.
[13] SUZUKI T, YASUI M, HONMA M. Mutator phenotype and DNA double-strand break repair in BLM helicase-deficient human cells[J]. Mol Cell Biol, 2016, 36(23): 2877-2889. DOI: 10.1128/MCB.00443-16.
[14] SHAMMAS MA, SHMOOKLER REIS RJ, KOLEY H, et al. Dysfunc?tional homologous recombination mediates genomic instability and progression in myeloma[J]. Blood, 2009, 113(10): 2290-2297. DOI: 10.1182/blood-2007-05-089193.
[15] FULDA S. Tumor resistance to apoptosis[J]. Int J Cancer, 2009, 124(3): 511-515. DOI: 10.1002/ijc.24064.
[16] SZAKAL B, BRANZEI D. Premature Cdk1/Cdc5/Mus81 pathway acti?vation induces aberrant replication and deleterious crossover[J]. EMBO J, 2013, 32(8): 1155-1167. DOI: 10.1038/emboj.2013.67.
[17] PALMA A, PUGLIESE GM, MURFUNI I, et al. Phosphorylation by CK2 regulates MUS81/EME1 in mitosis and after replication stress[J]. Nucleic Acids Res, 2018, 46(10): 5109-5124. DOI: 10.1093/nar/gky280.
[18] GUO YG, ZHU ZH, HUANG ZY, et al. CK2-induced cooperation of HHEX with the YAP-TEAD4 complex promotes colorectal tumorigen?esis[J]. Nat Commun, 2022, 13(1): 4995. DOI: 10.1038/s41467-022-32674-6.
[19] KWON J, ZHANG JM, MOK B, et al. CK2-mediated phosphorylation up?regulates the stability of USP13 and promotes ovarian cancer cell prolif?eration[J]. Cancers, 2022, 15(1): 200. DOI: 10.3390/cancers15010200.
[20] PITRODA SP, PASHTAN IM, LOGAN HL, et al. DNA repair pathway gene expression score correlates with repair proficiency and tumor sensitivity to chemotherapy[J]. Sci Transl Med, 2014, 6(229): 229ra42. DOI: 10.1126/scitranslmed.3008291.
[21] XU YH, LI J, ZHU KK, et al. FIBP interacts with transcription factor STAT3 to induce EME1 expression and drive radioresistance in lung adenocarcinoma[J]. Int J Biol Sci, 2023, 19(12): 3816-3829. DOI: 10.7150/ijbs.83134.
[22] WEINANDY A, PIROTH MD, GOSWAMI A, et al. Cetuximab induces eme1-mediated DNA repair: A novel mechanism for cetuximab resis?tance[J]. Neoplasia, 2014, 16(3): 207-220, 220. e1-220. e4. DOI: 10.1016/j.neo.2014.03.004.
收稿日期:2023-10-11; 錄用日期:2023-12-11
本文編輯:王瑩

