生物學(xué)軟件在核酸序列比對(duì)與系統(tǒng)發(fā)育分析中的應(yīng)用
王小國
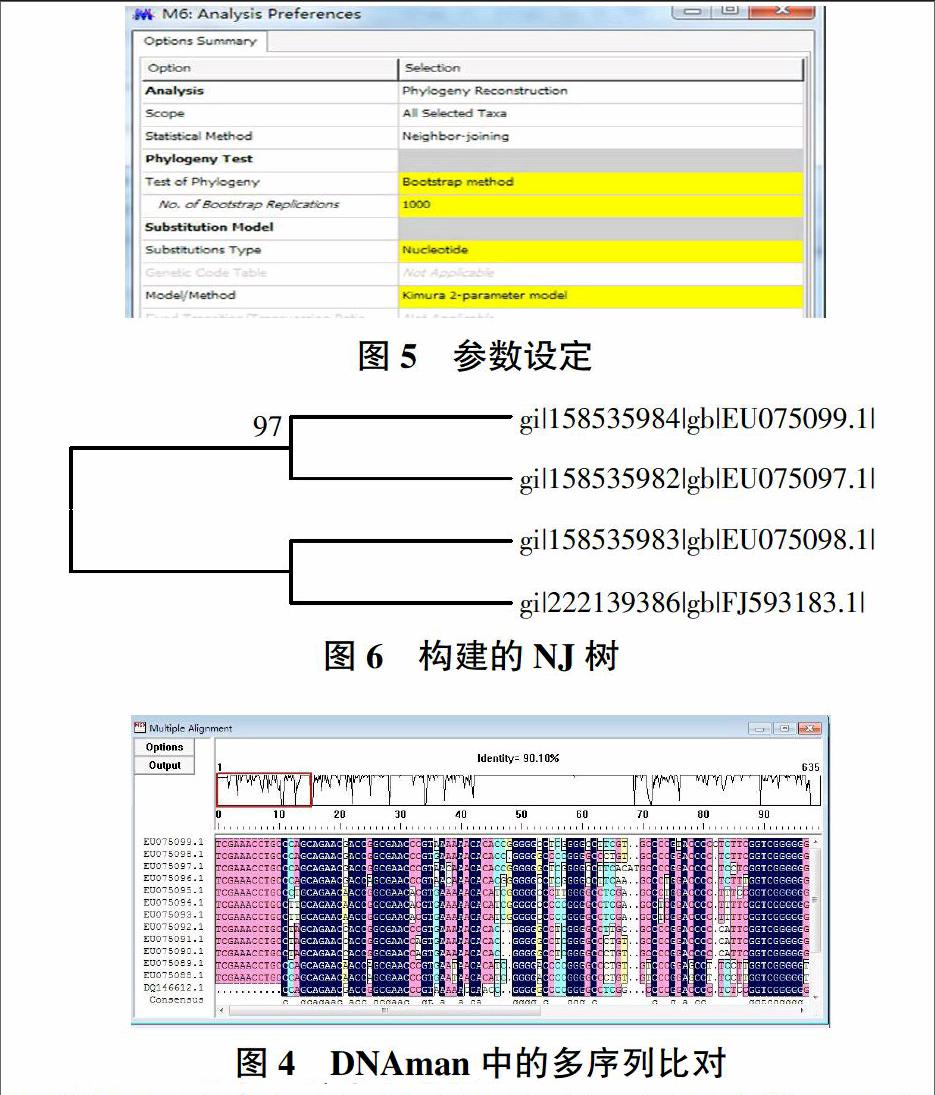
摘要 通過ClustalX2.1、Boxshade、DNAman和MEGA6等軟件的使用,介紹了生物學(xué)軟件在核酸序列比對(duì)、著色美化、序列分析和植物系統(tǒng)發(fā)育樹構(gòu)建中的應(yīng)用。并結(jié)合具體實(shí)例,采用鄰接法對(duì)核基因組中的內(nèi)轉(zhuǎn)錄間隔區(qū)序列進(jìn)行了構(gòu)樹,進(jìn)化枝的可信度較高,表明該方法適用于相似度較高、親緣關(guān)系較近序列的系統(tǒng)發(fā)育樹的構(gòu)建。
關(guān)鍵詞 序列比對(duì);鄰接法;系統(tǒng)發(fā)育樹;構(gòu)建
中圖分類號(hào) Q7;TP31 文獻(xiàn)標(biāo)識(shí)碼 A 文章編號(hào) 1007-5739(2015)12-0347-02
Application of Biology Software in the Nucleic Acid Sequence Alignment and Phylogenetic Analysis
WANG Xiao-guo
(Sanmenxia Polytechnic,Sanmenxia Henan 472000)
Abstract This paper introduced the application of biology software in the nucleic acid sequence alignment,beautification,sequence analysis and plant phylogenetic tree construction through the use of software such as ClustalX 2.1,Boxshade,DNAman and MEGA 6.Combined with the specific example,constructed neighbor-joined tree according to internal transcribed spacer of nuclear genome sequences,and the credibility of clade was higher,so the method was suitable for the close genetic relationship sequence in the construction of phylogenetic tree.
Key words sequence alignment;neighbor-joined method;phylogenetic tree;construct
核酸序列比對(duì)(Nucleic acid sequence alignment)又稱為核酸序列聯(lián)配,是通過2個(gè)或多個(gè)核酸序列的比對(duì),顯示出其中相似結(jié)構(gòu)區(qū)域的過程[1],是進(jìn)一步進(jìn)行相似性、序列間親緣關(guān)系和系統(tǒng)發(fā)育分析的基礎(chǔ)[2-3]。在系統(tǒng)發(fā)育分析中,最常用的可視化表示進(jìn)化關(guān)系的方法就是繪制系統(tǒng)發(fā)育進(jìn)化樹(Phylogenetic trees)。系統(tǒng)發(fā)育樹構(gòu)建有非加權(quán)分組平均法(Unweighted-pair-groop-method with arithmetic means,UPGMA)、鄰接法(Neighbor-joined,NJ)、最大簡約法(Maximum parsimony,MP)和最大似然法(Maximum likelihood,ML)等多種方法[4],由于鄰接法具有構(gòu)建的樹相對(duì)準(zhǔn)確、假設(shè)少且計(jì)算速度快的特點(diǎn),因而是目前最常用的方法之一。但NJ法所分析序列的遺傳距離不能太大,適用于相似度較高、親緣關(guān)系較近的短序列[5]。
核基因組序列中的內(nèi)轉(zhuǎn)錄間隔區(qū)(Internal transcribed spacer,ITS)主要編碼植物的核糖體RNA,屬于中度保守序列,因此可用于較低分類階元的系統(tǒng)發(fā)育研究[6-7]。本文以Ziziphus Mill.的部分ITS序列為對(duì)象,介紹了ClustalX2.1[8-9]、Boxshade、DNAman和MEGA6[10]等軟件在序列比對(duì)、著色美化、序列分析和植物系統(tǒng)發(fā)育樹構(gòu)建中的應(yīng)用。
1 序列獲取
從NCBI網(wǎng)站(http://www.ncbi.nlm.nih.gov/)下載Ziziphus Mill.的4條ITS核酸序列,以Fasta格式存儲(chǔ)于桌面文檔中,命名為four sequences.txt。
2 使用ClustalX進(jìn)行多序列比對(duì)
2.1 打……

